2. 深圳市宝安区妇幼保健院中心实验室, 广东 深圳 518133
健康成人携带微生物所编码的基因多于人体 自身基因组的100 倍[1, 2],被称为人类的“第二基 因组”[3],这些微生物大部分寄居在肠道,肠道菌 群对于消化吸收营养物质、抑制病菌及调节免疫 都发挥着重要作用[4]。新生儿、婴儿期是肠道菌群 定植、发育的重要阶段,此期肠道菌群的失衡可 能会影响远期甚至成年后的健康[1]。因而,了解新 生儿、婴儿期肠道菌群的发展模式及特点对研究 相关疾病、改善人口质量有重要意义。
对肠道微生态的研究以往多采用培养或传统 分子生物学(如PCR、变性梯度凝胶电泳)方法, 但它们对于研究复杂的肠道菌群有明显的局限性,而宏基因组学研究则可以避免上述方法的缺陷[5], 近年已逐渐应用于婴儿肠道微生态研究[2, 3],但多 集中于欧美发达国家,国内仅见零星报道[6]。由于 基因以及饮食等生活习惯的差异,不同地域人的 肠道菌群构成有显著差异[7, 8],因此有必要研究我 国婴儿肠道宏基因组特征。
婴儿肠道菌群的建立是一个连续、变化、受 很多因素影响的过程,以往研究多局限于一个时间 点,专注于某一种因素如喂养[6]、生产方式[9]、抗 生素[8] 等的影响,缺乏连续性认识。本研究将采 用以Illumia 为测序平台的16S rDNA 新一代高通量 测序技术对5 例健康婴儿1 岁以内的粪便细菌基因 组进行连续分析,探讨婴儿时期肠道宏基因组的动 态变化,为以后大规模研究中国婴儿肠道菌群分布 特征及婴儿肠道微生态相关疾病提供参照。 1 资料与方法 1.1 研究对象
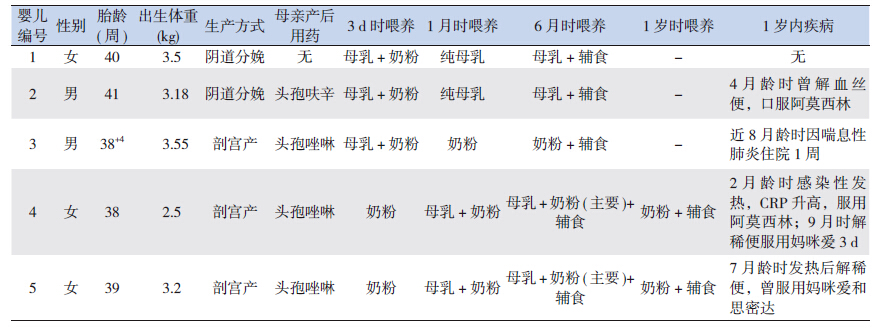
征得我院伦理委员会批准及婴儿父母知情同 意后,选择5 例出生于深圳市宝安区妇幼保健院、 胎龄37~40 周的健康足月新生儿为研究对象,记 录围产期信息及1 岁以内喂养方式、疾病和用药 信息(表 1)。
| 表 1 婴儿围产因素及喂养方式等信息 |
生后3 d、1 个月、6 个月和1 岁时用无菌标 本盒留取粪便样本共17 份,其中1~3 号婴儿样本 收集至生后6 个月各3 份,4、5 号婴儿收集至1 岁各4 份,提取细菌总DNA(QIAamp DNA Stool Mini Kit,德国QIAGEN 公司),保存于-70℃。 冷藏条件下送至深圳华大基因科技服务有限公司 进行测序及生物信息分析。 1.3 16S rDNA 宏基因组测序
检测合格的粪便DNA 样品用细菌通用引物进 行16S rRNA 基因V6 区PCR 扩增,引物序列为: 967F(v6) :CAACGCGAAGAACCTTACC,1046R (v6) :CGACAGCCATGCANCACCT; 回收主要 DNA 片段,用T4 DNA Polymerase、Klenow DNA Polymerase 和T4 PNK 将打断形成的粘性末端修复 成平末端,再通过3' 端加碱基“A”,使得DNA 片段能与3' 端带有“T”碱基的特殊接头连接以 构建文库;用合格的文库进行cluster 制备;应用 Hiseq 2000 测序仪(Illumina 公司,美国)对扩增 的16S rDNA V6 高变区序列进行测序,测序长度 为64 bp,测序类型:101 PE 测序;对原始的测序 数据进行处理(去除接头污染、含N 的碱基数和 低复杂度reads),获取clean data。 1.4 物种分类与丰度分析
将clean data reads 拼接成tags; 利用Mothur (version 1.27.0)软件包对tags 序列进行去冗余处 理,从中挑选出unique tag 序列;将unique tags 进 行物种注释并聚类成 Operational Taxonomic Units (OTU),tags 和OTU 的数量初步说明了该样品 是否具有丰富的物种;然后通过OTU 注释(基于 包含的tags 的物种注释结果)完成物种组成和分 类分析,可在界、门、纲、目、科、属、种水平, 将每个注释上的物种或OTU 在不同样品中的序列 数进行整理。本研究中,每个样品均有80% 以上的tags 在科水平上得到分类,因此最佳的分类水 平为科。 1.5 α 多样性分析
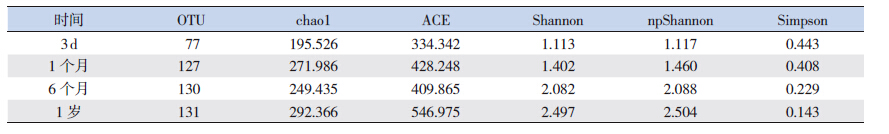
利用Mothur 软件计算样品的α 多样性值, 包括chao1 值、ACE 值、Shannon、npShannon 以及 Simpson 指数,α 多样性是对单个样品中物种多样 性的分析,前面4 个指标数值越大,最后一个指 标越小,说明样品中的物种越丰富。 2 结果 2.1 样品测序数据
17 份粪便细菌总DNA 样品共产生原始测序数 据为2 190.66 Mbp,每一样品产生的clean data 序 列平均为124 Mbp(116~131 Mbp),每一样品信 息的利用率都在94% 以上。 2.2 物种分类和丰度分析
OTU 数量最少为36,最多达308,随年龄的 增长而增加,表明婴儿粪便样品中微生物丰度随 时间而增加,见表 2。
| 表 2 不同时间点OTU 及α 多样性指数( 均值) |
20 个菌门被检出细菌基因组,但主要分布于 4 个菌门,分别是变形菌门 (Proteobacteria)、厚 壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes) 和放线菌门(Actinobacteria);不同个体之间,同 一个体的不同时段,其物种组成有差异,除1 号 婴儿集中于拟杆菌门和变形菌门外,其余婴儿样 品均集中于变形菌门和厚壁菌门。
图 1 为各样品丰度>1% 的菌群在为Family ( 科) 水平tags 的相对百分比。优势菌群包 括: 变形菌门下的Enterobacteriaceae( 肠杆菌 科)、Vibrionaceae( 弧菌科); 厚壁菌门下的 Lachnospiraceae(毛螺旋菌科)、Streptococcaceae (链球菌科)和Clostridiaceae(梭菌科);拟杆 菌门下的Bacteroidaceae(拟杆菌科);放线菌门 下的Bifidobacteriaceae(双歧杆菌科)。
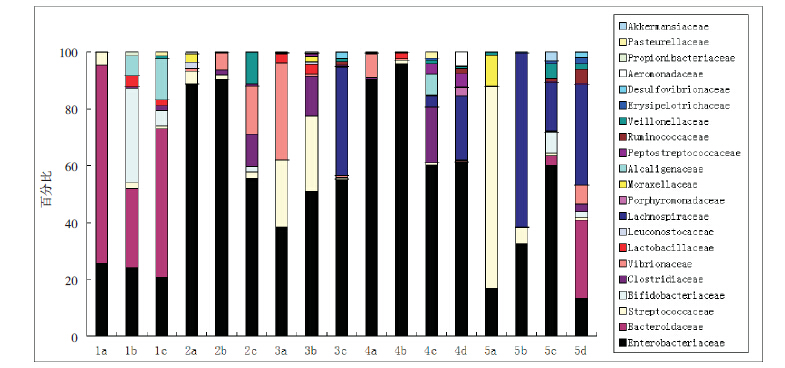 |
图 1 各样品tags 中>1% 的菌群在科水平的相对比例横轴数字1、2、3、4、5 为婴儿编号,a、b、c、d 分别代表 3 d、1 个月、6 个月和1 岁时间点。 |
从图中可以看出:虽然肠杆菌科出现于所有 婴儿粪便,甚至2 号婴儿生后1 个月时的粪便几 乎达90%,但不同婴儿之间菌群组成有明显差异, 例如1 号婴儿粪便中占主要比例的拟杆菌科在其 他婴儿的粪便中几乎没有出现或比例很小;出生3 d 时,虽然菌群种类存在差异,但总的趋势是结 构简单,主要由2~4 种物种组成,随着日龄延长, 物种组成逐渐丰富,菌群结构趋向复杂,生后1 个月、6 个月时样品中>1% 物种最多7 种,1 岁时 达10 种之多;同一婴儿不同时段的菌群构成虽随 着日龄而复杂化,但种类相似,如1 号婴儿粪便 一直以拟杆菌科占优势,而2、3、4 号婴儿粪便 中则肠杆菌科一直占优势;值得一提的是,益生 菌群双歧杆菌科在婴儿粪便中并不常见,仅纯母 乳喂养的1 号婴儿1 月时比例较高(达33%), 此后有所下降,所有婴儿生后3 d 的粪便中几乎不 含双歧杆菌科,虽然随着日龄和饮食结构复杂化 而出现,但一直没有超过10%;而毛螺旋菌科则 仅出现在奶粉或混合喂养的3 例婴儿粪便中,母 乳喂养的2 例婴儿粪便中未超过1%。 2.3 多样性分析
从表 2 可以看出,各α 多样性值中前4 个值 随着日龄而增加,而Simpson 指数随之减少,说明 粪便中菌群多样性随着日龄而增加,物种越来越 丰富。 3 讨论
本研究对5 例婴儿17 份粪便样本进行了16S rDNA 的V6 高变区新一代高通量测序及分析,表 明该技术能够全面分析婴儿肠道菌群的构成。以 Illumina 为测序平台的宏基因组测序技术以边合成 边测序为原理,可以一次性对几十万至几百万条 DNA 分子进行序列测定,在研究人类肠道宏基因 组方面发挥着重要作用[2, 3, 6]。16S rRNA 是细菌进 化以及分类研究最常用的靶分子,其基因序列(即 16S rDNA)的变化非常缓慢,可以用来标记生物 进化距离和亲缘关系,由于肠道微生物中绝大部 分为细菌,16S rDNA 宏基因测序技术能够满足肠 道宏基因组的研究。本研究表明,新生儿出生不 久肠道中即包含了丰富的细菌基因组。
国外有关肠道宏基因组的研究表明,欧美健 康成人及1~6 岁健康儿童肠道菌门以厚壁菌门和 拟杆菌门占优势[1, 10, 11]。国内研究不多,东北农业 大学Fan 等[6] 采用与本研究相同的技术对生后1~6 个月内24 份北方婴儿粪便细菌DNA 的测序表明, 与欧美不同,所有样本均以厚壁菌门和变形菌门为优势菌门;以肠杆菌科为优势菌科,但在母乳 喂养婴儿粪便中的含量明显低于人工喂养及混合 喂养婴儿。本研究中剖宫产出生、混合或人工喂 养、母亲产后应用抗生素的3~5 号婴儿1 岁以内 也以厚壁菌门和变形菌门为优势菌门,而阴道分 娩、纯母乳喂养、母亲未使用抗生素的1 号婴儿 肠道则以拟杆菌门和变形菌门为优势菌门,且一 直持续至生后6 个月,双歧杆菌科比例也是最高的; 同样为阴道分娩、纯母乳喂养的2 号婴儿肠道菌 群则与3~5 号婴儿类似,且持续至生后6 个月, 可能与母亲产后使用抗生素或其他围产期因素有 关。以上研究表明,婴儿肠道菌群的形成可能受 分娩方式、喂养、母亲围产期因素、甚至地域环 境等综合影响,中国婴儿甚至南北不同地域的婴 儿可能有其独特的菌群模式,但这些结论还需要 大样本、多地域人群的连续研究来证实。
Fan 等[6] 的研究仅有一个时间点,而本研究 显示,婴儿粪便中细菌的丰度及多样性随时间而 增加,提示随着饮食结构的复杂以及活动范围的 扩展,婴儿肠道菌群结构趋向复杂和多样。但值 得注意的是,出生不久即定植的菌群,1 岁以内可 能会一直存在,不同个体之间的菌群组成虽有明 显差异,而同一个体不同时段优势菌群构成相似, 表现出独特的个体特征,说明生命早期的因素如 生产方式、母亲用抗生素等可能会影响1 岁以内 甚至儿童、成人期的肠道菌群结构。
总之,本研究表明婴儿粪便中细菌物种丰度 及分类存在个体差异;从出生到1 岁,粪便中细 菌丰度及多样性随时间而增加,表明肠道菌群结 构趋向复杂和多样;在物种分类方面,不同个体 之间的菌群组成有明显差异,而同一个体不同时 段优势菌群构成相似,表现出独特的个体特征。 本研究的不足之处是样本数少,难以代表所有婴 儿肠道宏基因组特征,未来可行大样本、分组研究。
| [1] | Matamoros S, Gras-Leguen C, Le Vacon F, et al. Development of intestinal microbiota in infants and its impact on health[J]. Trends Microbiol, 2013, 21(4):167-173. |
| [2] | Qin J, Li R, Raes J, et al. A human gut microbial gene catalogue established by metagenomic sequencing[J]. Nature, 2010, 464(7285):59-65. |
| [3] | Song S, Jarvie T, Hattori M. Our second genome-human metagenome:how next-generation sequencer changes our life through microbiology[J]. Adv Microb Physiol, 2013, 62:119-144. |
| [4] | Di Mauro A, Neu J, Riezzo G, et al. Gastrointestinal function development and microbiota[J]. Ital J Pediatr, 2013, 39:15. |
| [5] | Berrington JE, Stewart CJ, Embleton ND, et al. Gut microbiota in preterm infants:assessment and relevance to health and disease[J]. Arch Dis Child Fetal Neonatal Ed, 2013, 98(4):F286-F290. |
| [6] | Fan W, Huo G, Li X, et al. Impact of diet in shaping gut microbiota revealed by a comparative study in infants during the six months of life[J]. J Microbiol Biotechnol, 2014, 24(2):133-143. |
| [7] | Yatsunenko T, Rey FE, Manary MJ, et al. Human gut microbiome viewed across age and geography[J]. Nature, 2012, 486(7402):222-227. |
| [8] | Fallani M, Young D, Scott J, et al. Intestinal microbiota of 6-week-old infants across Europe:geographic influence beyond delivery mode, breast-feeding, and antibiotics[J]. J Pediatr Gastroenterol Nutr, 2010, 51(1):77-84. |
| [9] | Biasucci G, Rubini M, Riboni S, et al. Mode of delivery affects the bacterial community in the newborn gut[J]. Early Hum Dev, 2010, 86(Suppl 1):13-15. |
| [10] | Arumugam M, Raes J, Pelletier E, et al. Enterotypes of the human gut microbiome [J]. Nature, 2011, 473(7346):174-180. |
| [11] | De Filippo C, Cavalieri D, Di Paola M, et al. Impact of diet in shaping gut microbiota revealed by a comparative study in children from Europe and rural Africa[J]. Proc Natl Acad Sci USA, 2010, 107(33):14691-14696. |
 2014, Vol. 16
2014, Vol. 16