近年研究表明黑皮素 4受体(melanocortin-4 receptor, MC4R)基因与体重调节密切相关。MC4R基因定位于染色体18q22,只有一个外显子,编码332个氨基酸残基组成的跨膜G蛋白耦联受体,具有七个跨膜区。MC4R存在于中枢神经系统的各个区域,包括大脑皮质、下丘脑、脑干和脊索;MC4R是人类体重调节的系统——瘦素-黑色素皮质激素系统的重要组成部分,可以通过影响食物摄入、能量消耗和代谢,与肥胖发生有密切关系[1]。
MC4R基因是导致人类单基因肥胖的基因之一。据文献报道约有2%~6%的重度肥胖儿童青少年携带有该基因的变异[2]。到目前为止,在人类MC4R基因编码区已发现约170个自然发生的突变[1, 3, 4]。大部分突变在欧美人群中发现,亚洲人群中MC4R基因的突变研究非常少[5]。一项在日本人中进行的小样本研究(50个肥胖病人)未发现MC4R基因编码区突变。中国大陆(杭州、南京和上海)人群和香港人群中进行的4项研究发现了10个突变。新加坡的华裔、马来裔和印度裔人群中进行的研究发现3个突变。Lee[5]对不同人群中MC4R基因编码区突变研究进行综述,发现尽管一些变异(如Val103Ile)在多项不同人群研究中被识别,但很多突变仅在原始研究人群中或者在几项研究中有过报道,即使相同种族的人群中也未多次发现这些突变。因此,不同区域的亚洲人群可能携带不同的MC4R基因编码区突变,有必要在中国人群中开展这方面的研究,以完善中国人群MC4R基因编码区突变谱。
本研究选择北京市160例7~18岁重度肥胖和100例体重正常的儿童青少年,对MC4R基因编码区进行突变筛查,分析筛出的变异与肥胖相关指标的关系,并预测变异可能的功能,以阐明MC4R基因编码区变异在肥胖发病机制中的作用。
1 资料与方法 1.1 研究对象根据2005年全国学生体质调研结果中的体重指数(BMI)百分位数[6],对北京市海淀区3所中学和2所小学的全体学生进行筛选。纳入标准:BMI在2005年全国学生同年龄同性别第97百分位数以上者为重度肥胖组,共160人,其中女生74例,男生86例,BMI 27.6±3.6 kg/m2,年龄11.2±2.7岁(7~18岁)。选择与肥胖组儿童性别、年龄分布相同的100名体重正常儿童作为对照组,BMI均在2005年全国学生同年龄同性别第15百分位数和50百分位数之间,其中女生49例,男生51例,BMI 16.5±1.6 kg/m2,年龄11.4±3.0(7~18)岁。对照组儿童均排除脂肪肝,血压、血糖、血脂四项(总胆固醇、三酰甘油、低密度脂蛋白胆固醇和高密度脂蛋白胆固醇)及肝功能均正常。
1.2 身体测量及血生化指标测量 依据2005年全国学生体质健康调研检测细则[7],对所有研究对象进行身高、体重、腰围、臀围的测量,计算BMI(体重/身高2)。采集空腹静脉血,血清分离后使用日立7600全自动血生化分析仪对空腹血糖、总胆固醇、三酰甘油、低密度脂蛋白胆固醇、高密度脂蛋白胆固醇水平进行测定。
1.3 DAN提取、PCR和酶切采用常规酚氯仿法从血凝块中提取基因组DNA。通过核对NCBI、Ensembl、HAPMAP等基因组数据库,发现该基因编码区为1个外显子。采用2对PCR引物扩增MC4R基因,引物设计参考文献[8]。第一对引物MC4R-1F:5'-ATCAATTCAGGGGGACACTG-3';MC4R-1R:5'-GACAGCACTACTATCTGAGT-3'(615 bp)。第二对引物MC4R-2F:5'-ATGCTCTCCAGTACCATAACA-3';MC4R-2R:5'-TGCAGAAGTACAATATTCAGG-3'(622 bp)。第一对引物的扩增产物使用内切酶Eco81I 和SspI进行酶切,产物大小分别为179、217、219 bp;第二对引物的扩增产物使用MspI进行酶切,产物大小为167、223、232 bp。
1.4 MC4R基因筛查和测序采用单链构象多态性分析(SSCP)方法对上述步骤中MC4R基因PCR-酶切产物进行突变筛查:(1)制备聚丙烯酰胺凝胶(30%丙烯酰胺-2%双丙烯酰胺);(2)按适当比例混合PCR产物及变性剂,混匀后96℃变性10 min,取出后立刻放入冰中冷却;(3)上样后于4℃环境中350 V电泳12~14 h;(4)电泳结束后通过银染的方法显色;(5)对发现可疑突变的样品进行测序,以确定突变的位置和类型。
1.5 突变功能预测使用生物信息学网络数据库PolyPhen-2(polymorphism phenotyping2)、SNAP(Screening for non-acceptable polymorphisms)和Mutation Taster对突变是否导致蛋白产物功能的改变进行预测分析。PolyPhen-2基于同源序列、同源蛋白质3D-结构的多重比对和系统发育学信息,估计突变型等位基因具有破坏蛋白质功能的可能性得分,得分范围介于0(良性)~1(破坏性)之间[9, 10]。SNAP参考基于序列得到的信息,如进化信息(同源序列家族内残基的保守性)、蛋白质结构信息(二级结构、可溶性)以及其他相关信息,分析氨基酸的置换是否导致蛋白功能的改变,并计算可靠性指数(reliability index,范围0~9)和预测准确率(范围0~1),后者是前者给定的基础上用来解释预测准确性的似然估计值,两者数字越大表明预测的准确性越高[11]。Mutation Taster整合来自不同生物数据库的信息(如蛋白质特征、序列和突变频率数据等),利用贝叶斯分类的方法计算序列中的变异是否具有致病性或者是有害变异的可能性。可能性参数越接近1,说明预测的准确性越高。从Ensembl 数据库下载序列数据,对新发现突变的物种保守性进行分析,即计算携带相同氨基酸物种的百分数。使用UniProt网站进行新突变功能域分析。
1.6 统计学分析采用SPSS 17.0统计软件进行统计学分析,计量资料使用均数±标准差( x±s)描述,组间比较采用两独立样本t检验;计数资料用频数和百分率(%)表示,组间比较采用卡方检验。P<0.05为差异有统计学意义。
2 结果如表 1所示,在160例肥胖儿童中检出错义突变3例,占1.9%;100例对照组中检出错义突变1例,占1.0%。肥胖组中的3个错义突变为:283G>A(Val95Ile)、496G>A(Val166Ile)、536T>C(Val179Ala)。对照组中的一个错义突变为653T>C(Met218Thr)。307G>A(Val103Ile)变异在肥胖、对照组中均出现,检出率分别为4.4%和6.0%,调整年龄和性别该变异与肥胖没有相关性(P=0.588)。肥胖组中536T>C(Val179Ala)为新发现的变异,其他研究中未见报道。
| 表 1 MC4R 基因变异在肥胖组和对照组的基因型频率分布 |
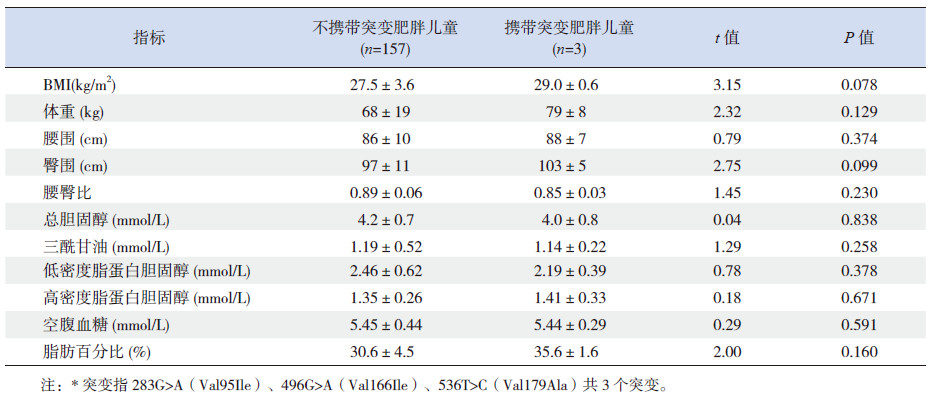
肥胖组中的3个错义突变283G>A(Val95Ile)、496G>A(Val166Ile)、536T>C(Val179Ala)均为女性杂合子突变携带者,BMI的水平分别是29.59、29.16和28.44 kg/m2;对照组发现的突变653T>C(Met218Thr)为男性杂合子突变携带者,其BMI为17.12 kg/m2 。比较携带这3个突变的肥胖儿童与未携带这3个突变的其他157例肥胖儿童的BMI水平,如表 2 所示,前者BMI平均值高于后者,但差异无统计学意义(P=0.078)。两组肥胖儿童体重、腰围、臀围、腰臀比、血脂、血糖和脂肪百分比的比较差异均无统计学意义(P>0.05)。
| 表 2 携带与不携带MC4R 基因突变的肥胖儿童相关指标水平的比较* |
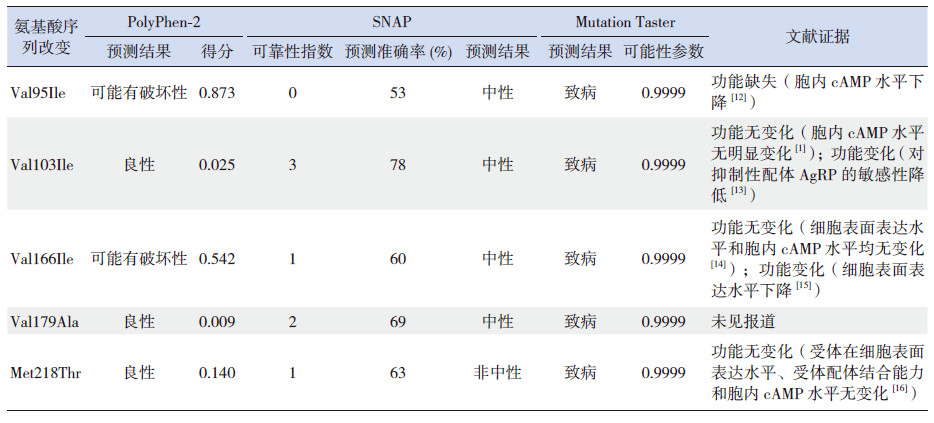
采用相关的网络数据库对突变的功能进行预测,结果见表 3。PolyPhen-2数据库预测结果显示Val95Ile 和Val166Ile突变可能破坏蛋白质的结构和功能,得分分别是0.873和0.542。SNAP数据库预测结果显示Met218Thr突变可能对蛋白质结构功能产生影响,可靠性指数是1,预测准确率为63%。Mutation Taster 数据库预测结果显示5个变异包括新发现突变Val179Ala均为致病性变异,可能性参数都是0.9999。从Ensembl数据库可获得的55个物种中,Val179Ala突变位置有49个物种均为缬氨酸(Val),物种间保守性百分数为89%。UniProt网站数据显示Val179Ala位于MC4R蛋白唯一的视紫红质超家族功能域的第4个跨膜螺旋区域。
| 表 3 MC4R 基因突变位点的功能预测 |
本研究对北京市160例7~18岁重度肥胖和100例体重正常的儿童青少年进行MC4R基因编码区的突变筛查,结果检出5个变异,肥胖组中发现3个错义突变:283G>A(Val95Ile)、496G>A(Val166Ile)、536T>C(Val179Ala);对照组中发现1个错义突变,即653T>C(Met218Thr);307G>A(Val103Ile)变异在肥胖组、对照组中均有发现,其发生频率分别为4.4%和6.0%,两组比较差异无统计学意义,提示该变异与肥胖无相关性,该结果与本课题组前期的一项meta分析结果不一致[17],可能与本研究样本量小有关。本研究在肥胖女童中检出的Val95Ile与Val166Ile突变,既往研究曾有报道,其中Val95Ile突变在一个挪威成年肥胖者中发现[18],Val166Ile突变在一个中国肥胖儿童中检出[19]。而Met218Thr突变的检出情况不同,本研究是在一个体重正常男性儿童中发现,而Rong等[16]在一个中国香港的肥胖女性中检出,提示该突变可能在病例和对照人群中均有可能携带,需要进一步大样本人群的验证。本研究首次发现Val179Ala突变,尚未见有研究报道,其携带者为一名女童,BMI为28.44 kg/m2。
研究发现MC4R基因突变率有种族差异。Tao[1]在一篇综述中对这种差异进行总结:在欧洲白人肥胖人群中,MC4R突变检出率介于1.72%~1.96%之间,北美印第安肥胖人群中检出率为2.25%~4.60%;东亚肥胖人群中的检出稍低,日本人研究(n=50)未检出MC4R突变,华裔肥胖人群中检出率介于0.3%~1.76%之间[16, 19, 20, 21, 22]。本研究在160例重度肥胖人群中检出MC4R突变3例,突变检出率为1.9%,略高于既往华裔人群研究检出率,可能与研究人群的肥胖程度不同有关。本研究人群为重度肥胖儿童,而前期的华裔人群研究为肥胖人群,重度肥胖儿童可能携带更多的MC4R突变。至于MC4R突变类型是否具有种族差异性,鉴于亚洲研究较少,目前尚不能进行定论,但是少数在中国人群中发现的突变(如Met218Thr)尚无其他种族的研究报道。
人类MC4R基因突变引起的肥胖表现多样,其肥胖多为青少年发病,且女性较重,与其他一些单基因突变引起的肥胖(如瘦素、瘦素受体缺陷等)相比,其内分泌代谢紊乱并不十分明显[1]。本研究比较携带突变位点的肥胖儿童与未携带突变位点的肥胖儿童的BMI、体重和腰臀围等指标,没有发现两组之间具有显著差异,血糖、血脂及脂肪含量的比较也未发现差异,与前期的研究结果一致[1]。
细胞膜上的MC4R与其配体α-MSH结合将启动G蛋白耦联的信号系统,活化腺苷酸环化酶,引起细胞内cAMP上升,介导细胞的一系列生理生化活动。MC4R基因编码区突变可能在蛋白产物合成、转运、受体配体结合作用效率等方向产生影响。本研究采用相关的网络数据库(PolyPhen2、SNAP和Mutation Taster)对本研究中的新突变Val179Ala是否对蛋白结构和功能产生影响进行预测。 SNAP借鉴了残基的可溶性信息(残基包埋在蛋白质高级结构内、暴露于表面或介于中间),使预测更加全面。而数据库中关于蛋白结构和功能的信息较多时,PolyPhen-2的预测稍好于SNAP[11]。Mutation Taster在参考蛋白质特征的基础上还参考变异在基因上的位置和频率等信息进行预测,提高预测的敏感性。鉴于不同的网络数据库进行预测的原理不同,本研究使用3个不同的数据库进行预测。Val179位于MC4R蛋白第4个跨膜区,物种间保守性较高,该位置的突变可能导致蛋白功能的改变。预测结果支持Val179Ala可能导致蛋白功能发生变化。
本研究比较了PolyPhen2、SNAP和Mutation Taster数据库对其他4个已知突变的预测与体外功能研究的结果。数据库预测结果显示4个突变均可能影响蛋白功能。文献报道Val95Ile突变可引起受体激动剂刺激的胞内cAMP水平降低[12];Val166Ile突变的作用在不同研究中结果不同,Hinney等[14]研究结果显示Val166Ile突变未导致胞内激动剂诱导的cAMP水平显著变化,而Rovite等[15]类似的体外实验则发现相反的结果,还发现Val166Ile可显著降低受体蛋白的细胞表面表达水平;Rong等[16]对Met218Thr的功能进行研究,未发现该突变对细胞表面受体的表达水平、受体配体结合能力和胞内cAMP水平产生影响,但是不排除该突变具有其他方面功能的可能性。对Val103Ile突变,体外研究显示与Val103相比,Ile103可能对AgRP抑制作用的敏感性较低,从而弱化食欲促进信号,使肥胖的风险降低,这一结果仍具有争议,至少4项研究未发现类似结果[1],但是人群数据证实Val103Ile与肥胖存在相关性[17]。Bromberg等[23]采用SNAP数据库对MC4R编码区突变功能进行预测,结果发现该数据库对受体与激动剂结合能力方向变化的预测优于受体与抑制剂结合能力变化的预测。数据库对于突变功能的预测与既往实验研究结果存在部分一致性,可为筛选功能重要的突变提供一定依据。需要注意的是,不同预测软件由于设计原理不同导致预测结果矛盾,预测结果与实验测试也存在一定矛盾,因此使用网络数据库对基因突变进行功能预测的结果仍需要进一步的实验研究来验证。
本研究在中国儿童青少年人群中对MC4R基因突变进行筛查,除了已知的4个变异,首次发现了Val179Ala突变。采用网络数据库预测MC4R基因突变的功能,发现包括新突变Val179Ala在内的5个变异可能对蛋白质功能产生影响。研究结果为完善中国人群MC4R基因编码区突变谱、阐明MC4R基因编码区变异在肥胖发病机制中的作用提供了依据。
| [1] | Tao YX. The melanocortin-4 receptor: physiology, pharmacology, and pathophysiology[J]. Endocr Rev, 2010, 31(4): 506-543. |
| [2] | Fan ZC, Tao YX. Functional characterization and pharmacological rescue of melanocortin-4 receptor mutations identified from obese patients[J]. J Cell Mol Med, 2009, 13(9B): 3268-3282. |
| [3] | Wang ZQ, Tao YX. Functional studies on twenty novel naturally occurring melanocortin-4 receptor mutations[J]. Biochim Biophys Acta, 2011, 1812(9): 1190-1199. |
| [4] | Tao YX. Mutations in melanocortin-4 receptor and human obesity[J]. Prog Mol Biol Transl Sci, 2009, 88: 173-204. |
| [5] | Lee YS. Melanocortin 3 receptor gene and melanocortin 4 receptor gene mutations: the Asian Perspective[J]. Diabetes Metab Res Rev, 2012, 28(Suppl 2): 26-31. |
| [6] | 中国学生体质与健康研究组. 2005年中国学生体质与健康调研报告[M]. 北京: 高等教育出版社, 2007: 193-197. |
| [7] | 中国学生体质与健康研究组. 2005年中国学生体质与健康调研检测细则[M]. 北京: 高等教育出版社, 2007. |
| [8] | Hinney A, Schmidt A, Nottebom K, et al. Several mutations in the melanocortin-4 receptor gene including a nonsense and a frameshift mutation associated with dominantly inherited obesity in humans[J]. J Clin Endocrinol Metab, 1999, 84(4): 1483-1486. |
| [9] | Adzhubei I A, Schmidt S, Peshkin L, et al. A method and server for predicting damaging missense mutations[J]. Nat Methods, 2010, 7(4): 248-249. |
| [10] | Adzhubei I, Jordan DM, Sunyaev SR. Predicting functional effect of human missense mutations using PolyPhen-2[J]. Curr Protoc Hum Genet, 2013, Chapter 7: t7-t20. |
| [11] | Bromberg Y, Yachdav G, Rost B. SNAP predicts effect of mutations on protein function[J]. Bioinformatics, 2008, 24(20): 2397-2398. |
| [12] | Hinney A, Hohmann S, Geller F, et al. Melanocortin-4 receptor gene: case-control study and transmission disequilibrium test confirm that functionally relevant mutations are compatible with a major gene effect for extreme obesity[J]. J Clin Endocrinol Metab, 2003, 88(9): 4258-4267. |
| [13] | Xiang Z, Litherland SA, Sorensen NB, et al. Pharmacological characterization of 40 human melanocortin-4 receptor polymorphisms with the endogenous proopiomelanocortinderived agonists and the agouti-related protein (AGRP) antagonist[J]. Biochemistry, 2006, 45(23): 7277-7288. |
| [14] | Hinney A, Bettecken T, Tarnow P, et al. Prevalence, spectrum, and functional characterization of melanocortin-4 receptor gene mutations in a representative population-based sample and obese adults from Germany[J]. J Clin Endocrinol Metab, 2006, 91(5): 1761-1769. |
| [15] | Rovite V, Petrovska R, Vaivade I, et al. The role of common and rare MC4R variants and FTO polymorphisms in extreme form of obesity[J]. Mol Biol Rep, 2014, 41(3): 1491-1500. |
| [16] | Rong R, Tao YX, Cheung BM, et al. Identification and functional characterization of three novel human melanocortin-4 receptor gene variants in an obese Chinese population[J]. Clin Endocrinol (Oxf), 2006, 65(2): 198-205. |
| [17] | Wang D, Ma J, Zhang S, et al. Association of the MC4R V103I polymorphism with obesity: a Chinese case-control study and meta-analysis in 55,195 individuals[J]. Obesity (Silver Spring), 2010, 18(3): 573-579. |
| [18] | Wangensteen T, Kolsgaard ML, Mattingsdal M, et al. Mutations in the melanocortin 4 receptor (MC4R) gene in obese patients in Norway[J]. Exp Clin Endocrinol Diabetes, 2009, 117(6): 266- 273. |
| [19] | Wang CL, Liang L, Wang HJ, et al. Several mutations in the melanocortin 4 receptor gene are associated with obesity in Chinese children and adolescents[J]. J Endocrinol Invest, 2006, 29(10): 894-898. |
| [20] | 蔡姝冰, 贾伟平, 方启晨, 等. F261S—肥胖患者中黑皮素 4 受体基因的新突变[J]. 中华内分泌代谢杂志, 2004, 20(4): 372-375. |
| [21] | Lee YS, Poh LK, Kek BL, et al. Novel melanocortin 4 receptor gene mutations in severely obese children[J]. Clin Endocrinol (Oxf), 2008, 68(4): 529-535. |
| [22] | 石星, 秦锐, 韩蓓. 南京地区单纯性肥胖患儿黑皮素 4 受体基因筛查[J]. 实用儿科临床杂志, 2010, 25(7): 488-490. |
| [23] | Bromberg Y, Overton J, Vaisse C, et al. In silico mutagenesis: a case study of the melanocortin 4 receptor[J]. FASEB J, 2009, 23(9): 3059-3069. |
 2015, Vol. 17
2015, Vol. 17