急性淋巴细胞白血病(acute lymphoblastic leukemia, ALL)是儿童中最常见的肿瘤。基因组学研究显示:在儿童ALL中,常发生涉及淋巴细胞分化及细胞周期调控的基因拷贝数异常[1, 2]。
目前,有多种方法可用于检测不同水平的基因拷贝数异常,如荧光原位杂交(FISH)、单核苷酸多态性(SNP)阵列及微阵列比较基因组杂交(Array-CGH)等,但都因实验程序繁琐、检测能力有限、价格昂贵而具有较大局限性[3, 4]。2002年荷兰Schouten等[5]研究建立起多重连接探针扩增(multiplex ligation-dependent probe amplification, MLPA)检测技术,它是一种结合分子杂交、连接和PCR的检测技术,于同一反应管内可同时检测出40多个不同核苷酸序列的拷贝数变化。该技术拥有不需进行细胞培养、高通量、高分辨率以及低成本等优点。在国外,该技术已广泛应用于临床[6]。
ETV6/RUNX1融合基因(又名TEL/AML1)是儿童ALL中最常见的融合基因。国内儿童ETV6/RUNX1+ALL的临床特征与国际上报道相似[7]。然而,中国儿童ETV6/RUNX1+ALL中基因拷贝数变异的研究较少。为了解我国儿童ETV6/RUNX1+ALL中基因拷贝数异常的发生情况,了解MLPA技术在该类患儿中应用的可行性,本研究应用MLPA技术对初诊ETV6/RUNX1+ALL患儿进行基因拷贝数异常的检测,并同时与Schwab等[8]的研究一样,与临床上广为应用的FISH检测结果相比较,现报告如下。
1 资料与方法 1.1 临床资料对2006年1月至2012年11月中国医学科学院血液病医院儿童血液病中心收治的初诊为ETV6/RUNX1+ALL住院患儿的临床资料进行回顾性分析,包括临床特征、染色体核型检测结果及FISH检测结果。最终有足量DNA标本的ETV6/RUNX1+ALL患儿共95例纳入本研究,经骨髓形态学、细胞组织化学染色、细胞免疫分型及细胞遗传学确诊。另选取38例健康志愿儿童作为正常对照。纳入本研究的患儿及健康儿童志愿者的监护人均知情同意。
1.2 基因组DNA提取取患儿和健康儿童志愿者骨髓液或外周血2 mL,采用DNA提取试剂盒(Axygen Biosciences公司产品,美国)提取基因组DNA,-20℃保存备用。
1.3 MLPA具体试验步骤MLPA试剂盒P335-A4(MRC-Holland公司,荷兰),包含涉及IKZF1基因(7p12.2)8个外显子的8个探针,PAX5基因(9p13.2)的7个探针,ETV6基因(12p13.2)的6个探针,RB1基因(13q14.2)的5个探针,BTG1及BTG1基因下游区域(12q21.33)的4个探针,EBF1基因(5q33.3)的4个探针,CDKN2A-CDKN2B基因(9p21.3)的3个探针,以及Xp22.33区域(其中包含 CRLF2、CSF2RA、IL3RA、P2RY8基因及SHOX区)。
取5 μL(20~30 ng/μL)的样品DNA,98℃处理5 min,25℃暂停;取出样品,降至室温,加入3 μL的杂交混合液[1.5 μL probemix(黑盖)+1.5 μL MLPA 缓冲液(黄盖)],95℃1 min,60℃温浴16~20 h;PCR仪降到20℃,取出PCR管;每管加入3 μL ligase buffer A和10 μL水,吹打混匀。取10 μL混合液到新的PCR管,分别标记为a、b两管;将所有管放回PCR仪,48℃暂停。往a管中加入10 μL连接酶混合液,混匀;往b管中加入10 μL连接酶-消化混合液,混匀;48℃温浴30 min;98℃加热5 min灭活酶;20℃暂停。从PCR仪中取出样品,降至室温;加入10 μL PCR混合液[PCR混合液:7.5 μL双蒸水+2 μL PCR Primer Mix(棕盖)+0.5 μL SALSA Polymerse(橙盖)];PCR扩增条件:95℃ 30 s,60℃ 30 s,72℃ 60 s,35个循环后,72℃ 20 min,15℃暂停。取0.5 μL样品+0.5 μL GeneScanner500LIZ? Size Standard+9 μL Hi-DiTM Formamide进行毛细管电泳,将所得电泳结果通过GeneMapper 4.0软件和Coffalyser 软件进行半定量分析,了解目的基因的片段缺失或扩增情况。
1.4 染色体核型及荧光原位杂交检测染色体核型分析:取患儿骨髓液2 mL,肝素抗凝,分离单个核细胞。经短期(24 h)培养后,按常规方法制备染色体标本,G显带分析20个分裂中期细胞,核型描述按《人类细胞遗传学国际命名体制(ISCN2009)》。
FISH探针与检测方法:探针选用双色双融合TEL/AML1 FISH探针(美国Vysis公司)。与处理过的骨髓标本进行杂交,荧光显微镜观察。实验方法及步骤详见参考文献[9]。
2 结果 2.1 患者一般资料95例ETV6/RUNX1+的ALL患儿中,男54例(57%),女41例(43%),中位年龄4(1~13)岁。中位初诊白细胞数8.68(1.61~239.80)×109/L,中位初诊血小板数47(10~441)×109/L。
2.2 MLPA检测结果95例患儿中,3例(3%)IKZF1基因出现拷贝数的减低,1例(1%)IKZF1基因出现拷贝数的扩增;13例(14%)PAX5基因出现拷贝数的减低,7例(7%)PAX5基因出现拷贝数的扩增;39例(41%)ETV6基因出现拷贝数的减低,13例(14%)ETV6基因出现拷贝数的扩增;7例(7%)RB1基因出现拷贝数的减低,3例(3%)RB1基因出现拷贝数的扩增;10例(11%)BTG1基因出现拷贝数的减低;9例(9%)EBF1基因出现拷贝数的减低,6例(6%)EBF1基因出现拷贝数的扩增;18例 (19%)CDKN2A/2B基因出现拷贝数的减低;5例(5%)出现Xp22.33区域拷贝数的减低,9例(9%)出现Xp22.33区域拷贝数的扩增;3例(3%)出现Jak2基因突变。在检测涉及的基因中,拷贝数变异率超10%的基因为EBF1、CDKN2A/2B、PAX5、ETV6、RB1、BTG1。
95例患儿基因拷贝数变异个数的中位数为 1(0~6)个。73例(77%)患儿具有至少一种基因拷贝数的改变,其中35例(37%)患儿有1种,20例(21%)患儿有2种,10例(11%)患儿有3种,8例(8%)患儿有4种或以上的基因拷贝数改变。
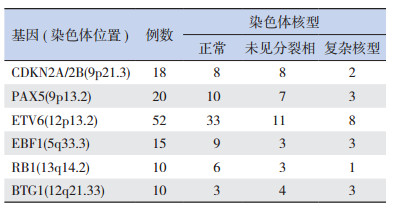
2.3 MLPA检测结果与染色体核型结果比较应用MLPA技术检测常见基因拷贝数异常的结果与染色体核型的结果进行比较,由表 1可见,常见基因拷贝数发生变异时,染色体核型常是正常的,或者未见分裂相。分析伴有染色体复杂核型的20例患者中,有2例累及12号染色体的异常,分别为46,XX,12p-,der(17)[2]/46,XX[2]和44-46,XY,der(12),8p+,9p+,der(11),12p-,-14,-17,+der(21),+12[cp6]/46,XY[2],涉及了ETV6基因拷贝数的改变;其余的伴有染色体复杂核型的患儿均未涉及相应的常见基因拷贝数变异。
| 表 1 常见基因拷贝数异常与染色体核型的相关性 |
未涉及CDKN2A/2B基因拷贝数改变的患儿中,有2例累及9号染色体异常,其中1例进行了MLPA检测,结果正常。未涉及ETV6基因拷贝数改变的患儿中,有2例累及12号染色体短臂异常,分别为46,XY,-11,12p+,+21[10]/46,XY[3]、47,XY,1q-,4q-,10q-,12p+,+21[cp9]/46,XY[6],均进行MLPA检测,结果正常。未涉及EBF1基因拷贝数改变的患儿中,未检测到累及5号染色体异常的患儿。未涉及RB1基因拷贝数改变的患儿中,未检测到累及13号染色体异常的患儿。
总之,发生拷贝数变异的基因涉及的染色体片段在染色体核型检测中常检测不到改变。
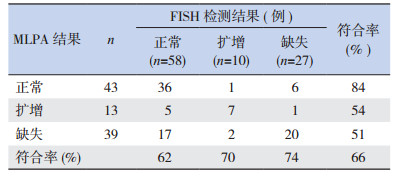
2.4 MLPA检测结果与FISH结果比较95例患儿应用TEL/AML1探针进行FISH检测,结果显示58例患儿TEL(ETV6)探针拷贝数正常(2个绿色荧光信号),27例患儿TEL(ETV6)探针拷贝数减低(<2个绿色荧光信号),10例患儿TEL(ETV6)探针拷贝数增加(>2个绿色荧光信号)。将FISH的检测结果与MLPA检测结果相比较后发现(表 2),两者的符合率为66%。两种检测方法结果差异最大的部分为:有22例(23%)经MLPA检测ETV6基因有扩增/缺失,而FISH检测ETV6基因结果正常。与FISH检测结果相比,MLPA检测结果的灵敏度为81.1%,特异度为62.1%。
| 表 2 MLPA 结果与 FISH 结果比较分析 |
基因组学研究显示:在儿童ALL中,常发生涉及淋巴细胞分化及细胞周期调控的基因拷贝数异常,甚至基因的拷贝数变异与预后及耐药均有关系[1, 2, 10, 11, 12]。SNP阵列及Array-CGH技术已经在儿童白血病的拷贝数改变的研究中起到了很大的作用[3, 4]。然而,由于费用昂贵、技术复杂,难以在临床中广泛应用。MLPA技术则由于技术简单、高通量、低成本等优点已广泛应用于临床。
Schwab等[6]应用MLPA(Kit 335-IKZF1)方法检测儿童ALL中基因拷贝数的变化,他们的研究发现:在ALL患儿中,59%的患儿有至少1种基因拷贝数异常,其中只有1种拷贝数改变的患儿占一半。在ETV6/RUNX1+ALL患儿中,ETV6缺失占54%,PAX5和CDKN2A/B缺失各占22%,BTG1缺失占15%,IKZF1缺失仅占3%[6]。本研究分析了ETV6/RUNX1+ALL患儿中涉及B淋巴细胞发育、细胞周期调控和血细胞生成的基因拷贝数的改变。尽管77%的患儿具有基因拷贝数的变异,但每个患儿发生变异的个数较少(中位数为1),并且主要集中在EBF1、CDKN2A/2B、PAX5、ETV6基因。基因发生改变的比例与Schwab等[6]得出的结论基本上是相似的。
染色体核型检测的方法常会遗漏小的异常改变[13]。MLPA技术则可以检测从单个碱基缺失到整条染色体数目变化的各个水平的基因组拷贝数变异[8]。Othman等[14]对61例染色体核型正常的ALL患儿应用MLPA及Array-CGH的方法进行分析后发现,可以检测到126个拷贝数异常,甚至在1例患儿中检测到了13个改变。我们将MLPA检测结果与常规染色体核型检测结果进行比较后发现,这两种方法的检测结果相关性较差。具有基因拷贝数改变的患者常无法检测到相应染色体改变,可能与染色体检测时无分裂相有关,亦可能与拷贝数改变的片段长度很短,染色体核型检测方法无法检测到有关。另外,在累及12号染色体短臂的2例患者,MLPA检测结果正常,其原因可能为发生改变的肿瘤细胞比例较低。与Schwab等[8]和Othman等[14]的报道是一致的。
我们将FISH的检测结果与MLPA检测结果相比较后发现,两者的相符率为66%。其原因可能为:当发生拷贝数变异的细胞比例低于20%时,MLPA技术无法检测到[8]。另外,两种检测方法结果差异最大的部分为MLPA检测有扩增/缺失,而FISH检测结果正常,占23%。其原因可能为当基因中发生改变的片段长度比较小时,FISH方法无法检测到,而MLPA技术可以检测到。与FISH检测结果相比,MLPA检测结果的灵敏度可以达到81.1%,因而,将这两种方法相结合应用将更有助于ETV6/RUNX1+ALL患儿中基因拷贝数的检测。
总之,在我国ETV6/RUNX1+ALL患儿中,同样存在基因拷贝数的变异,与国际上报道相似。MLPA方法具有良好的灵敏度,因而MLPA技术可应用于ETV6/RUNX1+ALL患儿中基因拷贝数变异的检测。
| [1] | Lilljebjörn H, Heidenblad M, Nilsson B, et al. Combined highresolution array-based comparative genomic hybridization and expression profiling of ETV6/RUNX1-positive acute lymphoblastic leukemias reveal a high incidence of cryptic Xq duplications and identify several putative target genes within the commonly gained region[J]. Leukemia, 2007, 21(10): 2137-2144. |
| [2] | Mullighan CG, Goorha S, Radtke I, et al. Genome-wide analysis of genetic alterations in acute lymphoblastic leukaemia[J]. Nature, 2007, 446(7137): 758-764. |
| [3] | Borst L, Wesolowska A, Joshi T, et al. Genome-wide analysis of cytogenetic aberrations in ETV6/RUNX1-positive childhood acute lymphoblastic leukaemia[J]. Br J Haematol, 2012, 157(4): 476-482. |
| [4] | Strefford JC, Worley H, Barber K, et al. Genome complexity in acute lymphoblastic leukemia is revealed by array-based comparative genomic hybridization[J]. Oncogene, 2007, 26(29): 4306-4318. |
| [5] | Schouten JP, McElgunn CJ, Waaijer R, et al. Relative quantification of 40 nucleic acid sequences by multiplex ligation-dependent probe amplification[J]. Nucleic Acids Res, 2002, 30(12): e57. |
| [6] | Schwab CJ, Chilton L, Morrison H, et al. Genes commonly deleted in childhood B-cell precursor acute lymphoblastic leukemia: association with cytogenetics and clinical features[J]. Haematologica, 2013, 98(7): 1081-1088. |
| [7] | 何军, 周剑彪, 薛永权, 等. 儿童急性淋巴细胞白血病TEL/ AML1 融合基因的检测及其临床意义[J]. 临床检验杂志, 2000, 18(6): 351-352. |
| [8] | Schwab CJ, Jones LR, Morrison H, et al. Evaluation of multiplex ligation-dependent probe amplification as a method for the detection of copy number abnormalities in B-cell precursor acute lymphoblastic leukemia[J]. Genes Chromosomes Cancer, 2010, 49(12): 1104-1113. |
| [9] | 周大文, 任兆瑞. 多重连接探针扩增技术及其应用[J]. 中华 医学遗传学杂志, 2009, 26(1): 45-49. |
| [10] | Nowak D, Liem NL, Mossner M, et al. Variegated clonality and rapid emergence of new molecular lesions in xenografts of acute lymphoblastic leukemia are associated with drug resistance[J]. Exp Hematol, 2015, 43(1): 32-43. |
| [11] | Loh ML, Mullighan CG. Advances in the genetics of highrisk childhood B-progenitor acute lymphoblastic leukemia and juvenile myelomonocytic leukemia: implications for therapy[J]. Clin Cancer Res, 2012, 18(10): 2754-2767. |
| [12] | Bokemeyer A, Eckert C, Meyr F, et al. Copy number genome alterations are associated with treatment response and outcome in relapsed childhood ETV6/RUNX1-positive acute lymphoblastic leukemia[J]. Haematologica, 2014, 99(4): 706-714. |
| [13] | Mrózek K, Harper DP, Aplan PD. Cytogenetics and molecular genetics of acute lymphoblastic leukemia[J]. Hematol Oncol Clin North Am, 2009, 23(5): 991-1010. |
| [14] | Othman MA, Melo JB, Carreira IM, et al. High rates of submicroscopic aberrations in karyotypically normal acute lymphoblastic leukemia[J]. Mol Cytogenet, 2015, 8: 45. |
 2016, Vol. 18
2016, Vol. 18