已有大量研究表明母亲宫内感染/炎症反应是导致胎儿早产的重要原因,并对早产儿神经系统发育造成严重影响[1]。近年来,对早产儿脑损伤分子生物学机制的研究取得了很大进展。多种蛋白编码基因及信号通路如 Cdk2基因、Olig2基因、Wnt/β-catenin、STAT-3、Notch信号通路等被证明参与早产脑损伤的发生及发展[2, 3],但未能完全阐明其关键点和机制。
近年来逐渐引起广泛关注的长链非编码RNA(long non-coding RNA,lncRNA)是长度大于200 nt的转录本[4, 5]。相比于miRNA及编码基因,lncRNA数量、种类更多,功能及作用机制相对更丰富,通过在染色体、转录、转录后、蛋白等多层次调控基因表达 [6, 7],参与生物体多种生理或病理过程,包括生物进化、细胞周期及机体信号传导,并调控感染、免疫反应的发生发展等[8, 9]。已有研究表明,lncRNA在脑发育及疾病中具有重要作用。如近期研究发现lncRNA表达谱在蛛网膜下腔出血所致早期脑损伤中发生明显变化[10];lncRNA FosDT能通过与相关蛋白互相作用而促进缺血性脑损伤的发展[11]。提示lncRNA的异常表达与脑损伤密切相关,但lncRNA在早产脑损伤中的研究尚未见报道。本研究中,我们应用脂多糖(lipopolysaccharide,LPS)作为炎症诱导因子,建立早产小鼠脑损伤模型,并采用lncRNA芯片技术筛选与炎症诱导早产脑损伤相关的差异表达lncRNA。本研究首次描述了早产脑损伤相关lncRNA表达谱,为探讨lncRNA与早产脑损伤的关系及其防治提供新思路。
1 材料与方法 1.1 主要材料与试剂雌性BALB/c小鼠与雄性C57小鼠由西安交通大学医学院动物中心提供;脂多糖购于美国Sigma公司;GeneChip Mouse Transcriptome(MT)Array1.0芯片购自美国Affymetrix公司;芯片数据分析由上海其明公司提供;TRIzol购自美国Invitrogen公司;RT-PCR试剂盒购于日本 Takara公司;StepOne Plus荧光定量PCR仪购自美国ABI公司;β-actin及2条lncRNA引物由上海生工公司合成。
1.2 炎症诱导早产动物模型制备成年雌性BALB/c小鼠28只与雄性C57小鼠14只于SPF级环境饲养。按雌雄2 : 1合笼,以检查到母鼠出现阴栓为妊娠0 d。将孕鼠随机分为对照组和LPS组单独饲养,每组14只。妊娠17 d时,LPS组于不同时间点连续两次腹腔注射LPS(血清型 055:B5,美国Sigma公司)50 μg/kg,对照组注射相同剂量磷酸盐缓冲液。对照组孕鼠于妊娠21 d时分娩的小鼠纳入足月组,LPS组孕鼠于妊娠18 d时分娩的小鼠纳入早产组[12]。
1.3 脑组织病理学检查两组各取6只仔鼠脑组织,去除小脑及脑干后置于4%多聚甲醛中固定,梯度乙醇脱水。常规石蜡包埋,冠状面切片厚度10 μm、行苏木精-伊红(HE)染色,40倍光镜下观察脑组织病理学变化。
1.4 芯片选择与探针设计利用 Gene Chip Mouse Transcriptome(MT)Array 1.0芯片,该芯片覆盖NCBI RefSeq、Ensemble、NONCODE、lncRNA db等多个权威数据库,可检测约55 000条lncRNA。芯片的探针为长度25-mer的寡核苷酸,用于芯片杂交中质量评估的外标基因有BioB、BioC、BioD和Cre X。
1.5 样本总RNA的提取与检测两组各取3只仔鼠脑组织,用TRIzol提取脑组织样本总RNA。紫外分光光度计检测总RNA浓度及A260/A280,结合琼脂糖凝胶电泳图,分析样本总RNA质量。
1.6 芯片实验两组各取3只仔鼠脑组织,按照Affymetrix公司的GeneChip WT PLUS Reagent Kit试剂盒说明书合成ss-cDNA,并对ss-cDNA进行片段化、标记,再与Affymetrix的基因芯片杂交。杂交后应用 Fluidics Station 450对芯片进行洗染。应用Affymetrix的GeneChip Scanner 3000 7G扫描仪对芯片的荧光强度进行扫描,再将CEL文件输入Expression Console软件进行数据标准化处理。以上芯片实验由上海其明公司完成。
1.7 Real-time PCR验证选取2条lncRNA进行real-time PCR验证,以β-actin为内参基因,引物序列见表 1。两组各取3只仔鼠脑组织,用TRIzol提取脑组织样本总RNA,将总RNA逆转录为cDNA。以cDNA为模板,采用Step One Plus荧光定量PCR仪,SYBR Green荧光染料法进行相对定量。采用20 μL反应体系,含SYBR Premix 10 μL,上、下游引物各0.5 μL,cDNA模板2 μL,DEPC水7 μL。反应条件:95℃变性30 s;95℃变性5 s,60℃退火30 s,35个循环。结果以Ct值表示,采用2-△△Ct法比较lncRNA在早产组和足月组小鼠脑组织中的表达差异。
|
|
表 1 lncRNA 及β-actin 引物序列 |
采用SPSS 17.0统计软件对数据进行统计学分析,计量资料以均数±标准差(x ±s)表示,采用独立样本t检验比较lncRNA在两组样本中的表达差异,P<0.05为差异有统计学意义。
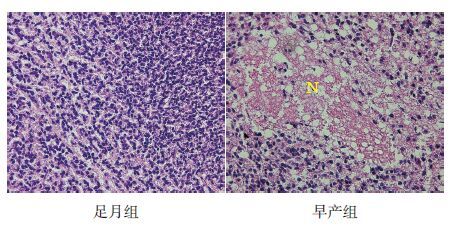
2 结果 2.1 脑组织病理学改变肉眼可见早产组小鼠脑组织充血、肿胀明显。光镜下观察:早产组小鼠脑组织细胞排列疏松,并出现大面积坏死灶,灶内神经元广泛溶解、消失,出现大量泡沫细胞;而足月组小鼠脑组织细胞排列较整齐紧密,细胞形态正常,未见明显坏死灶及泡沫细胞。见图 1。
|
图 1 新生鼠脑组织苏木精-伊红染色 (×40) 足月组小鼠脑组织细胞排列紧密,未见坏死灶;早产组小鼠脑组织可见坏死灶。N 为坏死灶。 |
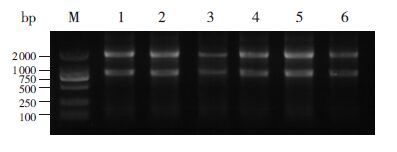
所有样本总RNA的A260/A280值均在1.8~2.2之间,琼脂糖凝胶电泳可见28S和18S两条核糖体RNA条带(图 2),质量检测结果全部通过(表 2),可用于后续芯片检测。
|
图 2 脑组织样本总RNA 电泳图 M 为Marker;1~3分别为早产组小鼠脑组织3 个样本;4~6 分别为足月组小鼠脑组织3 个样本。 |
|
|
表 2 小鼠脑组织RNA 质检结果 |
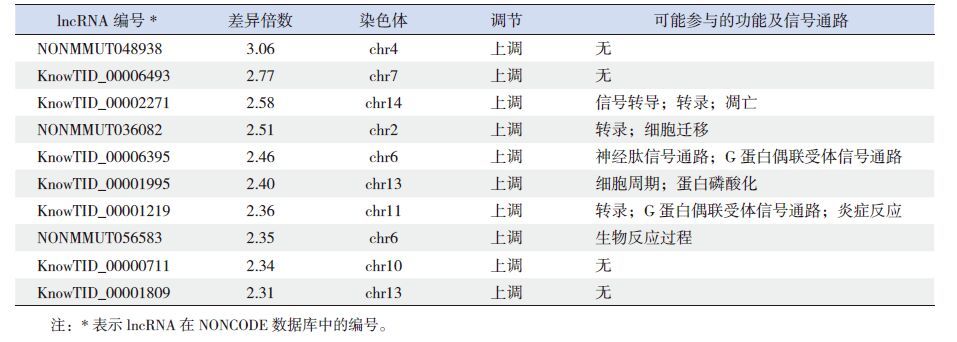
将质检合格的6份样本进行芯片检测,结果显示:在早产组与足月组中,显著差异表达的lncRNA共有1 978条(取P<0.05),包括上调lncRNA 786条,下调lncRNA 1 192条。其中差异表达1.5倍及以上的lncRNA共有29条。通过NONCODE数据库(www.bioinfo.org/NONCODEv4)对差异表达倍数最高的前10条lncRNA进一步分析,发现这些lncRNA参与众多生物学过程,如转录、信号转导、凋亡、细胞周期、炎症反应等,同时,还与G蛋白偶联受体(GPCRs)信号通路、神经肽信号通路有关(表 3)。
|
|
表 3 差异表达倍数最高的前10条lncRNA |
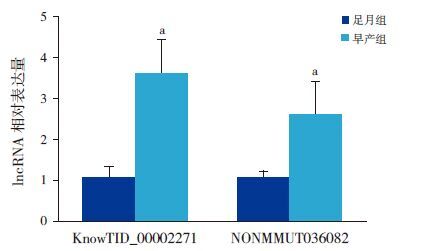
应用Real-time PCR对2条lncRNA在早产组和足月组脑组织中的表达进行验证,发现与芯片结果一致(图 3)。
|
图 3 Real-time PCR 检测脑组织差异表达lncRNA (n=3) lncRNA KnowTID_00002271、NONMMUT036082 在早产组小鼠脑组织中表达明显上调,与芯片结果一致。a示与足月组比较,P<0.01。 |
已有研究表明,lncRNA在多种神经系统疾病中发挥重要作用。如lncRNA BDNF-AS参与亨廷顿病、精神分裂症、抑郁症等的发生发展[8];lncRNA BACE1-AS在阿尔兹海默病患者脑组织中上调表达,与其发病机制密切相关[13]。然而lncRNA在与早产导致的脑损伤、脑发育异常的相关研究尚未见报道。高通量lncRNA芯片技术为寻找早产脑损伤诊治靶点提供了捷径。本研究成功建立了宫内炎症诱导早产小鼠脑损伤模型,通过lncRNA芯片技术对早产及足月产小鼠脑组织中lncRNA的表达谱进行分析。研究发现,在两组小鼠脑组织中,显著差异表达的lncRNA共有1978条,上调表达的lncRNA有786条,下调表达的lncRNA有1192条。这提示,脑损伤早产鼠脑组织中的lncRNA表达谱发生了明显变化,这些表达变化的lncRNA可能参与了早产脑损伤的发病过程。
脑发育过程中涉及一系列复杂的分子事件,需要对基因的时空表达进行精确的调控,从而形成复杂的神经网络系统[14]。lncRNA在发育及成熟的大脑中广泛表达[15],能够在多层面调控编码基因表达。如在表观遗传水平调节DNA甲基化、调节组蛋白修饰、调节基因组印记、诱发染色体重组等;lncRNA还可以通过与编码基因相互作用形成小干扰RNA调节基因表达水平等方式参与生物体多种生理或病理过程。为阐明lncRNA在早产脑损伤中的功能,本研究进一步对差异表达倍数较高的lncRNA进行分析后发现,这些变化的lncRNA参与众多生物学过程,如转录、信号转导、凋亡、细胞周期、炎症反应等。因此我们推测,lncRNA在早产脑损伤的发生中可能具有重要的作用,但其具体作用方式还需进一步研究。同时,还发现差异表达的lncRNA与GPCRs等信号通路相关,提示GPCRs可能参与了早产脑损伤的调控。GPCRs是人类最大的一类跨膜受体家族,具有丰富的种类和功能,通过与相应配体结合,完成细胞内外的信号转导。GPCRs介导的信号通路对体内神经系统、心血管系统、免疫系统、肿瘤的发生发展等发挥广泛调控作用,并与多条信号通路如Wnt、MAPK等信号通路具有交互作用。GPCRs被证明在神经系统的发育及疾病中有重要作用。如近年来研究较多的GPR56能够调控少突胶质细胞发育[16];其与配体collagen Ⅲ结合可激活RhoA信号通路,在脑皮质发育和分层中发挥重要作用[17, 18]。其他GPCRs如GPR3、GPR17等也被报道与神经系统发育密切相关。但至今尚无GPCRs与lncRNA在早产脑发育中相互作用关系的报道,二者是否以配体受体结合的方式发挥作用还不得而知,因此可将其作为下一步研究的一个新思路。
越来越多的研究表明lncRNA在神经系统发育及疾病中具有重要作用。而相对于蛋白编码基因,lncRNA在神经系统中的研究在国内外还处于起步阶段。我们虽然已筛选出在早产小鼠脑组织中差异表达的lncRNA,并对其表达谱进行了初步分析,但对于这些lncRNA的功能及与编码基因的作用关系还不明确。后续研究中,我们将建立lncRNA与编码基因等的共表达网络,并通过lncRNA相关生物技术对lncRNA的功能及作用机制进行深入探讨。
| [1] | Yuan TM, Sun Y, Zhan CY, et al. Intrauterine infection/ inflammation and perinatal brain damage: role of glial cells and Toll-like receptor signaling[J]. J Neuroimmunol, 2010, 229(1-2): 16-25. |
| [2] | Salmaso N, Jablonska B, Scafidi J, et al. Neurobiology of premature brain injury[J]. Nat Neurosci, 2014, 17(3): 341-346. |
| [3] | Fancy SP, Harrington EP, Baranzini SE, et al. Parallel states of pathological Wnt signaling in neonatal brain injury and colon cancer[J]. Nat Neurosci, 2014, 17(4): 506-512. |
| [4] | Nagano T, Fraser P. No-nonsense functions for long noncoding RNAs[J]. Cell, 2011, 145(2): 178-181. |
| [5] | Ponting CP, Oliver PL, Reik W. Evolution and functions of long noncoding RNAs[J]. Cell, 2009, 136(4): 629-641. |
| [6] | Mercer TR, Dinger ME, Mattick JS. Long non-coding RNAs: insights into functions[J]. Nat Rev Genet, 2009, 10(3): 155-159. |
| [7] | Wilusz JE, Sunwoo H, Spector DL. Long noncoding RNAs: functional surprises from the RNA world[J]. Genes Dev, 2009, 23(13): 1494-1504. |
| [8] | Ng SY, Lin L, Soh BS, et al. Long noncoding RNAs in development and disease of the central nervous system[J]. Trends Genet, 2013, 29(8): 461-468. |
| [9] | Qureshi IA, Mehler MF. Emerging roles of non-coding RNAs in brain evolution, development, plasticity and disease[J]. Nat Rev Neurosci, 2012, 13(8): 528-541. |
| [10] | Zheng B, Liu H, Wang R, et al. Expression signatures of long non-coding RNAs in early brain injury following experimental subarachnoid hemorrhage[J]. Mol Med Rep, 2015, 12(1): 967-973. |
| [11] | Mehta SL, Kim T, Vemuganti R. Long noncoding RNA FosDT promotes ischemic brain injury by interacting with RESTassociated chromatin-modifying proteins[J]. J Neurosci, 2015, 35(50): 16443-16449. |
| [12] | Wang F, Xiao M, Lin XJ, et al. Expression of heme oxygenase 1 and leukemia inhibitory factor in maternal plasma and placental tissue in a lipopolysaccharide induced late-pregnancy preterm birth mouse model[J]. J Reprod Med, 2016, 61(1-2): 39-46. |
| [13] | Faghihi MA, Modarresi F, Khalil AM, et al. Expression of a noncoding RNA is elevated in Alzheimer's disease and drives rapid feed-forward regulation of beta-secretase[J]. Nat Med, 2008, 14(7): 723-730. |
| [14] | 杨峰, 易凡, 曹慧青, 等. 长链非编码RNA 研究进展[J]. 遗传, 2014, 36(5): 456-468. |
| [15] | Hangauer MJ, Vaughn IW, McManus MT. Pervasive transcription of the human genome produces thousands of previously unidentified long intergenic noncoding RNAs[J]. PLoS Genet, 2013, 9(6): e1003569. |
| [16] | Giera S, Deng Y, Luo R, et al. The adhesion G proteincoupled receptor GPR56 is a cell-autonomous regulator of oligodendrocyte development[J]. Nat Commun, 2015, 6: 6121. |
| [17] | Bae BI, Tietjen I, Atabay KD, et al. Evolutionarily dynamic alternative splicing of GPR56 regulates regional cerebral cortical patterning[J]. Science, 2014, 343(6172): 764-768. |
| [18] | Singer K, Luo R, Jeong SJ, et al. GPR56 and the developing cerebral cortex: cells, matrix, and neuronal migration[J]. Mol Neurobiol, 2013, 47(1): 186-196. |
 2016, Vol. 18
2016, Vol. 18