6-巯基嘌呤(6-mercaptopurine, 6-MP)、6-硫鸟嘌呤(6-thioguanine, 6-TG)及其前体硫唑嘌呤均为嘌呤类衍生物,是贯穿儿童急性淋巴细胞白血病(acute lymphoblastic leukemia, ALL)治疗始终的重要药物,也在炎症性肠病、绒毛膜上皮癌等疾病的治疗中发挥作用。其结构与次黄嘌呤类似,在人体内的代谢途径(图 1)主要有两个方向[1-2]:(1)经黄嘌呤氧化酶(xanthine oxidase, XO)催化形成6-硫尿酸排出体外或经巯嘌呤甲基转移酶(thiopurine methyltransferase, TPMT)催化形成无活性的甲基化产物,如6-甲基巯嘌呤(6-methylmercaptopurine, 6-MMP)、6-甲基化巯基次黄嘌呤核苷磷酸盐(6-methylmercaptopurine nucleotides, 6-MMPN);(2)由次黄嘌呤鸟嘌呤磷酸核糖转移酶(hypoxanthine-guanine phosphoribosyltransferase, HGPRT)催化形成6-巯基次黄嘌呤核苷单磷酸(6-thio-inosine monophosphate, 6-TIMP),再经次黄嘌呤核苷酸脱氢酶(inosine monophosphate dehydrogenase, IMPDH)、鸟嘌呤核苷单磷酸合成酶(guanosine monophosphate synthetase, GMPS)作用,最终形成包括6-硫鸟嘌呤核苷酸三磷酸(6-thioguanosine triphosphate,6-TGTP)的6-硫鸟嘌呤核苷酸(6-thioguanine nucleotides, 6-TGNs)。6-TGTP可作为脱氧核糖核酸聚合酶底物替代鸟嘌呤(G)掺入DNA形成DNA-TG,在后续DNA复制中与胸腺嘧啶(T)发生错配,激活错配修复机制,导致DNA损伤、细胞死亡等[3-6]。
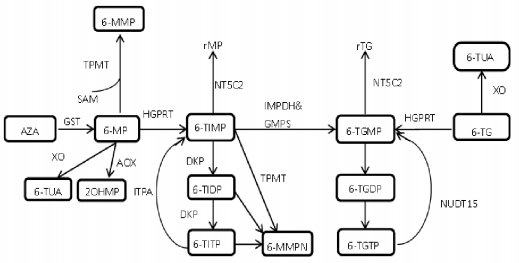
|
图 1 巯嘌呤代谢通路 [AOX]醛氧化酶;[AZA]硫唑嘌呤;[DKP]二磷酸核苷激酶;[GMPS]鸟苷单磷酸合成酶;[GST]谷胱甘肽转硫酶;[HGPRT]次黄嘌呤-鸟嘌呤磷酸核糖转移酶;[IMPDH]次黄嘌呤核苷酸脱氢酶;[6-MP] 6-巯基嘌呤;[6-MMP] 6-甲基巯嘌呤;[6-MMPN] 6-甲基化巯基次黄嘌呤核苷磷酸盐;[2-OHMP] 2-羟基巯嘌呤;[rMP]巯基嘌呤核苷;[rTG]硫鸟嘌呤核苷;[SAM] S-腺苷-L-甲硫氨酸;[TPMT]巯嘌呤甲基转移酶;[6-TG] 6-硫鸟嘌呤;[6-TIMP/TIDP/TITP] 6-巯基次黄嘌呤核苷单/二/三磷酸;[6-TGMP/TGDP/TGTP]6-硫鸟嘌呤核苷单/二/三磷酸;[6-TUA]6-硫尿酸;[XO]黄嘌呤氧化酶;[NT5C2] cytosolic 5'-nucleotidase Ⅱ;[NUDT15] nucleoside diphophate-linked moiety X-type motif 15。 |
MP代谢过程复杂,涉及多种酶;编码这些酶的基因变异所致酶活性变化影响其药理毒理作用。
1 TPMT基因多态性与巯嘌呤临床应用TPMT是目前研究最多的一种巯嘌呤代谢相关酶,在S-腺苷-L-甲硫氨酸(S-adenosy-L-methionine, SAM)提供甲基的情况下,可使代谢过程中多种产物甲基化,如6-MP形成6-MMP,6-TITP/6-TIDP/6-TIMP形成6-MMPN。6-MMPN/6-TGNs的浓度平衡与巯嘌呤疗效密切相关,TPMT高活性者6-MMPN浓度升高、肝毒性发生增多;TPMT活性下降,在同等巯嘌呤剂量下6-TGNs浓度升高,骨髓抑制发生风险提高[2, 7]。
1.1 TPMT影响巯嘌呤代谢的机制TPMT位于6号染色体,有多个多态性位点,最为常见的SNP为TPMT*2(c.238G > C)、*3A(c.460G > A、c.719A > G)、*3C(c.719A > G),这些多态性位点与90%的酶活性降低相关,亚裔人群以TPMT*3C多见[1, 8-9]。人群中约5%~10%为TPMT杂合子,有不同程度的酶活性下降,而纯和突变致功能缺失者仅占1/300 [9-10]。
体外实验证实上述几种突变不影响蛋白的转录及翻译,TPMT野生型的蛋白正常折叠、蛋白酶体降解、聚合体形成是维持正常酶功能的三个必要途径[11]。通过酵母细胞表达及不同基因型表达蛋白的稳定性研究发现,TPMT*3A、*2等变异型蛋白稳定性下降、半衰期明显缩短、稳态水平降低;TPMT*3A等变异型的蛋白由于空间结构变化,更容易发生错误折叠或形成蛋白聚合体,而被蛋白酶水解[11-12]。
1.2 TPMT与巯嘌呤的临床应用 1.2.1 药物耐受性在ALL维持治疗中,需要6-MP发挥一定的骨髓抑制作用,使白细胞维持在稍低于正常水平的一定范围内,通常为2.0~3.0×109/L。有文献报道,ALL维持治疗期间如果能使患者中性粒细胞绝对计数维持在2×109/L以下,复发率将降低10%以上[1]。另一方面,巯嘌呤可导致中性粒细胞造血功能损害并继发感染,尤其是当中性粒细胞计数小于0.5×109/L时发生严重感染的机会明显增加[1]。临床医生正探索如何更好地平衡这种利弊。
对于TPMT酶活性下降或缺失者,标准剂量的6-MP就可能蓄积大量6-TGNs,进而产生毒副作用,因此部分国家建议使用嘌呤类药物前进行TPMT基因型检测,并发布了根据TPMT基因型调整药物用量的指南。美国临床药物基因组学实施联盟建议,纯合突变致酶功能缺失(携带2个如下等位基因:TMPT*2、TPMT*3、TPMT*4)的个体嘌呤类药物初始剂量应当减少90%或降低频次;杂合子(有1个正常的等位基因TPMT*1,并同时携带上述任意一个无功能等位基因)个体则建议以正常剂量的30%~70%起始,并根据骨髓抑制情况进行调整;荷兰药物基因组学指南则建议杂合子、纯合子个体换用其他药物或者分别减少50%、90%的剂量[13]。
1.2.2 与ALL预后的关系6-MP是ALL维持治疗的重要药物,红细胞内6-TGNs的浓度可能影响ALL预后[14]。因此TPMT与ALL的预后密切相关。北美及欧洲儿童肿瘤协作组报道,TPMT酶活性低者红细胞内6-TGNs水平相对较高,复发风险较低,但第二肿瘤发生比例增高[15-16]。为了进一步研究TPMT与预后的关系,北欧儿童血液肿瘤协会比较了采用NOPHO-ALL92及ALL2000两组方案治疗的ALL患儿预后情况,后者在维持治疗中调整了TPMT杂合个体的巯嘌呤初始剂量(由75 mg/m2降至50 mg/m2),结果提示降低剂量患儿的第二肿瘤发生风险降低,但复发风险增高[16-17]。
2 NUDT15基因多态性亚裔人群6-MP耐受剂量明显低于欧非拉地区,不良反应的发生率亦不低,但其TPMT突变频率远不如欧非拉等地区的5%~10%[18-19],这意味着亚裔人群可能存在其他与6-MP代谢关系密切的基因。2014年首先在炎症性肠病患者中发现NUDT15(nucleoside diphophate-linked moiety X-type motif 15)可能与巯嘌呤相关的白细胞减少密切关联,随后在ALL患者尤其是亚裔人群发现NUDT15可能与巯嘌呤耐受、骨髓抑制相关[18-19]。
2.1 NUDT15基因多态性及其影响巯嘌呤代谢的机制NUDT15隶属于NudiX(nucleoside diphosphate linked to another moiety X)水解酶家族,也被称为MTH2(MutT homologue 2)。NudiX水解酶家族含有保守的由23个氨基酸残基组成的MutT相关序列,广泛存在于病毒、细菌、真核生物,催化与其他基团(-X)结合的核苷酸磷酸盐水解,并释放焦磷酸;他们含有特征性的序列“NudiX box” -GX5EX7REUXEEXGU,位于α螺旋-β折叠-α螺旋结构域上,可协调与镁离子的结合,形成与底物特异结合的区域[3, 6, 20]。电离辐射、氧化还原反应等产生的活性氧可损伤细胞核苷池中的dNTP;损伤的异常核苷酸,如8-oxo-dGTP、2-OH-dATP参与DNA复制、合成过程,导致基因突变频率增高、遗传稳定性破坏甚至细胞死亡[4, 6]。NudiX水解酶可水解这些异常核苷酸,阻止其掺入DNA链,提高DNA复制的准确性和稳定性[5]。而NUDT15功能缺失的细胞株暴露于异常核苷酸可导致突变频率增加1.5倍[6]。相对于同家族成员MTH1对于8-oxo-dGTP的强活性,NUDT15对GTP、dGTP,6-TGTP、6-TdGTP等底物有更高的活性,可将其水解为单磷酸盐;而后两者正是巯嘌呤在体内代谢的主要活性成分[6]。
NUDT15基因的变异主要有下列4种情况:(1)c.52G > A,导致缬氨酸-异亮氨酸改变;(2)c.36_37insGGAGTC,导致移码突变;(3)c.415C > T,导致精氨酸-半胱氨酸改变;(4)c.416G > A,导致精氨酸-组氨酸改变;其中,第2、3种情况可能存在连锁不平衡现象[21]。
目前c.415C > T变异受关注较多,但其造成6-MP不耐受的原因尚不明确。美国St. Jude儿童医院的研究[21]以TGTP/TdGTP为底物,检测不同突变NUDT15酶的催化效率,发现突变个体的酶催化效率较野生型有不同程度降低,以c.415C > T活性最低,c.52G > A、c.416G > A次之;c.36_37insGGAGTC和c.415C > T两种变异在同一序列的同时存在并没有使酶活性降到更低;而敲除NUDT15基因的细胞株具有更高的TGTP/TGMP比例、DNA-TG水平和细胞凋亡率,提示NUDT15通过对TGTP的水解影响巯嘌呤药物代谢,并且DNA-TG水平随着6-MP药物浓度提高而增加;硫鸟嘌呤、咪唑巯嘌呤等同一类别药物的试验也具有类似结果。
Valerie等[22]则发现NUDT15c.415C > T突变型与野生型在细胞外有类似的酶活性,且细胞内的mRNA表达水平持平,可能是突变改变了蛋白的空间结构及稳定性,使其在细胞内、人体内更易被分解破坏。c.415C > T造成了位于结合区的编码蛋白改变(Arg139Cys),与相邻的氨基酸残基(Cys140)形成二硫键,破坏了原本与α螺旋的GLU143氢键结合,使NUDT15的稳定性降低。
2.2 NUDT15基因多态性与巯嘌呤临床应用 2.2.1 药物耐受性NUDT15(c.415C > T)基因在亚裔人群的突变频率为8.5%~16%,远高于TPMT,亚裔人群对6-MP耐受更差可能与此相关[19, 23-25]。台湾地区的研究[23]发现,NUDT15野生型及杂合、纯合突变ALL患儿可耐受的6-MP剂量分别为(44.1±15.3)mg/m2、(30.7±11.7)mg/m2、(9.4± 5.7)mg/m2(P < 0.001),纯合突变的个体耐受剂量不足标准剂量(60 mg/m2)的20%,杂合突变的剂量则在50%左右。日本、泰国、印度等研究结果也类似[23-25]。NUDT15基因突变携带者更易发生粒细胞缺乏,且发生风险随着巯嘌呤使用时间增加有增高趋势[23, 25]。部分研究认为NUDT15与巯嘌呤的耐受性关系更密切,可用于预测巯嘌呤相关的不良反应,帮助临床调整巯嘌呤剂量[26-27]。
另外,TPMT与NUDT15在影响巯嘌呤耐受方面可能存在协同作用,存在任一基因杂合突变者对巯嘌呤敏感性提高,剂量需要降低50%以上;同时存在TPMT、NUDT15杂合突变个体的巯嘌呤耐受剂量低于仅有一个突变的个体[19]。
2.2.2 与预后的关系NUDT15基因多态性与巯嘌呤的耐受性密切相关,但与ALL预后的关系尚不明确。日本一项研究[24]表明,ALL患者中NUDT15 c.415C > T各基因型的复发率、5年无事件生存率的差异均没有统计学意义,但TC/TT突变组的生存率低于野生型。台湾地区的研究[23]中,NUDT15 CC、TC基因型的ALL患儿5年无事件生存率分别为(87.6±2.4)%、(90.2±3.8)%(P=0.478),复发率、治疗相关死亡率的差异也无统计学意义。
3 其他巯嘌呤代谢相关基因多态性 3.1 NT5C2NT5C2(cytosolic 5'-nucleotidase Ⅱ)属于5'-核苷酸酶(5'-nucleotidase, 5'-NT)家族,基因位于10q24.32,广泛表达于人类各组织细胞:在核酸代谢活跃的组织如肝、脾、淋巴结等活性高,在骨骼肌、红细胞等活性低。它既可从5'端水解单磷酸核苷GMP、IMP,也具有磷酸转移酶功能,参与调节和稳定核苷酸库,维持细胞的正常生理功能,对于抗肿瘤核苷类似物的单磷酸盐如6-TIMP、6-TGMP具有去磷酸化作用[28-29]。
研究认为,NT5C2突变可造成酶活性升高,与ALL复发尤其是早期复发相关,可能与影响抗肿瘤核苷类似物的代谢、导致巯嘌呤耐药有关[28, 30]。Tzoneva等[28]通过对ALL复发患者进行全外显子测序发现NT5C2多种突变,其中频率较高的为K359Q、R367Q、D407A,酶活性较野生型升高;而引入变异蛋白的细胞较野生型及空白对照组对巯嘌呤的耐受性不同程度提高。K359Q位于ATP与NT5C2结合的激活区附近,K359Q变异使得氨基酸残基355-364构成的α螺旋稳定性增强,继而Asp356取代Phe354形成催化中心而提高酶活性。
3.2 PRPS1PRPS1(phosphoribosyl pyrophosphate synthetase 1)基因位于Xq22-24,编码具有变构效应的磷酸核糖焦磷酸合成酶,它的产物磷酸核糖焦磷酸参与体内嘌呤、嘧啶的从头合成和补救合成,是该过程的重要调节因子。在正常细胞内,PRPS1活性受磷酸核糖焦磷酸的负反馈调节,维持在较低水平[31]。
Li等[31]发现,PRPS1突变可能与ALL早期复发相关:在358例ALL复发病人中,PRPS1变异频率为6.7%,以A190T、N144S较多见;而PRPS1突变在初发时未被检测到,在复发前呈指数式快速增长。突变的氨基酸残基多临近于变构结合区,可能影响抑制剂与酶相互作用,抑制磷酸核糖焦磷酸对酶活性的负反馈调节,使得嘌呤合成增多,胞内核苷酸浓度提高;代谢中间产物次黄嘌呤增多,竞争性抑制巯嘌呤向活性产物的代谢。所以PRSP1突变可能造成肿瘤细胞对巯嘌呤耐药,导致疾病复发;而敲除嘌呤从头合成途径相关酶的编码基因或应用嘌呤合成通路抑制剂,可解除PRPS1突变细胞的巯嘌呤耐受,这可能是ALL耐药病人个体化治疗的研究方向[31-33]。
3.3 三磷酸肌苷焦磷酸酶三磷酸肌苷焦磷酸酶(inosine triphosphate pyrophosphatase, ITPA)广泛存在于体内各组织器官,可水解肌苷三磷酸(inosine triphosphate, ITP)为肌苷一磷酸(inosine monophosphate, IMP)。在巯嘌呤代谢过程中,ITPA可水解6-TITP为6-TIMP,避免毒性产物6-TITP的堆积。
ITPA基因位于20号染色体,其变异在亚洲人群中较为常见,频率为11%~19%,白种人仅1%~2%[34-35]。ITPA基因突变主要有4个位点:c.94C > A、IVS2+21A > C、c.138G > A、IVS3+101G > A,均可造成ITPA酶活性下降,尤其是c.94C > A杂合突变者仅有22.5%残余酶活性,纯和突变则几乎无酶活性[34, 36]。ITPA酶活性低的个体更容易发生粒细胞缺乏、肝损害,或是流感样症状、皮疹、发热等[36]。即使根据TPMT调整巯嘌呤用药,ITPA酶活性缺乏者发生中性粒细胞减少伴发热的风险仍相对偏高[37]。另一方面,ITPA低活性导致肝功能损害可能与甲基化代谢产物6-MMPN增多有关[38]。而研究[37-39]发现,TPMT野生型伴ITPA突变者6-MMPN浓度最高,均野生型者次之,TPMT突变、ITPA野生型者则最低。说明肝损的发生可能是ITPA活性降低导致代谢产物6-TITP积累,后者又能生成6-MMPN,继而影响肝功能。
4 叶酸代谢相关酶基因多态性SAM是TPMT等甲基转移酶的重要甲基供体,与TPMT活性及蛋白表达相关。在体外,SAM可保证TPMT原始结构的稳定,减少TPMT降解[40]。生成SAM的蛋氨酸由同型半胱氨酸和5-甲基四氢叶酸合成,而后者是叶酸循环的最终产物,因此影响叶酸代谢的相关基因可能与SAM体内浓度有关,继而影响TPMT酶活性和巯嘌呤类药物的代谢[41]。
亚甲基四氢叶酸还原酶(methylene-tetrahydrofolate reductase, MTHFR)可将5, 10-亚甲基四氢叶酸还原为5-甲基四氢叶酸,后者与同型半胱氨酸一起形成蛋氨酸及SAM。MTHFR酶活性降低可导致SAM生成减少,从而影响TPMT活性及稳定性[40]。MTHFR位于1号染色体,与巯嘌呤代谢相关的主要是两个突变:c.677C > T(Ala222Val)和c.1298A > C(Glu429Ala),携带c.677C > T改变者更易发生血细胞减少而致用药中断,但不同基因突变者巯嘌呤耐受剂量并无明显差异[42-43]。
5 别嘌醇对巯嘌呤代谢的影响别嘌醇是一种黄嘌呤氧化酶抑制剂,上世纪60年代曾与6-MP联合治疗白血病,但由于毒副作用严重而被弃用,此后主要用于痛风的预防。90年代,小剂量别嘌醇联合咪唑嘌呤、环孢素成功地应用于1例肾移植患者的免疫抑制治疗,别嘌醇再一次引起人们的关注,并逐渐被应用于炎症性肠病患者[44]。近年亦有别嘌醇联合小剂量巯嘌呤用于ALL维持治疗的报道[45-47],发现巯嘌呤相关的肝功能损害、血糖异常、胰腺炎等不良反应减少,可能与别嘌醇的联合用药使巯嘌呤甲基化产物6-MMP减少、活性产物6-TGNs增加有关。别嘌醇还可能通过代谢产物巯基羟基嘌呤(thioxanthine, TX)抑制TPMT活性,提高HGPRT活性等途径发挥作用。Blaker等[48]发现别嘌醇、巯嘌呤联合应用后TX产生增加,且TX可抑制TPMT活性,造成6-MMP减少。Seinen等[44]监测6-MP联合别嘌醇应用于炎症性肠病患者的酶活性情况,发现12周后HGPRT活性明显升高,XO活性下降,但TPMT无明显变化。另外,也有认为在XO被抑制情况下,6-MP更多地经醛脱氢酶催化生成2-OHMP(2-hydroxy-6-mercaptopurine),后者可抑制TPMT活性[49]。
6 展望6-MP对于儿童ALL具有十分重要的意义,与6MP代谢相关的基因如TPMT、ITPA的多态性则与药物治疗反应的个体差异密切相关;NUDT15与亚裔人群对6-MP的耐受性也密切相关,NT5C2、PRPS1等基因与6-MP耐受及ALL的早期复发相关。而更多6-MP代谢相关基因的发现及基因多态性与药物代谢关系的研究将在未来更好地服务于临床。
| [1] | Schmiegelow K, Nielsen SN, Frandsen TL, et al. Mercaptopurine/methotrexate maintenance therapy of childhood acute lymphoblastic leukemia:clinical facts and fiction[J]. J Pediatr Hematol Oncol, 2014, 36 (7): 503–517. DOI:10.1097/MPH.0000000000000206 |
| [2] | Roberts RL, Barclay ML. Update on thiopurine pharmacogenetics in inflammatory bowel disease[J]. Pharmacogenomics, 2015, 16 (8): 891–903. DOI:10.2217/pgs.15.29 |
| [3] | Cai JP, Ishibashi T, Takagi Y, et al. Mouse MTH2 protein which prevents mutations caused by 8-oxoguanine nucleotides[J]. Biochem Biophys Res Commun, 2003, 305 (4): 1073–1077. DOI:10.1016/S0006-291X(03)00864-7 |
| [4] | Takagi Y, Setoyama D, Ito R, et al. Human MTH3(NUDT18) protein hydrolyzes oxidized forms of guanosine and deoxyguanosine diphosphates:comparison with MTH1 and MTH2[J]. J Biol Chem, 2012, 287 (25): 21541–21549. DOI:10.1074/jbc.M112.363010 |
| [5] | Song MG, Bail S, Kiledjian M. Multiple Nudix family proteins possess mRNA decapping activity[J]. RNA, 2013, 19 (3): 390–399. DOI:10.1261/rna.037309.112 |
| [6] | Carter M, Jemth AS, Hagenkort A, et al. Crystal structure, biochemical and cellular activities demonstrate separate functions of MTH1 and MTH2[J]. Nat Commun, 2015, 6 : 7871. DOI:10.1038/ncomms8871 |
| [7] | Ebbesen MS, Nersting J, Jacobsen JH, et al. Incorporation of 6-thioguanine nucleotides into DNA during maintenance therapy of childhood acute lymphoblastic leukemia-the influence of thiopurine methyltransferase genotypes[J]. J Clin Pharmacol, 2013, 53 (6): 670–674. DOI:10.1002/jcph.v53.6 |
| [8] | Fotoohi AK, Coulthard SA, Albertioni F. Thiopurines:factors influencing toxicity and response[J]. Biochem Pharmacol, 2010, 79 (9): 1211–1220. DOI:10.1016/j.bcp.2010.01.006 |
| [9] | Relling MV, Gardner EE, Sandborn WJ, et al. Clinical pharmacogenetics implementation consortium guidelines for thiopurine methyltransferase genotype and thiopurine dosing[J]. Clin Pharmacol Ther, 2011, 89 (3): 387–391. DOI:10.1038/clpt.2010.320 |
| [10] | Relling MV, Gardner EE, Sandborn WJ, et al. Clinical pharmacogenetics implementation consortium guidelines for thiopurine methyltransferase genotype and thiopurine dosing:2013 update[J]. Clin Pharmacol Ther, 2013, 93 (4): 324–325. DOI:10.1038/clpt.2013.4 |
| [11] | Wang L, Weinshilboum R. Thiopurine S-methyltransferase pharmacogenetics:insights, challenges and future directions[J]. Oncogene, 2006, 25 (11): 1629–1638. DOI:10.1038/sj.onc.1209372 |
| [12] | Li F, Wang L, Burgess RJ, et al. Thiopurine S-methyltransferase pharmacogenetics:autophagy as a mechanism for variant allozyme degradation[J]. Pharmacogenet Genomics, 2008, 18 (12): 1083–1094. DOI:10.1097/FPC.0b013e328313e03f |
| [13] | 李志玲, 王鹤尧, 孙华君. 巯嘌呤类药物用于儿童急性淋巴细胞性白血病患者个体化治疗的研究进展[J]. 上海医药, 2015, 36 (19): 12–15. |
| [14] | Schmiegelow K, Schrøder H, Gustafsson G, et al. Risk of relapse in childhood acute lymphoblastic leukemia is related to RBC methotrexate and mercaptopurine metabolites during maintenance chemotherapy. Nordic Society for Pediatric Hematology and Oncology[J]. J Clin Oncol, 1995, 13 (2): 345–351. DOI:10.1200/JCO.1995.13.2.345 |
| [15] | Bo J, Schrøder H, Kristinsson J, et al. Possible carcinogenic effect of 6-mercaptopurine on bone marrow stem cells:relation to thiopurine metabolism[J]. Cancer, 1999, 86 (6): 1080–1086. DOI:10.1002/(ISSN)1097-0142 |
| [16] | Schmiegelow K, Forestier E, Kristinsson J, et al. Thiopurine methyltransferase activity is related to the risk of relapse of childhood acute lymphoblastic leukemia:results from the NOPHO ALL-92 study[J]. Leukemia, 2009, 23 (3): 557–564. DOI:10.1038/leu.2008.316 |
| [17] | Levinsen M, Rotevatn EO, Rosthoj S, et al. Pharmacogenetically based dosing of thiopurines in childhood acute lymphoblastic leukemia:influence on cure rates and risk of second cancer[J]. Pediatr Blood Cancer, 2014, 61 (5): 797–802. DOI:10.1002/pbc.v61.5 |
| [18] | Yang SK, Hong M, Baek J, et al. A common missense variant in NUDT15 confers susceptibility to thiopurine-induced leukopenia[J]. Nat Genet, 2014, 46 (9): 1017–1020. DOI:10.1038/ng.3060 |
| [19] | Yang JJ, Landier W, Yang W, et al. Inherited NUDT15 variant is a genetic determinant of mercaptopurine intolerance in children with acute lymphoblastic leukemia[J]. J Clin Oncol, 2015, 33 (11): 1235–1242. DOI:10.1200/JCO.2014.59.4671 |
| [20] | 施逸怡, 杨深, 李凯歌, 等. NTP焦磷酸酶的生物学功能及临床相关性研究进展[J]. 生命科学, 2015, 27 (2): 113–119. |
| [21] | Moriyama T, Nishii R, Perez-Andreu V, et al. NUDT15 polymorphisms alter thiopurine metabolism and hematopoietic toxicity[J]. Nat Genet, 2016, 48 (4): 367–373. DOI:10.1038/ng.3508 |
| [22] | Valerie NC, Hagenkort A, Page BD, et al. NUDT15 hydrolyzes 6-Thio-DeoxyGTP to mediate the anticancer efficacy of 6-thioguanine[J]. Cancer Res, 2016, 76 (18): 5501–5511. DOI:10.1158/0008-5472.CAN-16-0584 |
| [23] | Liang DC, Yang CP, Liu HC, et al. NUDT15 gene polymorphism related to mercaptopurine intolerance in Taiwan Chinese children with acute lymphoblastic leukemia[J]. Pharmacogenomics J, 2016, 16 (6): 536–539. DOI:10.1038/tpj.2015.75 |
| [24] | Tanaka Y, Kato M, Hasegawa D, et al. Susceptibility to 6-MP toxicity conferred by a NUDT15 variant in Japanese children with acute lymphoblastic leukaemia[J]. Br J Haematol, 2015, 171 (1): 109–115. DOI:10.1111/bjh.2015.171.issue-1 |
| [25] | Chiengthong K, Ittiwut C, Muensri S, et al. NUDT15 c.415C>T increases risk of 6-mercaptopurine induced myelosuppression during maintenance therapy in children with acute lymphoblastic leukemia[J]. Haematologica, 2016, 101 (1): e24–e26. DOI:10.3324/haematol.2015.134775 |
| [26] | Shah SA, Paradkar M, Desai D, et al. Nucleoside diphosphate-linked moiety X-type motif 15 C415T variant as a predictor for thiopurine induced toxicity in Indian patients[J]. J Gastroenterol Hepatol, 2017, 32 (3): 620–624. DOI:10.1111/jgh.2017.32.issue-3 |
| [27] | Zhu X, Wang XD, Chao K, et al. NUDT15 polymorphisms are better than thiopurine S-methyltransferase as predictor of risk for thiopurine-induced leukopenia in Chinese patients with Crohn's disease[J]. Aliment Pharmacol Ther, 2016, 44 (9): 967–975. DOI:10.1111/apt.2016.44.issue-9 |
| [28] | Tzoneva G, Perez-Garcia A, Carpenter Z, et al. Activating mutations in the NT5C2 nucleotidase gene drive chemotherapy resistance in relapsed ALL[J]. Nat Med, 2013, 19 (3): 368–371. DOI:10.1038/nm.3078 |
| [29] | Galmarini CM, Jordheim L, Dumontet C. Role of IMP-selective 5'-nucleotidase (cN-Ⅱ) in hematological malignancies[J]. Leuk Lymphoma, 2003, 44 (7): 1105–1111. DOI:10.1080/1042819031000077142 |
| [30] | Meyer JA, Wang J, Hogan LE, et al. Relapse-specific mutations in NT5C2 in childhood acute lymphoblastic leukemia[J]. Nat Genet, 2013, 45 (3): 290–294. DOI:10.1038/ng.2558 |
| [31] | Li B, Li H, Bai Y, et al. Negative feedback-defective PRPS1 mutants drive thiopurine resistance in relapsed childhood ALL[J]. Nat Med, 2015, 21 (6): 563–571. DOI:10.1038/nm.3840 |
| [32] | Mullighan CG. Mutant PRPS1:a new therapeutic target in relapsed acute lymphoblastic leukemia[J]. Nat Med, 2015, 21 (6): 553–554. DOI:10.1038/nm.3876 |
| [33] | Thiopurine resistance in childhood ALL is mediated by PRPS1 mutations[J]. Cancer Discov, 2015, 5(7):693. |
| [34] | Marsh S, King CR, Ahluwalia R, et al. Distribution of ITPA P32T alleles in multiple world populations[J]. J Hum Genet, 2004, 49 (10): 579–581. DOI:10.1007/s10038-004-0183-y |
| [35] | Cao H, Hegele RA. DNA polymorphisms in ITPA including basis of inosine triphosphatase deficiency[J]. J Hum Genet, 2002, 47 (11): 620–602. DOI:10.1007/s100380200095 |
| [36] | Simone PD, Pavlov YI, Borgstahl GE. ITPA (inosine triphosphate pyrophosphatase):from surveillance of nucleotide pools to human disease and pharmacogenetics[J]. Mutat Res, 2013, 753 (2): 131–146. DOI:10.1016/j.mrrev.2013.08.001 |
| [37] | Adam de Beaumais T, Fakhoury M, Medard Y, et al. Determinants of mercaptopurine toxicity in paediatric acute lymphoblastic leukemia maintenance therapy[J]. Br J Clin Pharmacol, 2011, 71 (4): 575–584. DOI:10.1111/bcp.2011.71.issue-4 |
| [38] | Nygaard U, Toft N, Schmiegelow K. Methylated metabolites of 6-mercaptopurine are associated with hepatotoxicity[J]. Clin Pharmacol Ther, 2004, 75 (4): 274–281. DOI:10.1016/j.clpt.2003.12.001 |
| [39] | Stocco G, Cheok MH, Crews KR, et al. Genetic polymorphism of inosine triphosphate pyrophosphatase is a determinant of mercaptopurine metabolism and toxicity during treatment for acute lymphoblastic leukemia[J]. Clin Pharmacol Ther, 2009, 85 (2): 164–172. DOI:10.1038/clpt.2008.154 |
| [40] | Karas-Kuzelicki N, Milek M, Mlinaric-Rascan I. MTHFR and TYMS genotypes influence TPMT activity and its differential modulation in males and females[J]. Clin Biochem, 2010, 43 (1-2): 37–42. DOI:10.1016/j.clinbiochem.2009.09.003 |
| [41] | 谢偲, 岳丽杰. 基因多态性对巯嘌呤类药物作用影响的研究进展[J]. 中国临床药理学与治疗学, 2014, 19 (7): 818–824. |
| [42] | Tanaka Y, Manabe A, Nakadate H, et al. Methylenetetrahydrofolate reductase gene haplotypes affect toxicity during maintenance therapy for childhood acute lymphoblastic leukemia in Japanese patients[J]. Leuk Lymphoma, 2014, 55 (5): 1126–1131. DOI:10.3109/10428194.2013.825902 |
| [43] | Karas-Kuzelicki N, Jazbec J, Milek M, et al. Heterozygosity at the TPMT gene locus, augmented by mutated MTHFR gene, predisposes to 6-MP related toxicities in childhood ALL patients[J]. Leukemia, 2009, 23 (5): 971–974. DOI:10.1038/leu.2008.317 |
| [44] | Seinen ML, van Asseldonk DP, de Boer NK, et al. The effect of allopurinol and low-dose thiopurine combination therapy on the activity of three pivotal thiopurine metabolizing enzymes:results from a prospective pharmacological study[J]. J Crohns Colitis, 2013, 7 (10): 812–819. DOI:10.1016/j.crohns.2012.12.006 |
| [45] | Giamanco NM, Cunningham BS, Klein LS, et al. Allopurinol use during maintenance therapy for acute lymphoblastic leukemia avoids mercaptopurine-related hepatotoxicity[J]. J Pediatr Hematol Oncol, 2016, 38 (2): 147–151. DOI:10.1097/MPH.0000000000000499 |
| [46] | Zerra P, Bergsagel J, Keller FG, et al. Maintenance treatment with low-dose mercaptopurine in combination with allopurinol in children with acute lymphoblastic leukemia and mercaptopurine-induced pancreatitis[J]. Pediatr Blood Cancer, 2016, 63 (4): 712–715. DOI:10.1002/pbc.25841 |
| [47] | Brackett J, Schafer ES, Leung DH, et al. Use of allopurinol in children with acute lymphoblastic leukemia to reduce skewed thiopurine metabolism[J]. Pediatr Blood Cancer, 2014, 61 (6): 1114–1117. DOI:10.1002/pbc.v61.6 |
| [48] | Blaker PA, Arenas-Hernandez M, Smith MA, et al. Mechanism of allopurinol induced TPMT inhibition[J]. Biochem Pharmacol, 2013, 86 (4): 539–547. DOI:10.1016/j.bcp.2013.06.002 |
| [49] | Blaker PA, Arenas M, Fairbanks L, et al. A biochemical mechanism for the role of allopurinol in TMPT inhibition[J]. Gastroenterology, 2011, 140 (5): S–769. |
 2017, Vol. 19
2017, Vol. 19

