呼吸道合胞病毒(respiratory syncytial virus, RSV)是世界范围内婴幼儿下呼吸道感染(lower respiratory tract infection, LRTI)最主要的病原,至今尚无安全有效的疫苗[1]。RSV是非节段性、单股负链RNA病毒,包括10个蛋白编码基因表达11种蛋白,其中G蛋白是最易发生抗原变异的包膜糖蛋白。病毒发生抗原变异后可逃避宿主的免疫攻击,是导致RSV感染暴发和重复感染的重要原因之一[2],G蛋白基因的变异及进化特征已成为RSV分子流行病学的研究热点。本研究对浙江南部3个地区5个连续流行年度住院儿童进行RSV检测及亚型分型,并对RSV A亚型G蛋白进行基因测序及进化分析,探讨A亚型基因型分布及其G蛋白基因变异特征,为RSV疾病防控及疫苗研制提供参考。
1 资料与方法 1.1 研究对象及标本采集以2009年7月1日至2014年6月30日,浙江省温州、台州及丽水地区3家三级甲等医院因肺炎和毛细支气管炎住院的5岁以下患儿为研究对象。患儿入院48 h内取鼻咽分泌物约1 mL置于2 mL生理盐水中,应用直接免疫荧光法(DIF)筛查RSV抗原。按照简单随机抽样法,每个流行年度(以每年的7月1日到次年6月30日)抽取RSV抗原阳性标本200份以检测RSV亚型。
1.2 主要仪器及设备DIF试剂购自美国Chemicon公司;RNA提取试剂盒购自北京博凌科为生物科技有限公司,逆转录试剂盒均购自于TaKaRa(大连)有限公司;PCR MasterMix购自北京天根科技有限公司;DNAMarker购自上海捷瑞生物有限公司;ABI-PCR仪购自美国ABI公司;电泳仪BIO-RAD power/PAC 3000型购自北京六一仪器厂。
1.3 实验方法(1)病毒总RNA提取及反转录:取标本上清200 μL,采用RNA提取试剂盒提取患儿标本上清中的RSV RNA。反转录采用TaKaRa公司的试剂盒进行。以反转录体系20 μL为例:取RNA 5 μL,按照说明书加入5xPrimeScript®Buffer 4 μL,PrimeScript®RT Enzyme Mix I、Oligo dTPrime各1 μL、Random 6 mers 4 μL,取RNA 4 μL,加入RNase free dH2O补足至20 μL;反应条件为37℃逆转录反应15 min,85℃逆转酶失活5 s;-20℃保存。
(2)RSV亚型鉴定:引物设计及扩增反应参照文献[3]。第一步PCR扩增产物覆盖G蛋白全基因序列,反应体系(40 μL):cDNA模板2 μL,P1、P2各1 μL,2×PCR Master Mix 20 μL,ddH2O 16 μL;反应条件为:预变性94℃、3 min;94℃、60℃、72℃各1 min扩增35个循环;72℃延伸7 min,4℃保存。第二步PCR扩增的产物长度为250 bp的A亚型基因片段或长度为341 bp的B亚型基因片段,反应体系(20 μL):PCR第一步产物1.0 μL,P3、P4、P5各0.5 μL,2×PCR Master Mix 10 μL,ddH2O 7.5 μL;反应条件为:94℃预变性5 min;94℃、50℃各30 s,70℃、40 s,扩增30个循环;72℃延伸7 min,4℃保存。取第二步PCR产物5 μL,加入制备的2%琼脂糖凝胶加样孔,电泳缓冲液为1×TBE 100 V恒压,电泳30~40 min,阴性对照为ddH2O,阳性对照为标准株A2株(首都儿科研究所馈赠)及CH18537株(购自美国ATCC),电泳结束后将琼脂糖凝胶放入凝胶成像系统拍照并记录结果。
(3)基因测序:使用SPSS 19.0统计软件自带抽样功能,每个流行年度随机抽取25株A亚型G蛋白基因序列产物(依次编号为WENZHOUA1-WENZHOUA125等),送上海英骏公司完成测序。
(4)系统进化分析:使用DNAstar软件中EditSeq和MegAlign等方法,对A亚型的核苷酸和氨基酸序列与GenBank中已有RSV原型株的序列进行比较和分析,并在MEGA6.06软件上应用N-J法绘制基因进化树进行分析,比较方法采用Clustal W法。
1.4 临床资料收集由专人收集所有RSV阳性患儿的临床资料,包括性别、年龄、发病季节、临床诊断、诊治经过等。
1.5 统计学分析采用SPSS 19.0统计软件对数据进行统计学分析,计数资料以例数和百分率(%)表示,组间比较采用卡方检验。计量资料以均数±标准差(x±s)表示。P < 0.05为差异有统计学意义。
2 结果 2.1 RSV亚型的检出情况2009~2014年度共送检25 449份下呼吸道分泌物标本,其中6 416份(25.21%)标本RSV阳性,各年度检出率依次为20.31%(941/4 631)、26.37%(1 184/4 490)、36.79%(1 799/4 890),20.96%(1 071/5 109)和22.45%(1 421/6 329)。
所抽取的1 000份RSV阳性标本经过巢氏PCR扩增共检出A亚型株有462株(46.2%),B亚型株有538株(53.8%)。A亚型依次占2009~2014年各年度的22.5%(45/200)、74.5%(149/200)、84.5%(169/200)、19.0%(38/200)、30.5%(61/200),B亚型依次占各年度的77.5%(155/200)、25.5%(51/200)、15.5%(31/200)、81.0%(162/200)、69.5%(139/200)。结果显示2010~2012年度中,与B亚型相比,A亚型占主导,差异具有统计学意义(P < 0.05)。图 1示2009~2014年5个年度1 000份阳性标本RSV亚型检出数及不同月份RSV亚型检出情况,可见每年的12月到次年的3~4月,RSV检出阳性率最高,其中2010~2012两个年度RSV以A亚型流行为主,其余3个年度均以B亚型流行为主。
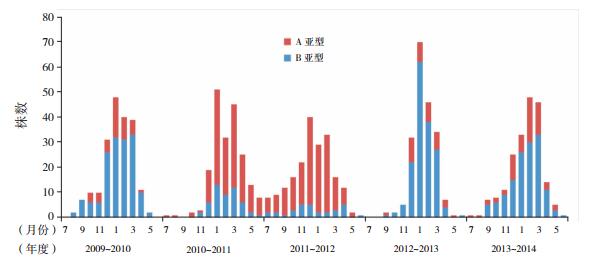
|
图 1 2009~2014年度RSV亚型检出情况 显示2010~2012两个年度RSV以A亚型流行为主,其余3个年度均以B亚型流行为主。 |
462例A亚型感染患儿中,男307例(66.5%),女155例(33.5%),男女比为1.98 : 1;其中小于6月龄258例(55.8%),6月龄至2岁184例(39.8%),2岁以上至5岁20例(4.3%)。感染发生在每年的12月至次年4月有354例(76.6%)。临床诊断为毛细支气管炎261例(56.5%),支气管肺炎201例(43.5%)。145例(31.4%)有低热,151例(32.7%)出现呼吸费力,12例有肝功能损害,8例合并心功能不全。89例有基础疾病,64例需吸氧,入住重症监护室41例,机械通气14例。
538例B亚型感染患儿中,男360例(66.9%),女178例(33.1%),男女比为2.02 : 1;其中小于6月龄302例(56.1%),6月龄至2岁234例(43.5%),2岁以上至5岁2例(0.4%)。感染发生在每年的12月至次年4月有413例(76.8%)。临床诊断为毛细支气管炎333例(61.9%),支气管肺炎205例(38.1%)。236例(43.9%)有低热,198例(36.8%)出现呼吸费力,15例有肝功能损害,10例合并心功能不全。112例有基础疾病,82例需吸氧,入住重症监护室23例,机械通气6例。
2.3 RSV亚型的基因型分布每个流行年度随机抽取25株A亚型基因共125株进行测序,获得52株A亚型G蛋白基因序列,对第二高变区(HVR2)区域进行基因序列比对及构建系统进化树,结果显示A亚型基因型分布于4个分枝,其中39株基因序列与NA1原型株NG-016-04的同源性达94.1%~99.6%,属于NA1基因型,均于2009~2013年度检出,是这4个流行年度的优势基因型。10株HVR2含有72个重复核苷酸序列插入,与ON1原型株ON138-0111A的同源性达98.2%~100%,属于ON1基因型,其中首株(WENZHOUA68)于2011年12月检出,其余9株均于2013~2014年度检出,并且是该年度A亚型唯一的基因型。2株(WENZHOUA15、WENZHOUA22)与GA2原型株CH28的同源性达99.3%,属GA2基因型,于2009~2010年度检出。WENZHOUA64与原型株BJ/36578的同源性达99.3%,属NA4基因型,于2011~2012年度检出。B亚型中以BA9基因型最多(44/60,73.3%),5个流行年度均有检出,是除2010~2011年度外B亚型的优势基因型,并且是2013~2014年度B亚型唯一基因型,其次为BA8基因型(6/60,10.0%)。本研究将针对A亚型及其各类基因型进行分析与讨论,结果详见表 1、图 2。
| 表 1 2009~2014 5个流行年度A亚型基因型检出分布 |
|
|
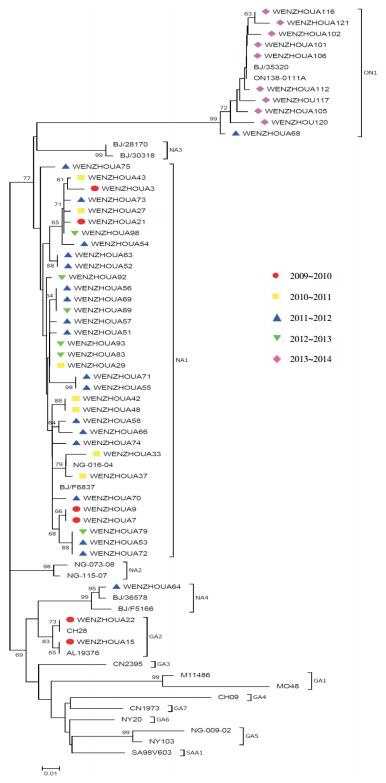
|
图 2 A亚型G蛋白系统进化树 依据RSV A亚型HVR2构建无根进化树,采用neighbor-joining方法,每种基因型的参考序列来自GenBank; Bootstrap 1 000次,计算方法采用Kimura 2-parameter model,标尺表示遗传距离,进化树分枝上仅显示Bootstrap value ≥ 60%的数值。显示A亚型基因型分布于4个分枝,其中39株属于NA1基因型,10株属于ON1基因型,2株属于GA2基因型,1株属于NA4基因型。 |
A亚型G蛋白基因与原型株A2的核苷酸同源性为80.7%~89.3%,氨基酸同源性为74.4%~82.6%;52株A亚型之间的核苷酸及氨基酸同源性分别为81.5%~100%和80.2%~100%。
NA1和ON1基因终止密码子全部为TGA,GA2和NA4基因终止密码子均为TAG。通过氨基酸序列比对发现,N237糖基化存在于NA1和GA2基因型中;N251糖基化存在于除WENZHOUA53、72、79外的其他NA1基因型中;除WENZHOUA42、48外,其他A亚型均存在N294糖基化。ON1基因型插入的23个重复氨基酸与原型株之间也存在差异,WENZHOU112第262位、WENZHOU121第271位均发生E突变为K(E262K、E271K),WENZHOU105第295位发生S突变为N(S295N),但其对应位点286、295、271没有改变。也有对应氨基酸位点同时突变为同一个氨基酸残基的,如WENZHOU117氨基酸第274位点L突变为P,其对应第298位点也发生L突变为P。此外,HVR2其他位点的突变如E232G、T253K存在于所有ON1基因株中。WENZHOUA64替代包括N242I、P274Q、T293S、N251D。RSV A亚型G蛋白氨基酸序列比较见图 3。

|
图 3 RSV A亚型G蛋白氨基酸序列比较 显示N237糖基化存在于NA1和GA2基因型中,ON1基因型插入的23个重复氨基酸与原型株之间也存在差异,包括突变、替代。E:谷氨酸;K:赖氨酸;S:丝氨酸;N:天冬酰胺;L:亮氨酸;P:脯氨酸;G:甘氨酸;T:苏氨酸;I:异亮氨酸;Q:谷氨酰胺;D:天冬氨酸。 |
RSV感染可全年散发但以冬春季为主,流行常在每年的11月或12月开始,1月或2月达高峰,持续4~5个月[4-5]。本研究结果显示浙江南部地区儿童RSV感染流行季节为每年的12月到次年的3~4月,与Yoshihara等[6]报道一致。2009~2014各流行年度均可检出A亚型,其中A亚型是2010~2012两个流行年度的优势亚型,与北京、上海、重庆的报道一致[7-9]。然而深圳市2012~2013流行年度中以B亚型为主[10],武汉市2014~2015流行年度中以A亚型检出为多[11],北京地区2006~2016流行年度中,大部分流行年度以一个亚型为主,但也有两个流行年度为A、B亚型共同主导[12]。显示不同流行年度我国不同地区不同气候下RSV优势亚型存在差异,因此有必要连续监测本地区RSV亚型流行趋势,为预防和控制RSV感染提供指导,为筛选疫苗株提供参考。
本研究在2009~2014连续5个流行年度共检出4种A亚型基因型(NA1、NA4、ON1、GA2),其中前4个流行年度的优势基因型为NA1,最后一个流行年度仅检出ON1基因型,表明存在不同RSV A亚型基因型的共循环。NA1基因型曾经是多个地区A亚型的优势基因型[6-7, 10, 12-13]。然而自2010年加拿大首次检出ON1基因型后[14],ON1基因型已成为部分国家A亚型的优势基因型[11, 15-16]。韩国学者研究发现,2010~2013连续3个流行年度中ON1基因型依次占A亚型的0.0%、17.4%、94.6%,表明ON1基因型存在快速传播趋势[16]。ON1基因型也是意大利2010~2013流行年度A亚型的优势基因型,其快速传播可能由G蛋白变异所致[17]。ON1基因型在避免当前宿主免疫方面具有优势[18]。研究ON1基因型的传播对于了解RSV遗传变异对其流行病学特征的影响非常重要[11]。国内多地也于2011~2012年度检出少量ON1基因型[7-9],Cui等[18]报道ON1已快速取代其他基因型成为2012~2014年度北京地区的优势基因型。本研究于2011年12月在浙江南部地区首次检出1株ON1型RSV菌株,2013~2014年度检出数增至9株,并且是A亚型菌株唯一的基因型,表明ON1已成为本地区该流行年度的优势基因型,有必要进一步监测未来ON1基因型的流行趋势。
各地检出的RSV亚型株之间核苷酸及氨基酸的同源性有明显差异,其中北京分离株之间核苷酸的同源性在87.8%~100%之间, 氨基酸同源性在77.9%~100%之间[19],重庆A亚型分离株之间核苷酸序列和氨基酸序列的差异分别为13%~14.8%和20.5%~26.1%[18]。本研究显示2009~2014浙江南部地区RSV A亚型分离株之间核苷酸及氨基酸的同源性分别为80.7%~89.3%和74.4%~82.6%,分离株之间的遗传变异较大,表明2009~2014年度有多个不同的RSV A亚型基因型毒株在浙江南部地区共循环。
RSV A亚型G蛋白核苷酸的变异并同时存在氨基酸变异。NA4基因型的变异特点是N242I、P274Q和T293S的替代突变,WENZHOUA64属于NA4基因型,与原型株BJ/36578的同源性达99.3%,显示两者有非常近的亲缘关系[7],这些变异可使其逃避宿主的免疫攻击,导致反复感染[20]。ON1基因型的变异特点则是G蛋白C末端23个重复氨基酸序列的插入,经过与ON1原型株(ON67-1210A、JN257693)及GenBank中的代表株比对,本研究中ON1基因型插入的重复氨基酸也存在替代突变,如WENZHOU67(L274P、L298P),WENZHOU62(E262K)和WENZHOU71(E271K)的突变,但其对应位点286、295却没有发生突变。ON1基因型的替代突变与欧洲学者的报道相似[16-17]。ON1基因型插入片段已经发生了核苷酸的替换,这些变异可能帮助病毒逃逸,可能是ON1基因型取代其他基因型成为优势基因型的原因,这一特征对提供人群免疫力的疫苗具有潜在影响,在确定疾病严重程度中的作用需要进一步研究[20]。另外,Kim等[17]报道了8株ON1基因型的框移突变,即G蛋白HVR2缺失1个核苷酸,从而导致比其他的ON1基因型多两个氨基酸残基,但本研究未发现上述突变。G蛋白HVR2的变异是RSV逃避宿主免疫的基础,也可能是亚型内新基因型流行的原因[7]。NA1基因型的变异特点是N糖基化位点突变,N237、N250糖基化位点消失[21],但本研究检出10株存在N237糖基化位点的NA1基因型,与北京地区报道一致[7]。有学者认为G蛋白N-糖基化位点的变异最终使病毒的抗原性改变,是病毒进化的优势[16],基于N-糖基化位点的阳性选择可能在驱动病毒多样性中发挥重要作用[22]。
本研究针对浙江南部地区不同城市RSV分子流行病学研究,地域较为局限,且由于课题经费有限,外送基因型检测的样本量偏小,因此有必要进一步监测和测序RSV病毒株,以确定这些优势基因型是否仍在本地区流行,是否这些优势基因型会持续主导或随时间推移而逐年波动。
综上所述,RSV A亚型为浙江南部地区2010~2012流行年度的主导亚型,存在多种优势基因型共循环。ON1基因型已成为2013~2014流行年度的优势基因型。A亚型变异显著,包括替代、插入和重复突变。
| [1] |
Vanover D, Smith DV, Blanchard EL, et al. RSV glycoprotein and genomic RNA dynamics reveal filament assembly prior to the plasma membrane[J]. Nat Commun, 2017, 8(1): 667. DOI:10.1038/s41467-017-00732-z (  0) 0) |
| [2] |
Fuentes S, Coyle EM, Beeler J, et al. Antigenic fingerprinting following primary RSV infection in young children identifies novel antigenic sites and reveals unlinked evolution of human antibody repertoires to fusion and attachment glycoproteins[J]. PLoS Pathog, 2016, 12(4): e1005554. DOI:10.1371/journal.ppat.1005554 (  0) 0) |
| [3] |
丁雅馨, 田润, 钱渊, 等. 人呼吸道合胞病毒快速抗原检测方法的床旁检测适用性探讨[J]. 中华儿科杂志, 2017, 55(2): 139-143. DOI:10.3760/cma.j.issn.0578-1310.2017.02.020 (  0) 0) |
| [4] |
Noveroske DB, Warren JL, Pitzer VE, et al. Local variations in the timing of RSV epidemics[J]. BMC Infect Dis, 2016, 16(1): 674. DOI:10.1186/s12879-016-2004-2 (  0) 0) |
| [5] |
Pitzer VE, Viboud C, Alonso WJ, et al. Environmental drivers of the spatiotemporal dynamics of respiratory syncytial virus in the United States[J]. PLoS Pathog, 2015, 11(1): e1004591. DOI:10.1371/journal.ppat.1004591 (  0) 0) |
| [6] |
Yoshihara K, Le MN, Okamoto M, et al. Association of RSV-A ON1 genotype with increased pediatric acute lower respiratory tract infection in Vietnam[J]. Sci Rep, 2016, 6: 27856. DOI:10.1038/srep27856 (  0) 0) |
| [7] |
Cui G, Zhu R, Qian Y, et al. Genetic variation in attachment glycoprotein genes of human respiratory syncytial virus subgroups A and B in children in recent five consecutive years[J]. PLoS One, 2013, 8(9): e75020. DOI:10.1371/journal.pone.0075020 (  0) 0) |
| [8] |
Liu J, Mu Y, Dong W, et al. Genetic variation of human respiratory syncytial virus among children with fever and respiratory symptoms in Shanghai, China, from 2009 to 2012[J]. Infect Genet Evol, 2014, 27: 131-136. DOI:10.1016/j.meegid.2014.07.011 (  0) 0) |
| [9] |
Ren L, Xiao Q, Zhou L, et al. Molecular characterization of human respiratory syncytial virus subtype B:a novel genotype of subtype B circulating in China[J]. J Med Virol, 2015, 87(1): 1-9. DOI:10.1002/jmv.v87.1 (  0) 0) |
| [10] |
吴春利, 陈洁润, 阳帆, 等. 2012~2013年深圳市呼吸道合胞病毒流行规律及分子变异分析[J]. 中华微生物学和免疫学杂志, 2014, 34(10): 737-741. DOI:10.3760/cma.j.issn.0254-5101.2014.10.002 (  0) 0) |
| [11] |
董琳, 歹丽红, 樊节敏, 等. 浙南地区下呼吸道感染儿童呼吸道合胞病毒基因型流行病学特征及与病情的关系[J]. 中华儿科杂志, 2015, 53(7): 537-541. DOI:10.3760/cma.j.issn.0578-1310.2015.07.014 (  0) 0) |
| [12] |
张拓慧, 邓洁, 钱渊, 等. 毛细支气管炎患儿呼吸道合胞病毒分子生物学及临床特征分析[J]. 中华儿科杂志, 2017, 55(8): 586-592. DOI:10.3760/cma.j.issn.0578-1310.2017.08.008 (  0) 0) |
| [13] |
Ren L, Xia Q, Xiao Q, et al. The genetic variability of glycoproteins among respiratory syncytial virus subtype A in China between 2009 and 2013[J]. Infect Genet Evol, 2014, 27: 339-347. DOI:10.1016/j.meegid.2014.07.030 (  0) 0) |
| [14] |
Martinelli M, Frati ER, Zappa A, et al. Phylogeny and population dynamics of respiratory syncytial virus (RSV) A and B[J]. Virus Res, 2014, 189: 293-302. DOI:10.1016/j.virusres.2014.06.006 (  0) 0) |
| [15] |
Eshaghi A, Duvvuri VR, Lai R, et al. Genetic variability of human respiratory syncytial virus A strains circulating in ontario:a novel genotype with a 72 nucleotide G gene duplication[J]. PLoS One, 2012, 7(3): e32807. DOI:10.1371/journal.pone.0032807 (  0) 0) |
| [16] |
Prifert C, Streng A, Krempl CD, et al. Novel respiratory syncytial virus A genotype, Germany, 2011-2012[J]. Emerg Infect Dis, 2013, 19(6): 1029-1030. DOI:10.3201/eid1906.121582 (  0) 0) |
| [17] |
Kim YJ, Kim DW, Lee WJ, et al. Rapid replacement of human respiratory syncytial virus A with the ON1 genotype having 72 nucleotide duplication in G gene[J]. Infect Genet Evol, 2014, 26: 103-112. DOI:10.1016/j.meegid.2014.05.007 (  0) 0) |
| [18] |
Cui G, Zhu R, Deng J, et al. Rapid replacement of prevailing genotype of human respiratory syncytial virus by genotype ON1 in Beijing, 2012-2014[J]. Infect Genet Evol, 2015, 33: 163-168. DOI:10.1016/j.meegid.2015.04.025 (  0) 0) |
| [19] |
Zhang ZY, Du LN, Chen X, et al. Genetic variability of respiratory syncytial viruses (RSV) prevalent in Southwestern China from 2006 to 2009:emergence of subgroup B and A RSV as dominant strains[J]. J Clin Microbiol, 2010, 48(4): 1201-1207. DOI:10.1128/JCM.02258-09 (  0) 0) |
| [20] |
Otieno JR, Kamau EM, Agoti CN, et al. Spread and evolution of respiratory syncytial virus A genotype ON1, Coastal Kenya, 2010-2015[J]. Emerg Infect Dis, 2017, 23(2): 264-271. DOI:10.3201/eid2302.161149 (  0) 0) |
| [21] |
Palomo C, Cane PA, Melero JA. Evaluation of the antibody specificities of human convalescent-phase sera against the attachment (G) protein of human respiratory syncytial virus:influence of strain variation and carbohydrate side chains[J]. J Med Virol, 2000, 60(4): 468-474. DOI:10.1002/(ISSN)1096-9071 (  0) 0) |
| [22] |
Do LA, Wilm A, Van Doorn HR, et al. Direct whole-genome deep-sequencing of human respiratory syncytial virus A and B from Vietnamese children identifies distinct patterns of interand intra-host evolution[J]. J Gen Virol, 2015, 96(12): 3470-3483. DOI:10.1099/jgv.0.000298 (  0) 0) |
 2018, Vol. 20
2018, Vol. 20


