Cornelia de Lange综合征(CdLS;MIM #122470,300590,610759,300882,614701)又称为Brachmann de Lange综合征,是一类罕见的累及多器官系统的遗传异质性疾病,发病率估计为1 : 10 000到1 : 30 000[1-4]。多为散发病例,无家族史。
目前已报道的Cornelia de Lange综合征相关基因有NIPBL、SMC1A、SMC3、HDAC8、RAD21、EP300和ANKRD11 7个,其中NIPBL基因突变所致占50%[5-6]。本研究采用基因捕获测序技术对2例临床提示Cornelia de Lange综合征患儿的7个相关基因进行检测,并应用Sanger测序法对突变基因的家系分布进行分析和验证,发现2种未见报道的NIPBL基因新突变。
1 资料与方法 1.1 研究对象患儿1:男,3个月,因生长缓慢就诊。患儿无反复发热及呕吐、腹泻、抽搐等。患儿为第一胎、第一产,足月出生,出生身长48 cm(-1.3 SD)、体重3.0 kg(-0.8 SD),Apgar评分不详。母乳喂养,吃奶正常,睡眠正常。新生儿期发现房间隔缺损、肺动脉瓣狭窄。患儿父母健康,否认近亲结婚,家族中无类似患者。体格检查:身长53 cm(-2.5 SD),体重5.7 kg(-1.5 SD)。毛发浓密,浓眉、弓形眉、连心眉,睫毛长且弯曲,短鼻,上唇薄、唇角下斜,小下颌,前囟2.5 cm×2.5 cm,心脏听诊未闻及杂音,双手通贯掌,双手小指均为两个指节。心脏彩超提示房间隔缺损、肺动脉狭窄。
患儿2:女,45 d,以体重不增就诊。患儿无反复发热及呕吐、腹泻、抽搐等。出生胎龄36+5周,出生体重1.65 kg,出生无窒息史。生后发现腭裂。双耳听力筛查未通过。早产儿奶粉人工喂养,吃奶正常,大便正常,睡眠正常。患儿父母健康,否认近亲结婚,家族中无类似患者。体格检查:身长43.1 cm(-5 SD),体重1.8 kg(-5 SD)。头围30.2 cm(-3 SD)。毛发浓密,弓形眉、连心眉,睫毛长且弯曲,短鼻,上唇薄、唇角下斜,腭裂,小下颌。前囟2.5 cm×2.5 cm,心肺听诊无异常,腹软、肝脾不大,双小指内弯,无特殊皮纹。患儿4月龄时复诊,身长47.0 cm(-6.2 SD)、体重2.6 kg(-5.3 SD);左耳客观听阈中度增高,脑干听觉诱发电位听神经和中脑神经传导速度减低;7月龄时身长53.1 cm(-5.9 SD)、体重3.1 kg(-5.8 SD),双耳客观听阈均增高提示患儿中重度耳聋,脑干听觉诱发电位听神经及脑桥和中脑神经传导速度减低。
1.2 高通量基因捕获测序、突变验证及分析采集患儿及其父母空腹静脉血各2 mL(EDTA抗凝),采用QIAamp DNA提取试剂盒(QIAGEN公司)提取基因组DNA,DNA酶片段化后用磁珠法纯化,随后进行PCR扩增并连接接头序列,经定制的Panel探针(illumina Inc, USA)两次捕获及纯化,经PCR扩增和纯化,获得最终文库,在NextSeq500测序仪(illumina Inc, USA)上对Panel基因NIPBL、SMC1A、SMC3、HDAC8、RAD21、EP300和ANKRD11的外显子区进行测序。对可疑候选突变的位点设计PCR引物进行扩增及进行Sanger测序验证,并对患儿父母的相应位点进行检测(广州金域医学检验中心完成)。患儿1测序平均深度176.14,覆盖率≥20X为98.19%;患儿2测序平均深度114,覆盖率≥10X为97.65%。引物设计根据GeneBank人类NIPBL基因序列(NG_006987.1),见表 1。
| 表 1 NIPBL基因引物序列 |
|
|
本研究获得中国医科大学附属盛京医院伦理审查及患儿父母知情同意。
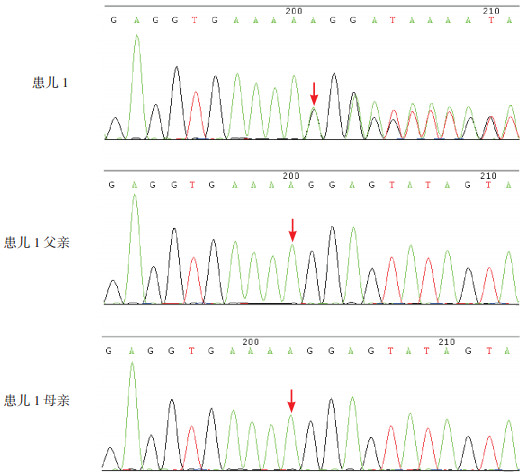
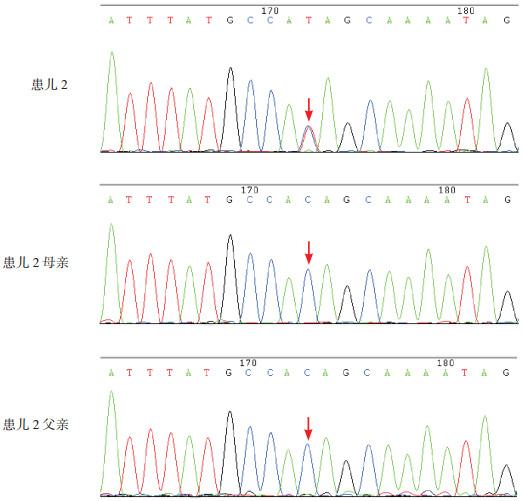
2 结果高通量捕获测序发现患儿1在NIPBL基因第45外显子发生移码突变,Exon 45 R2612fsX*20(c.7834dupA),导致突变位点后第20个密码子出现终止密码子TGA、翻译提前终止、蛋白截短。应用PCR-Sanger测序进行患儿突变验证及其父母和50例无关正常个体的检测,证实患儿父母及正常对照的该基因位点正常。见图 1。患儿2在NIPBL基因检测到一个杂合的无义突变,Q169X(c.505C > T),位于第6外显子,导致蛋白截短。应用Sanger测序进行患儿突变验证及父母分析、50例无关正常个体的检测,患儿父母及正常对照的该基因位点正常。见图 2。
|
图 1 患儿1及其父母的NIPBL基因测序 患儿1在第45外显子发生碱基插入:c.7834dupA,突变位点如箭头所示。其父母该位点正常。 |
|
图 2 患儿2及其父母的NIPBL基因测序 患儿2在第6外显子发生碱基置换,c.505C > T,突变位点如箭头所示,其父母该位点正常。 |
经查询HGMD(Human Gene Mutation Database)数据库以及ESP6500si v2_ALL、千人基因组(1 000 g 2015aug_ALL)、dbSNP147数据库,NIPBL基因的c.7834dupA和c.505C > T两种突变均未见报道和收录,并且在50例无关健康个体也未发现这两个位点突变,因此本文检测到的两种NIPBL基因突变可能为致病性突变。
3 讨论Cornelia de Lange综合征是一种先天发育异常综合征,是一类罕见的累及多器官系统的遗传异质性疾病。该综合征典型特征包括(1)特殊面容:弓形眉、连体眉,睫毛长且弯曲;上睑下垂,眼球震颤;短鼻、鼻孔前倾;小下颌,腭弓高,上唇薄,口角向下弯。(2)生长迟缓:包括宫内生长迟缓以及身材矮小。(3)多毛。(4)头围小且扁。(5)智力发育落后,20%的患儿有抽搐。(6)肢体发育异常:手臂细小,短肢或缺指,肘部弯曲挛缩。(7)心脏及其他畸形:心脏畸形以室间隔缺损多见,其他畸形包括幽门狭窄、肠旋转不良、隐睾、尿道下裂。(8)其他:听力丧失,孤独症样行为。
本研究两例均在婴儿期因生长缓慢就诊。婴儿期导致体重不增、生长迟缓的疾病复杂多样,包括消化系统疾病、遗传代谢性疾病、染色体病等,临床诊断存在一定困难。但两例患儿均具有特殊面容如毛发浓密、弓形眉、连体眉,睫毛长且弯曲,以及短鼻、小下颌;患儿1伴有先心病(房间隔缺损、肺动脉瓣狭窄)和特殊皮纹,患儿2有腭裂、中重度耳聋,因此均高度提示Cornelia de Lange综合征。值得注意的是,患儿2的听力障碍随年龄增长逐渐加重,提示对于Cornelia de Lange综合征患儿有必要进行听力的动态监测,以及时发现听力障碍。
已报道的与Cornelia de Lange综合征发病相关基因包括NIPBL、SMC1A、SMC3、HDAC8、RAD21、EP300和ANKRD11 7个基因,其中NIPBL基因突变所致的占50%[5-6]。NIPBL基因位于5p13.2,目前发现的突变包括所有突变类型,如缺失、重复、错义、无义、剪接、插入等,以截短突变表型最为严重,剪接突变的表型严重程度为中度,而错义突变的表型相对较轻。钟秋连[7]报道两例Cornelia de Lange综合征,均存在已报道的NIPBL基因突变:c.4321G > T和新发突变:c.6589+5G > C。杨保旺等[8]报告一例具有典型特征的Cornelia de Lange综合征,存在NIPBL基因无义突变c.7176T > A,p.Cys2392Ter。本研究两例患儿均检测到国内外尚未报道的NIPBL基因突变,可能为新发致病突变。
SMClA基因定位在X染色体,SMClA基因突变所致的Cornelia de Lange综合征占5%[9]。SMC3基因定位于10号染色体[10]。HDAC8和RAD21基因相继于2012和2013年被发现(OMIM#300882,#614701)[11-12]。NIPBL、HDAC8基因是粘连蛋白调控基因,SMC1A、SMC3和RAD21基因编码粘连蛋白。粘连蛋白的功能主要是调控细胞有丝分裂和减数分裂期姐妹染色体单体凝聚,同时在基因表达、染色质结构变化和发育调节等方面起到重要作用。
2014年Woods等[13]报道一例多发先天畸形、智力发育迟缓病例,其典型的特殊面容和复杂表型提示Cornelia de Lange综合征可能,NIPBL、SMC1A、SMC3、HDAC8、RAD21 5个基因突变检测均为阴性,但全外显子测序发现EP300基因一个新发突变。EP300基因与HDAC8基因均为粘连蛋白调控基因,提示EP300基因突变也可能与Cornelia de Lange综合征有关。2016年Parenti等[14]报道的一例4岁女孩和一例15岁男孩均有典型的Cornelia de Lange综合征临床特征,均存在ANKRD11基因突变。
染色体核型分析及高通量测序常常是伴有特殊面容及其他畸形的发育迟缓患儿病因诊断的常用检查手段,但本研究两例由于存在Cornelia de Lange综合征的特征性表现,首选高通量捕获测序方法确诊,避免了不必要的检查。因此,对于发育迟缓并伴连心眉、弓形眉、多毛、短鼻等特征的患儿要注意Cornelia de Lange综合征可能,可首选高通量捕获测序方法对其7个相关基因进行突变检测,从而在基因水平确定诊断并为遗传咨询提供依据。
| [1] |
Oliver C, Bedeschi MF, Blagowidow N, et al. Cornelia de Lange syndrome:extending the physical and psychological phenotype[J]. Am J Med Genet A, 2010, 152A(5): 1127-1135. DOI:10.1002/ajmg.a.v152a:5 (  0) 0) |
| [2] |
Dorsett D, Krantz ID. On the molecular etiology of Cornelia de Lange syndrome[J]. Ann N Y Acad Sci, 2009, 1151: 22-37. DOI:10.1111/j.1749-6632.2008.03450.x (  0) 0) |
| [3] |
Kline AD, Krantz ID, Sommer A, et al. Cornelia de Lange syndrome:Clinical review, diagnostic and scoring systems, and anticipatory guidance[J]. Am J MedGenet, 2007, 143A(12): 1287-1296. (  0) 0) |
| [4] |
Mannini L, Cucco F, Quarantotti V, et al. Mutation spectrum and genotype-phenotype correlation in Cornelia de Lange syndrome[J]. Hum Mutat, 2013, 34(12): 1589-1596. DOI:10.1002/humu.22430 (  0) 0) |
| [5] |
Krantz ID, McCallum J, DeScipio C, et al. Cornelia de Lange syndrome is caused by mutations in NIPBL, the human homolog of Drosophila melanogaster Nipped-B[J]. Nat Genet, 2004, 36(6): 631-635. DOI:10.1038/ng1364 (  0) 0) |
| [6] |
Tonkin ET, Wang TJ, Lisgo S, et al. NIPBL, encoding a homolog of fungal Scc2-type sister chromatid cohesion proteins and fly Nipped-B, is mutated in Cornelia de Lange syndrome[J]. Nat Genet, 2004, 36(6): 636-641. DOI:10.1038/ng1363 (  0) 0) |
| [7] |
钟秋连. 运用细胞和分子遗传学方法对Cornelia de Lange综合征患者进行遗传诊断[D]. 中南大学, 2011. http://cdmd.cnki.com.cn/Article/CDMD-10533-1011181814.htm
(  0) 0) |
| [8] |
杨保旺, 徐菱阳, 王爱华. 德朗综合征1例报告及基因分析[J]. 临床儿科杂志, 2017, 35(3): 207-209. (  0) 0) |
| [9] |
Musio A, Selicorni A, Focarelli ML, et al. X-linked Cornelia de Lange syndrome owing to SMC1L1 mutations[J]. Nat Genet, 2006, 38(5): 528-530. DOI:10.1038/ng1779 (  0) 0) |
| [10] |
Deardorff MA, Kaur M, Yaeger D, et al. Mutations in cohesin complex members SMC3 and SMC1A cause a mild variant of Cornelia de Lange syndrome with predominant mental retardation[J]. Am J Hum Genet, 2007, 80(3): 485-494. DOI:10.1086/511888 (  0) 0) |
| [11] |
Deardorff MA, Bando M, NakatoR, et al. HDAC8 mutations in Cornelia de Lange syndrome affect the cohesin acetylation cycle[J]. Nature, 2012, 489(7415): 313-317. DOI:10.1038/nature11316 (  0) 0) |
| [12] |
Minor A, Shinawi M, Hogue JS, et al. Two novel RAD21 mutations in patients with mild Cornelia de Lange syndromelike presentation and report of the first familial case[J]. Gene, 2014, 537(2): 279-284. DOI:10.1016/j.gene.2013.12.045 (  0) 0) |
| [13] |
Woods SA, Robinson HB, Kohler LJ, et al. Exome sequencing identifies a novel EP300 frame shift mutation in a patient with features that overlap Cornelia de Lange syndrome[J]. Am J Med Genet A, 2014, 164A(1): 251-258. (  0) 0) |
| [14] |
Parenti I, Gervasini C, Pozojevic J, et al. Broadening of cohesinopathies:exome sequencing identifies mutations in ANKRD11 in two patients with Cornelia de Lange-overlapping phenotype[J]. Clin Genet, 2016, 89(1): 74-81. DOI:10.1111/cge.12564 (  0) 0) |
 2018, Vol. 20
2018, Vol. 20

