儿童恶性肿瘤是指发生在儿童期的恶性肿瘤,其发病率呈逐年上升趋势,成为仅次于意外事故的儿童主要死因之一[1-2],其致病因素主要与遗传有关,肿瘤潜伏期短、生长迅速、侵袭性强。儿童恶性肿瘤可分为血液系统肿瘤和实体瘤两大类,血液系统肿瘤主要为白血病和淋巴瘤,实体瘤主要分为中枢神经系统肿瘤、皮肤与软组织肿瘤、腹部与胃肠道肿瘤、泌尿生殖系统肿瘤以及骨肿瘤[3]。儿童恶性肿瘤的早期防治有助于更快的制定治疗策略,能显著提高疾病的诊治效果。
近年来,随着基因组学和蛋白质组学的不断进步,研究发现肿瘤细胞膜表面独特表达、过度表达或突变的蛋白受体可作为肿瘤细胞的生物标志物,同时也是肿瘤诊治的主要药物靶点,目前大多数药物或治疗方法针对这些靶点设计并抑制其致癌活性[4],其中核酸适体(aptamer)技术在恶性肿瘤的早期诊断和靶向治疗方面展现出巨大潜力和优势[5]。早在1981年,Sidney Altman和Thomas Cech发现RNA不仅是遗传信息的载体,还具有像蛋白质一样的催化活性,并因此获得1989年诺贝尔化学奖,之后一系列的研究证明核酸能进行基因表达调控、催化、配体结合等多种生化反应[6]。在当时为了探究如何才能找到与某个特定靶标相结合的RNA分子,Ellington等[7]和Tuerk等[8]在体外构建了随机核苷酸文库,筛选分离得到了对靶标具有特异性结合能力的RNA序列。Tuerk等[8]将这种筛选方法称为指数富集的配体系统进化技术(systematic evolution of ligands by exponential enrichment, SELEX),此后Ellington等[9]又筛选出了具有结合能力的DNA序列。
通过SELEX技术筛选得到的寡核苷酸序列被称为核酸适体,核酸适体通常是指长度为20~80个碱基的单链DNA或RNA序列,具有高亲和力和特异识别某种靶标分子的能力[10],其识别靶标的原理是通过核苷酸碱基互补配对、π-π堆积、氢键、静电力等多种作用力发生自身适应性折叠后形成特定的三维结构[11],并依靠这种三维结构和靶标之间的分子作用力与靶标高亲和力、高特异性结合[12](图 1),其结合解离常数能达到纳摩尔和皮摩尔水平,与单克隆抗体相当。此外,与抗体相比核酸适体还具有靶标分子范围广泛、热稳定性好、批次差异小、合成价格低、免疫原性低、组织穿透性好、易于化学修饰等多种独特的优势[13],例如针对低免疫原性分子和毒素,抗体较难以制备,而核酸适体却能够在体外顺利完成筛选[14],因此核酸适体作为一种极有前景、有望与抗体媲美的药物,在生物医学领域被广泛研究。
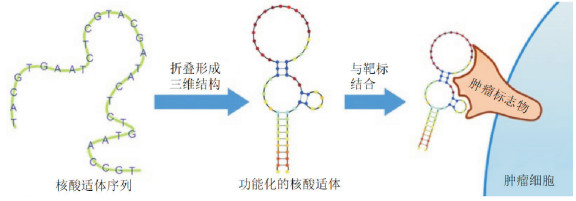
|
图 1 核酸适体与靶标分子结合[13] 核酸适体折叠成特殊三维构象并通过多种分子作用力与靶标分子发生结合。 |
SELEX技术的基本思想来源于自然选择进化理论,其筛选过程一般包括以下几步[15]:首先体外化学合成DNA寡核苷酸文库,每个寡核苷酸序列由中间的随机序列和两边用于结合引物的固定序列组成,随机序列通常含有20~40个碱基,文库中包括1013~1015个不同的DNA序列,足够大的库容量可以产生大量的结构多样性的分子,有利于筛选出和靶标特异性结合的核酸分子。若要筛选RNA适体,DNA文库将会被转录形成RNA。然后,将寡核苷酸文库和靶标分子在一定条件下孵育,将与靶标分子结合的核苷酸序列分离出来,并进行纯化,最后,经过PCR或RT-PCR扩增分离出的结合序列,得到次一级文库,用于下一轮筛选。一般经过8~15轮反复筛选,即可富集到和靶标分子具有高亲和力和特异性结合的核酸适体序列。在肿瘤研究领域,SELEX技术主要分为基于蛋白的protein-SELEX技术和基于细胞的cell-SELEX技术,筛选得到的一系列核酸适体能与蛋白或者肿瘤细胞高亲和力特异性结合,其技术流程见图 2。作为肿瘤诊断和给药的载体,其中某些核酸适体本身还具备肿瘤治疗的能力。
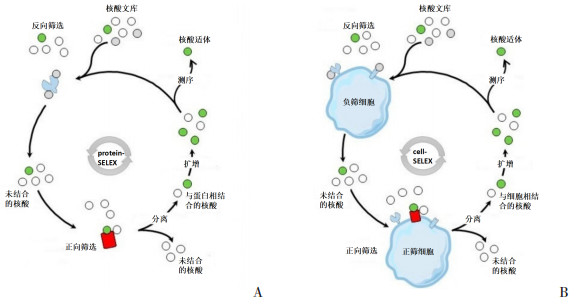
|
图 2 SELEX技术示意图[17] 图A为基于蛋白筛选的protein-SELEX,图B为基于细胞筛选的cell-SELEX,首先将核酸文库与反向筛选的分子或细胞孵育,去除与之结合的核酸,将余下的核酸与正向筛选的靶标分子或靶细胞进行孵育,并扩增与之结合的核酸,经过多轮的筛选后进行测序鉴定并挑选出高亲和力的核酸序列,即为核酸适体。 |
Protein-SELEX是最先运用在肿瘤研究中的核酸适体技术。通常的筛选方法[16]是首先将蛋白基因序列构入质粒载体,通过原核或真核表达并纯化出蛋白分子,利用目标蛋白-标签蛋白的分子进行正向筛选,用只带标签蛋白的分子反向筛选,并且通常会利用标签蛋白(如谷胱甘肽巯基转移酶)固定在固相载体如琼脂糖珠或磁珠上与核酸文库孵育,待核酸分子与蛋白充分结合后,洗掉液相中未结合的核酸分子,将结合的核酸进行PCR扩增得到次一级文库,最后经过测序分析后挑选出富集程度较高的核酸序列即为核酸适体,它能与目标蛋白结合,而不结合标签蛋白。但这种方法不适合于体外表达的蛋白与该蛋白在体内的天然构象不一致的情况,因此又发展出了纯化蛋白与细胞交叉筛选的技术,从而可得到靶向蛋白分子胞外区域的核酸适体[17]。近年来,多种与儿童肿瘤相关的蛋白被筛选出核酸适体,相关文章总结见表 1。
| 表 1 通过protein-SELEX筛选的核酸适体 |
|
|
Cell-SELEX技术以活细胞作为靶标进行核酸适体筛选,由于传统的protein-SELEX筛选一般需要体外表达并纯化出蛋白分子,而蛋白在体内环境或细胞中的构象可能与体外不一致,因此基于细胞筛选的cell-SELEX应运而生[32]。它可以在不清楚细胞表面标志物的条件下进行筛选,筛选过程中核酸适体靶向细胞膜表面的不同分子,并且保持了细胞表面蛋白分子的天然构象,还可以利用筛选到的核酸适体发现新的靶标分子或者肿瘤标志物。Cell-SELEX一般包括两个过程:对靶向细胞的正向筛选和对非靶向细胞的反向筛选,利用这个方法筛选出的核酸适体不仅能够识别不同的肿瘤细胞,还可以用来检测肿瘤细胞与正常细胞或者两类肿瘤细胞在分子水平的差异情况,发现新的肿瘤细胞表面标志物。Cell-SELEX在肿瘤早期诊断、成像及肿瘤治疗方面开辟了新的途径。运用Cell-SELEX技术筛选儿童肿瘤核酸适体的相关文章总结见表 2。
| 表 2 通过cell-SELEX筛选的核酸适体 |
|
|
核酸适体作为与抗体相似的识别元件,具有高亲和力、高特异性、低免疫原性等特点,在生物标志物的发现、肿瘤诊断、生物成像以及药物靶向运输方面等多个生物医学领域拥有良好的应用前景,目前已有8种核酸适体药物进入临床试验或上市[48]。本文总结了核酸适体技术在儿童肿瘤中的应用。
2.1 核酸适体用于儿童恶性肿瘤的诊断肿瘤的早期发现是肿瘤诊治中极其重要的医疗策略,核酸适体与纳米技术联合运用后可被设计成生物传感器,通过生物传感器与靶标分子间的结合作用,将信号输出放大变成可测量的信号,用于分子检测、肿瘤细胞分离富集、疾病早期诊断,具有易于分析研究、记录和成像[49]的优点,使其有望成为肿瘤早期筛查的有效方法。
2.1.1 光学传感器光学核酸适体传感器通常使用灵敏度高的荧光素或者能进行显色的物质作为标记,以光强度作为输出检测的信号。急性淋巴细胞白血病是儿童中最常见的恶性肿瘤疾病,高灵敏度早期检测白血病是临床医生面临的重大挑战。Bahreyni等[50]设计双适体(sgc8c和ATP适体)-功能化的石墨烯氧化物复合物来检测molt-4细胞(人急性淋巴细胞白血病T细胞),该检测依赖于sgc8c作为分子识别探针,在溶酶体中存在大量ATP的环境中,复合物释放带有FAM标记的ATP核酸适体,产生强烈的荧光发射,极大地提高了急性淋巴细胞白血病的诊断率。Tan等[51]基于核酸适体修饰的荧光二氧化硅纳米粒子(FSNPs)开发了一种简单、高灵敏度的检测白血病细胞的方法,在该策略中,胺标记的sgc8核酸适体通过氨基和羧基之间的酰胺偶联与羧基修饰的FSNP缀合,使用流式细胞术和荧光显微镜评估sgc8-FSNPs的敏感性和特异性,结果表明sgc8-FSNPs以高灵敏度和特异性识别白血病细胞。可见核酸适体修饰的FSNPs在检测白血病方面具有极大的潜力。
2.1.2 电化学传感器电化学传感器是目前比较热门的传感器类型,它拥有更高的检测敏感度、可适性,并且能微型化,成本也更加低廉,通过核酸适体与靶标结合后适体结构变化,从而直接或间接引起电极表面的电流发生变化,从而更加灵敏地进行检测。Khoshfetrat等[52]将硫醇化的sgc8c核酸适体固定在金纳米颗粒包被的磁性Fe3O4纳米颗粒Apt-GMNPs上,加入到核酸适体发卡中的溴化乙锭提供了用于量化白血病细胞的输出信号,将白血病细胞结合Apt-GMNPs后,适体发卡结构被破坏,嵌入分子被释放,导致电化学信号减弱。在最佳条件下,适体传感器在10~106 cell/mL的白血病细胞的动态范围内呈线性变化,该方法提供了一个高度灵敏、有选择性、简单且强大的白血病早期检测方法。此外,制作的传感器已用于检测人血浆等复杂病症中的白血病细胞,且无任何严重干扰。Xi等[53]报道了一种基于核酸适体-纳米孔的生物传感器用于检测伯基特淋巴瘤细胞株Romas细胞,识别Romas细胞的核酸适体与Romas细胞结合后,预先互补结合在核酸适体上的cDNA序列脱落下来并触发酶促DNA扩增,最终导致电流释放并被记录,这种方法展现出优异的灵敏度,可以检测少至5个Romas细胞。
2.2 核酸适体治疗儿童恶性肿瘤核酸适体能与各种药物相连接,利用核酸适体的靶向作用进行精准治疗[54],可以减少传统药物在杀伤肿瘤细胞的同时也在破坏正常细胞、组织的副作用。此外,某些核酸适体本身就具有治疗能力[55],可以通过结合靶标分子并封闭其结合位点的作用,抑制靶标分子及下游信号通路的功能,发挥出抗肿瘤效应。
2.2.1 核酸适体对肿瘤的直接治疗作用核酸适体能通过与靶蛋白结合,抑制靶蛋白结合位点的活性从而发挥作用。Amero等[45]通过cell-SELEX筛选到一条靶向神经胶质瘤细胞U87MG的核酸适体GL43.T,经鉴定其靶标分子为酪氨酸激酶受体Eph,Eph受体和其配体ephrin在多种肿瘤中高表达,是药物治疗的有效靶点,GL43.T与Eph受体结合后,阻止其与配体结合,抑制肿瘤细胞的增殖和迁移能力,并促进细胞凋亡。Kishida等[28]筛选了靶向midkine蛋白的RNA核酸适体,适体能阻断midkine蛋白与神经母细胞瘤TNB1细胞结合,进而通过减少Notch2和HES1的表达,抑制了TNB1细胞和小鼠异体移植瘤的增殖能力。Subramanian等[56]发现核仁素蛋白在视网膜母细胞瘤组织和细胞表面高表达,能促进肿瘤发生和血管生成,使用靶向核仁素的核酸适体AS1411处理视网膜母细胞肿瘤株Y79和WERI-Rb1后,能下调miRNA-18a的表达,显著抑制肿瘤细胞的生长,在体内的实验中也表现出相同的效应。
2.2.2 核酸适体连接药物核酸适体的高特异性及高亲和力的性质使其能作为一种出色的化疗给药系统,通常用非共价或者共价结合的方式使核酸适体与药物相连接,其中运用最广泛的是阿霉素(DOX),作为一种经典化疗药物,DOX能插入DNA链中,阻滞DNA的复制与转录。CD123是急性髓细胞白血病的肿瘤标志物,Wu等[18]使用protein-SELEX的方法筛选出能特异性结合CD123的两条核酸适体,通过改造使其携带DOX形成偶联复合物,在小鼠异体移植瘤模型中,该复合物可显著缓解DOX对正常细胞的毒性,抑制肿瘤生长并延长小鼠存活时间,这为CD123高表达的AML肿瘤治疗提供了一条新策略。Zhao等[23]利用CD117的特异核酸适体与化疗药物甲氨蝶呤相偶联,能特异性诱导AML细胞凋亡、阻滞细胞周期,并且在患者样本中能特异性抑制原代AML细胞生长,而对正常骨髓细胞没有毒性作用。
2.2.3 核酸适体与基因技术结合核酸适体还能与siRNA和miRNA,甚至目前热门的CRISPR/Cas9技术结合进行应用。Liang等[47]开发了一种核酸适体与CRISPR/Cas9相结合的技术用于骨肉瘤的治疗,他们将筛选得到的靶向骨肉瘤细胞的核酸适体与聚乙二醇、聚乙烯亚胺和胆固醇共价结合形成脂质聚合物,并将带有剪切血管内皮生长因子A(VEGFA)的CRISPR/Cas9质粒包裹在该聚合物中,降低了骨肉瘤细胞中VEGFA表达和分泌,抑制了小鼠骨肉瘤的血管生成、肺转移以及骨损伤。
3 结语随着核酸适体技术的不断研究,各种快速高效的筛选方法被开发出来,其中基于细胞的cell-SELEX技术可筛选出靶向细胞表面未知蛋白的核酸适体,进而促进新的生物标志物的发现,为疾病特别是肿瘤的早期诊断、成像、治疗以及预后提供了更好的方法和技术手段。另外,核酸适体的各种应用相关技术也越来越丰富,尤其是与药物和纳米材料的联合应用将有可能成为肿瘤治疗方法中的一大利器。不过核酸适体也存在一些缺陷,比如单链DNA及RNA在体内易被降解,半衰期短,靶向特异性不足,缺乏临床研究数据等等。但不可质疑的是,核酸适体的诸多优点以及基于此的诸多应用已经日益受到关注,相信未来的研究将会促进和推动核酸适体技术在临床肿瘤诊断与治疗方面发挥更大作用。
| [1] |
Kaatsch P. Epidemiology of childhood cancer[J]. Cancer Treat Rev, 2010, 36(4): 277-285. DOI:10.1016/j.ctrv.2010.02.003 (  0) 0) |
| [2] |
周艳玲, 安嘉璐, 田玲. 我国儿童恶性肿瘤的流行病学分析[J]. 中国当代儿科杂志, 2015, 17(7): 649-654. DOI:10.7499/j.issn.1008-8830.2015.07.001 (  0) 0) |
| [3] |
赵崇涛, 张立英. 我国儿童肿瘤疾病的发展状况及预防的初 探[J]. 中国民康医学, 2015, 27(12): 81-83. DOI:10.3969/j.issn.1672-0369.2015.12.045 (  0) 0) |
| [4] |
Jacobi N, Seeboeck R, Hofmann E, et al. ErbB family signalling: A paradigm for oncogene addiction and personalized oncology[J]. Cancers (Basel), 2017, 9(4): E33. (  0) 0) |
| [5] |
Xiang D, Shigdar S, Qiao G, et al. Nucleic acid aptamer-guided cancer therapeutics and diagnostics: the next generation of cancer medicine[J]. Theranostics, 2015, 5(1): 23-42. DOI:10.7150/thno.10202 (  0) 0) |
| [6] |
Serganov A, Patel DJ. Ribozymes, riboswitches and beyond: Regulation of gene expression without proteins[J]. Nat Rev Genet, 2007, 8(10): 776-790. DOI:10.1038/nrg2172 (  0) 0) |
| [7] |
Ellington AD, Szostak JW. In vitro selection of RNA molecules that bind specific ligands[J]. Nature, 1990, 346(6287): 818-822. DOI:10.1038/346818a0 (  0) 0) |
| [8] |
Tuerk C, Gold L. Systematic evolution of ligands by exponential enrichment: RNA ligands to bacteriophage T4 DNA polymerase[J]. Science, 1990, 249(4968): 505-510. DOI:10.1126/science.2200121 (  0) 0) |
| [9] |
Ellington AD, Szostak JW. Selection in vitro of singlestranded DNA molecules that fold into specific ligand-binding structures[J]. Nature, 1992, 355(6363): 850-852. DOI:10.1038/355850a0 (  0) 0) |
| [10] |
Li X, Zhao Q, Qiu L. Smart ligand: aptamer-mediated targeted delivery of chemotherapeutic drugs and siRNA for cancer therapy[J]. J Control Release, 2013, 171(2): 152-162. DOI:10.1016/j.jconrel.2013.06.006 (  0) 0) |
| [11] |
de-los-Santos-Álvarez N, Lobo-Castañón MJ, Miranda-Ordieres AJ, et al. Aptamers as recognition elements for label-free analytical devices[J]. Trends Analyt Chem, 2008, 27(5): 437-446. DOI:10.1016/j.trac.2008.03.003 (  0) 0) |
| [12] |
Hermann T, Patel DJ. Adaptive recognition by nucleic acid aptamers[J]. Science, 2000, 287(5454): 820-825. DOI:10.1126/science.287.5454.820 (  0) 0) |
| [13] |
Sun H, Zhu X, Lu PY, et al. Oligonucleotide aptamers: new tools for targeted cancer therapy[J]. Mol Ther Nucleic Acids, 2014, 3: e182. DOI:10.1038/mtna.2014.32 (  0) 0) |
| [14] |
Setlem K, Mondal B, Ramlal S, et al. Immuno affinity SELEX for simple, rapid, and cost-effective aptamer enrichment and identification against aflatoxin B1[J]. Front Microbiol, 2016, 7: 1909. (  0) 0) |
| [15] |
Blind M, Blank M. Aptamer selection technology and recent advances[J]. Mol Ther Nucleic Acids, 2015, 4: e223. DOI:10.1038/mtna.2014.74 (  0) 0) |
| [16] |
Jijakli K, Khraiwesh B, Fu W, et al. The in vitro selection world[J]. Methods, 2016, 106: 3-13. DOI:10.1016/j.ymeth.2016.06.003 (  0) 0) |
| [17] |
Mercier MC, Dontenwill M, Choulier L. Selection of nucleic acid aptamers targeting tumor cell-surface protein biomarkers[J]. Cancers (Basel), 2017, 9(6): E69. (  0) 0) |
| [18] |
Wu H, Wang M, Dai B, et al. Novel CD123-aptamer-originated targeted drug trains for selectively delivering cytotoxic agent to tumor cells in acute myeloid leukemia theranostics[J]. Drug Deliv, 2017, 24(1): 1216-1229. DOI:10.1080/10717544.2017.1367976 (  0) 0) |
| [19] |
Amano R, Takada K, Tanaka Y, et al. Kinetic and thermodynamic analyses of interaction between a high-affinity RNA aptamer and its target protein[J]. Biochemistry, 2016, 55(45): 6221-6229. DOI:10.1021/acs.biochem.6b00748 (  0) 0) |
| [20] |
Fukunaga J, Nomura Y, Tanaka Y, et al. The Runt domain of AML1 (RUNX1) binds a sequence-conserved RNA motif that mimics a DNA element[J]. RNA, 2013, 19(7): 927-936. DOI:10.1261/rna.037879.112 (  0) 0) |
| [21] |
Kotula JW, Sun J, Li M, et al. Targeted disruption of β-arrestin 2-mediated signaling pathways by aptamer chimeras leads to inhibition of leukemic cell growth[J]. PLoS One, 2014, 9(4): e93441. DOI:10.1371/journal.pone.0093441 (  0) 0) |
| [22] |
Faryammanesh R, Lange T, Magbanua E, et al. SDA, a DNA aptamer inhibiting E- and P-selectin mediated adhesion of cancer and leukemia cells, the first and pivotal step in transendothelial migration during metastasis formation[J]. PLoS One, 2014, 9(4): e93173. DOI:10.1371/journal.pone.0093173 (  0) 0) |
| [23] |
Zhao N, Pei SN, Qi J, et al. Oligonucleotide aptamer-drug conjugates for targeted therapy of acute myeloid leukemia[J]. Biomaterials, 2015, 67: 42-51. DOI:10.1016/j.biomaterials.2015.07.025 (  0) 0) |
| [24] |
Zümrüt HE, Batool S, Van N, et al. Structural optimization of an aptamer generated from Ligand-Guided Selection (LIGS) resulted in high affinity variant toward mIgM expressed on Burkitt's lymphoma cell lines[J]. Biochim Biophys Acta, 2017, 1861(7): 1825-1832. DOI:10.1016/j.bbagen.2017.03.020 (  0) 0) |
| [25] |
Parekh P, Kamble S, Zhao N, et al. Immunotherapy of CD30- expressing lymphoma using a highly stable ssDNA aptamer[J]. Biomaterials, 2013, 34(35): 8909-8917. DOI:10.1016/j.biomaterials.2013.07.099 (  0) 0) |
| [26] |
Soldevilla MM, Villanueva H, Bendandi M, et al. 2-fluoro-RNA oligonucleotide CD40 targeted aptamers for the control of B lymphoma and bone-marrow aplasia[J]. Biomaterials, 2015, 67: 274-285. DOI:10.1016/j.biomaterials.2015.07.020 (  0) 0) |
| [27] |
Hicke BJ, Marion C, Chang YF, et al. Tenascin-C aptamers are generated using tumor cells and purified protein[J]. J Biol Chem, 2001, 276(52): 48644-48654. DOI:10.1074/jbc.M104651200 (  0) 0) |
| [28] |
Kishida S, Mu P, Miyakawa S, et al. Midkine promotes neuroblastoma through Notch2 signaling[J]. Cancer Res, 2013, 73(4): 1318-1327. DOI:10.1158/0008-5472.CAN-12-3070 (  0) 0) |
| [29] |
Zhai G, Iskandar M, Barilla K, et al. Characterization of RNA aptamer binding by the Wilms' tumor suppressor protein WT1[J]. Biochemistry, 2001, 40(7): 2032-2040. DOI:10.1021/bi001941r (  0) 0) |
| [30] |
Nalini V, Deepa PR, Raguraman R, et al. Targeting HMGA2 in retinoblastoma cells in vitro using the aptamer strategy[J]. Ocul Oncol Pathol, 2016, 2(4): 262-269. DOI:10.1159/000447300 (  0) 0) |
| [31] |
Camorani S, Esposito CL, Rienzo A, et al. Inhibition of receptor signaling and of glioblastoma-derived tumor growth by a novel PDGFRβ aptamer[J]. Mol Ther, 2014, 22(4): 828-841. DOI:10.1038/mt.2013.300 (  0) 0) |
| [32] |
Sefah K, Shangguan D, Xiong X, et al. Development of DNA aptamers using Cell-SELEX[J]. Nat Protoc, 2010, 5(6): 1169-1185. DOI:10.1038/nprot.2010.66 (  0) 0) |
| [33] |
Shangguan D, Li Y, Tang Z, et al. Aptamers evolved from live cells as effective molecular probes for cancer study[J]. Proc Natl Acad Sci U S A, 2006, 103(32): 11838-11843. DOI:10.1073/pnas.0602615103 (  0) 0) |
| [34] |
Sefah K, Tang ZW, Shangguan DH, et al. Molecular recognition of acute myeloid leukemia using aptamers[J]. Leukemia, 2009, 23(2): 235-244. DOI:10.1038/leu.2008.335 (  0) 0) |
| [35] |
Yang M, Jiang G, Li W, et al. Developing aptamer probes for acute myelogenous leukemia detection and surface protein biomarker discovery[J]. J Hematol Oncol, 2014, 7: 5. DOI:10.1186/1756-8722-7-5 (  0) 0) |
| [36] |
Shangguan D, Cao ZC, Li Y, et al. Aptamers evolved from cultured cancer cells reveal molecular differences of cancer cells in patient samples[J]. Clin Chem, 2007, 53(6): 1153-1155. DOI:10.1373/clinchem.2006.083246 (  0) 0) |
| [37] |
Tang Z, Shangguan D, Wang K, et al. Selection of aptamers for molecular recognition and characterization of cancer cells[J]. Anal Chem, 2007, 79(13): 4900-4907. DOI:10.1021/ac070189y (  0) 0) |
| [38] |
Parekh P, Kamble S, Zhao N, et al. Biostable ssDNA aptamers specific for Hodgkin lymphoma[J]. Sensors (Basel), 2013, 13(11): 14543-14557. DOI:10.3390/s131114543 (  0) 0) |
| [39] |
Wu Q, Wu L, Wang Y, et al. Evolution of DNA aptamers for malignant brain tumor gliosarcoma cell recognition and clinical tissue imaging[J]. Biosens Bioelectron, 2016, 80: 1-8. DOI:10.1016/j.bios.2016.01.031 (  0) 0) |
| [40] |
Tan Y, Shi YS, Wu XD, et al. DNA aptamers that target human glioblastoma multiforme cells overexpressing epidermal growth factor receptor variant Ⅲ in vitro[J]. Acta Pharmacol Sin, 2013, 34(12): 1491-1498. DOI:10.1038/aps.2013.137 (  0) 0) |
| [41] |
Kim Y, Wu Q, Hamerlik P, et al. Aptamer identification of brain tumor-initiating cells[J]. Cancer Res, 2013, 73(15): 4923-4936. DOI:10.1158/0008-5472.CAN-12-4556 (  0) 0) |
| [42] |
Kang D, Wang J, Zhang W, et al. Selection of DNA aptamers against glioblastoma cells with high affinity and specificity[J]. PLoS One, 2012, 7(10): e42731. DOI:10.1371/journal.pone.0042731 (  0) 0) |
| [43] |
Wu X, Liang H, Tan Y, et al. Cell-SELEX aptamer for highly specific radionuclide molecular imaging of glioblastoma in vivo[J]. PLoS One, 2014, 9(6): e90752. (  0) 0) |
| [44] |
Aptekar S, Arora M, Lawrence CL, et al. Selective targeting to glioma with nucleic acid aptamers[J]. PLoS One, 2015, 10(8): e0134957. DOI:10.1371/journal.pone.0134957 (  0) 0) |
| [45] |
Amero P, Esposito CL, Rienzo A, et al. Identification of an interfering ligand aptamer for EphB2/3 receptors[J]. Nucleic Acid Ther, 2016, 26(2): 102-110. DOI:10.1089/nat.2015.0580 (  0) 0) |
| [46] |
Wang H, Liang J, Ma Y, et al. Identification of a novel molecular probe for recognition of human osteosarcoma cell using the cellSELEX method[J]. Int J Clin Exp Med, 2015, 8(10): 18151-18157. (  0) 0) |
| [47] |
Liang C, Li F, Wang L, et al. Tumor cell-targeted delivery of CRISPR/Cas9 by aptamer-functionalized lipopolymer for therapeutic genome editing of VEGFA in osteosarcoma[J]. Biomaterials, 2017, 147: 68-85. DOI:10.1016/j.biomaterials.2017.09.015 (  0) 0) |
| [48] |
Li W, Wang K, Zhao M, et al. Development of aptamer oligonucleotides as anticoagulants and antithrombotics for cardiovascular diseases: current status[J]. Thromb Res, 2014, 134(4): 769-773. DOI:10.1016/j.thromres.2014.05.021 (  0) 0) |
| [49] |
Ozalp VC, Kavruk M, Dilek O, et al. Aptamers: molecular tools for medical diagnosis[J]. Curr Top Med Chem, 2015, 15(12): 1125-1137. DOI:10.2174/1568026615666150413154233 (  0) 0) |
| [50] |
Bahreyni A, Yazdian-Robati R, Ramezani M, et al. Identification and imaging of leukemia cells using dual-aptamer-functionalized graphene oxide complex[J]. J Biomater Appl, 2017, 32(1): 74-81. DOI:10.1177/0885328217712111 (  0) 0) |
| [51] |
Tan J, Yang N, Hu Z, et al. Aptamer-functionalized fluorescent silica nanoparticles for highly sensitive detection of leukemia cells[J]. Nanoscale Res Lett, 2016, 11(1): 298. DOI:10.1186/s11671-016-1512-8 (  0) 0) |
| [52] |
Khoshfetrat SM, Mehrgardi MA. Amplified detection of leukemia cancer cells using an aptamer-conjugated gold-coated magnetic nanoparticles on a nitrogen-doped graphene modified electrode[J]. Bioelectrochemistry, 2017, 114: 24-32. DOI:10.1016/j.bioelechem.2016.12.001 (  0) 0) |
| [53] |
Xi D, Li Z, Liu L, et al. Ultrasensitive detection of cancer cells combining enzymatic signal amplification with an aerolysin nanopore[J]. Anal Chem, 2018, 90(1): 1029-1034. DOI:10.1021/acs.analchem.7b04584 (  0) 0) |
| [54] |
Wu X, Chen J, Wu M, et al. Aptamers: active targeting ligands for cancer diagnosis and therapy[J]. Theranostics, 2015, 5(4): 322-344. DOI:10.7150/thno.10257 (  0) 0) |
| [55] |
Ueki R, Sando S. A DNA aptamer to c-Met inhibits cancer cell migration[J]. Chem Commun (Camb), 2014, 50(86): 13131-13134. DOI:10.1039/C4CC06016D (  0) 0) |
| [56] |
Subramanian N, Srimany A, Kanwar JR, et al. Nucleolinaptamer therapy in retinoblastoma: molecular changes and mass spectrometry-based imaging[J]. Mol Ther Nucleic Acids, 2016, 5(8): e358. (  0) 0) |
 2018, Vol. 20
2018, Vol. 20

