2. 南方医科大学附属深圳市妇幼保健院新生儿科, 广东 深圳 518028
近年,侵袭性真菌感染(invasive fungal infection, IFI)成为新生儿重症监护室(neonatal intensive care unit, NICU)院内感染的重要原因。IFI仍然是早产儿死亡和发病的重要原因[1]。新生儿真菌感染的临床表现缺乏特异性,容易延误诊断及治疗。该病进展迅速,判断真菌感染所需的长时间等待往往导致抗真菌药物开始治疗的延迟[2]。因此,早期准确地识别真菌感染是至关重要的。
目前,真菌学诊断的黄金标准仍然是血液或脑脊液等培养阳性或显微镜下发现真菌[3]。据报道,真菌的血培养阳性率为8%[4]。真菌培养需时较长,培养出真菌的时间为3 d [5],而早产儿感染真菌败血症缺乏典型的症状和体征,病情进展迅速,易错过最佳诊治时间[6]。近年来,分子方法,如PCR检测,逐渐崛起成为有可能代替传统检测真菌感染的方法[7]。分子检测方法具有更高的灵敏度,允许在临床样品中检测少量DNA,更适用于新生儿[8]。
微滴式数字聚合酶链反应技术(droplet digital PCR, ddPCR)是一种新的不依赖于标准曲线对目标序列进行绝对定量的技术[9-10]。ddPCR系统在传统的PCR扩增前将一个大的反应体系进行微滴化处理,即利用油包水技术将其“分割”为数万个纳升级的微滴,每个微滴或不含待检核酸靶分子,或含有至少一个待检核酸靶分子,且每个微滴都是一个独立的PCR反应体系。PCR扩增完成后,利用微滴分析仪逐个对每个微滴进行检测,根据其记录的荧光信号的幅度,有荧光信号的微滴判读为“1”,没有荧光信号的微滴判读为“0”。由于DNA片段随机、独立地分离成液滴,泊松算法可以独立于标准曲线来确定原始样本中的绝对拷贝数[10],从而实现对最初反应体系中核酸靶分子数的绝对定量。近年来,ddPCR技术已在疾病病因、诊断及治疗效果分析等研究中被普遍应用,并且效果显著[11-12]。
本研究的目的是建立一种基于ddPCR技术检测真菌的方法,并探讨此方法在快速、精确检测NICU内IFI中的临床应用价值。
1 资料与方法 1.1 菌种的选择本研究中使用的菌株及来源列于表 1中。为评价ddPCR法的特异性,以3株念珠菌(白假丝酵母菌、近平滑假丝酵母菌、光滑假丝酵母菌)为阳性对照,以10株非真菌菌株为阴性对照,进行ddPCR试验和常规血液培养的平行检测。所有样本行血液培养后,在BACTECTM FX40全自动血培养系统(Becton, Dickinson and Company,美国)中进行鉴定,阳性样本进一步接种于血琼脂平板上,用全自动细菌鉴定药敏分析系统(Biomérieuxc,法国)进行物种鉴定。
| 表 1 本研究中的菌株及来源 |
|
|
ND-1000分光光度计(Nanodrop,美国);C1000 TouchTM PCR仪、QX200TM ddPCR系统(Bio-Rad,美国)。
1.3 基因组DNA的提取(1)真菌基因组DNA提取:将真菌在沙氏液体培养基中增菌培养后,用QIAamp UCP Pathogen Mini Kit及Pathogen Lysis Tubes(QIAGEN,德国)提取真菌基因组DNA,后续操作按照QIAamp UCP Pathogen Mini Kit手册中所建议的真菌DNA的提取方法进行。提取的DNA置-20℃保存备用。
(2)细菌基因组DNA提取:将各种细菌在相应的培养基中增菌培养后,用QIAamp DNA Blood Mini Kit(QIAGEN,德国)提取细菌基因组DNA,后续操作按照QIAamp DNA Blood Mini Kit手册中所建议的细菌DNA的提取方法进行。提取的DNA置-20℃保存备用。
1.4 引物和探针的设计及优化根据GenBank数据库中真菌高度保守的18S rRNA基因序列[13]设计PCR引物和探针序列。正向引物为5'-TTGGTGGAGTGATTTGTCTGCT-3',反向引物为5'-TCTAAGGGCATCACAGACCTG-3'。TaqMan探针选择引物之间的区域,其在5'末端用FAM作为报告染料进行荧光标记,在3'末端用BHQ1作为猝灭剂(FAM- TTAACCTACTAAATAGTGCTGCTAGC-BHQ1)。引物和TaqMan探针来自上海捷瑞生物工程有限公司。采用设计好的引物和探针建立ddPCR方法,包括扩增循环条件的优化及引物和探针浓度的优化。
1.5 ddPCR反应体系和条件ddPCR反应包括4个步骤:配制体系、生成微滴、扩增循环和信号读取[14]。配制体系使用10 μL 2 × ddPCRTM Supermix for Probes,正、反向引物各2 μL,探针0.5 μL,DNA模板1 μL,用超纯水补足体积至20 μL。将配制好的20 μL PCR反应液,转移至微滴发生卡孔中,再加入70 μL微滴发生油至油孔中,利用QX200TMddPCR系统的微滴生成器制备反应微滴。将每个样品的微滴分别转移至96孔PCR反应板对应的反应孔中,用铝膜热封(180℃ 5 s)后,于普通PCR仪上进行扩增,本实验的PCR扩增是在C1000 TouchTM PCR仪上完成的,其中退火温度是经过温度梯度优化后的温度。ddPCR扩增使用3步法,反应条件如下:95℃预变性10 min;94℃变性30 s,59.2℃退火60 s,共40个循环;扩增结束后进行98℃热变性10 min。每个模板重复3个平行检测。将PCR扩增后的96孔板放入QX200TM ddPCR系统的微滴分析器中,检测FAM的荧光信号。仪器会自动分析样品的各微滴荧光信号,然后由QuantaSoft1.7.4软件完成对数据的自动处理。
1.6 临床试验为了评价新建立的ddPCR技术检测新生儿IFI的诊断价值,本研究选取2016年5月至2018年5月于深圳市妇幼保健院NICU住院的有深部真菌感染高危因素和/或临床表现[15-16]的新生儿83例为研究对象,采集所有患儿血液或脑脊液等无菌体液进行真菌培养及ddPCR检测。参考欧洲癌症研究治疗组织及真菌研究组(EORTC/MSG)的诊断标准[16]和中国IFI工作组制定的临床诊断侵袭性真菌病的诊断标准[17],分为确诊、临床诊断、拟诊及非IFI。将确诊及临床诊断病例作为IFI组(n=22),拟诊及非IFI病例作为非IFI组(n=61)。比较ddPCR检测结果与IFI诊断标准结果的一致性。
1.7 统计学分析采用SPSS 22.0统计软件对数据进行统计学分析,通过Kappa一致性检验评价ddPCR法检测与IFI组的诊断一致性,计算灵敏度、特异度、阳性预测值、阴性预测值和约登指数评价ddPCR法在所有分析样本中的诊断准确性,P<0.05为差异有统计学意义。
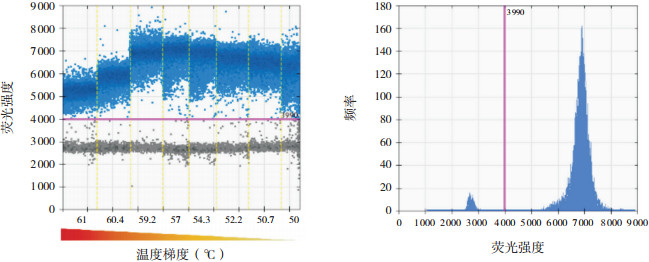
2 结果 2.1 最佳温度的摸索以白假丝酵母菌为例,用优化的引物、探针及目标序列浓度,通过梯度PCR后,摸索出在59.2℃处阳性微滴与阴性微滴区分得最开,且之间空白处微滴数目最少(图 1左),峰图显示59.2℃时阳性峰和阴性峰显著分开,中间没有任何干扰(图 1右),表明经过优化确定的ddPCR探针浓度和扩增体系适合对真菌进行定量分析。
|
图 1 微滴式数字PCR法梯度温度扩增白假丝酵母菌基因组DNA的一维散点图(左)及最佳温度(59.2℃)下的峰图(右) 图左中蓝色点代表阳性微滴,灰色点代表阴性微滴。不同的温度下,阳性微滴及阴性微滴区分的情况不同。图右中左侧为阴性峰,右侧为阳性峰,两者显著分开,中间无干扰。图中红线为阳性微滴及阴性微滴区分得最开时,即59.2℃下的阳性微滴振幅低值及阴性微滴振幅高值的中位数。 |
紫外分光光度法测定提取的白假丝酵母菌基因组DNA核酸浓度,然后进行10倍梯度稀释,ddPCR检测到经106倍稀释的白假丝酵母菌基因组DNA,检出限为3.2 copies/μL,见图 2。

|
图 2 微滴式数字PCR法梯度稀释扩增白假丝酵母菌基因组DNA的一维散点图 蓝色点代表阳性微滴,灰色点代表阴性微滴。进行10倍梯度稀释时,阳性微滴成梯度减少,到稀释倍数为107时,检测不到阳性微滴。 |
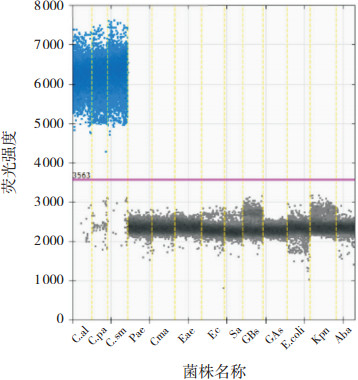
为了证实引物的特异性,使用设计好的引物及探针对3种本研究中使用的真菌菌株分别进行RT-PCR反应,反应产物均进行测序。测序所得结果显示与GenBank数据库中真菌18S rRNA基因的序列100%同源。此外,将3株真菌菌株及10种细菌菌株(表 1)进行增菌培养后,提取相应的基因组DNA,采用已优化的ddPCR检测方法对上述所有DNA同时进行检测。3株真菌菌株基因组DNA的ddPCR检测结果可见大量阳性微滴,而10株细菌菌株基因组DNA的ddPCR检测结果均未见阳性微滴,见图 3。
|
图 3 微滴式数字PCR法梯度扩增3株真菌基因组DNA及10株细菌基因组DNA的一维散点图 蓝色点代表阳性微滴,灰色点代表阴性微滴。3株真菌基因组DNA可见大量阳性微滴。10株细菌基因组DNA均未见阳性微滴。C.al:白假丝酵母;C.pa:近平滑假丝酵母;C.sm:光滑假丝酵母菌;Pae:铜绿假单胞菌;Cma:麦氏棒杆菌;Eae:产气杆菌;Ec:阴沟肠杆菌;Sa:金黄色葡萄球菌;GBs:B族链球菌;GAs:A族链球菌;E.coli:大肠杆菌;Kpn:肺炎克雷伯菌;Aba:鲍曼不动杆菌。 |
按已优化的ddPCR检测条件进行重复性检测,批内及批间重复性实验均以白假丝酵母菌基因组DNA为模板,使用新建立的体系进行3个浓度的ddPCR检测。批间重复性实验是对同一样本在反应体系相同的条件下进行连续3 d重复测定,结果显示相同浓度样本的反应之间阳性微滴数接近,且变异系数分别为1.00%、2.73%、2.42%(表 2);批内重复性实验是对同一样本在同一反应体系中同时进行3次重复测定,结果显示相同浓度模板的反应之间阳性微滴数接近,变异系数分别为2.82%、2.24%、2.71%(表 3),说明建立的ddPCR检测体系重复性好。
| 表 2 不同浓度的白假丝酵母菌基因组DNA批间重复性检测结果 |
|
|
| 表 3 不同浓度的白假丝酵母菌基因组DNA的批内重复性检测结果 |
|
|
以诊断标准[16-17]为依据,在本研究中,ddPCR检测IFI组的阳性例数为19例(n=22),检测非IFI组的阳性例数为2例(n=61)。ddPCR技术检测新生儿IFI阳性的敏感度为86%(19/22),特异度为97%(59/61),阳性预测值为91%(19/21),阴性预测值为95%(59/62),约登指数为0.830。ddPCR法检测与IFI组的诊断一致性好(Kappa=0.843,P<0.001)。
3 讨论在NICU的极低出生体重儿中,念珠菌败血症是晚发感染的第4位病因[18]。近期,有报道显示IFI病例病死率为30.8%~36.2%[19],新生儿真菌感染病情进展迅速,判断真菌感染所需的长时间等待往往导致抗真菌药物开始治疗的延迟[2]。因此,早期准确地识别真菌感染是至关重要的。
目前,真菌学诊断的黄金标准仍然是血液或脑脊液等培养阳性或显微镜下发现真菌[3]。真菌培养虽可明确真菌感染病原菌并进行体外药敏试验,但血培养阳性率低。1, 3-β-D-葡聚糖是一种真菌细胞壁成分,已被用于辅助诊断各种真菌感染[14]。有些因素如纱布、棉球、一些抗生素类药物、静脉注射白蛋白或丙种球蛋白、血浆、组织凝血活酶等有时可造成1, 3-β-D-葡聚糖检测(G试验)呈假阳性结果[20]。半乳甘露聚糖抗原是曲霉菌细胞壁的主要成分,在曲霉菌生长过程中由菌丝释放出来,半乳糖甘露醇聚糖抗原检测(GM试验)主要针对曲霉菌感染,但GM试验结果亦受诸多因素影响,如哌拉西林钠/三唑巴坦、大剂量激素的使用均可致假阳性的出现。基于几项研究,分子方法如PCR检测,已经取得了令人鼓舞的结果,并且灵敏度可等于或高于血培养[8]。此外,实时荧光定量PCR技术有利于量化临床标本中的真菌负担[21]。然而,这项技术存在固有的局限性,如需制备标准品,通过标准曲线定量等,可能无法精确和准确地量化病原体载量[10]。
ddPCR技术是检测和定量细菌、真菌、病毒等遗传物质的一种很有前途的新技术。本研究选择真菌高度保守的18S rRNA基因序列,通过引物设计,体系优化,建立了ddPCR检测真菌体系。3株念珠菌菌株基因组DNA的ddPCR检测见大量阳性微滴,而10株细菌基因组DNA均未见阳性微滴,证明该体系能够检测真菌DNA而不与其他细菌病原体有交叉杂交,特异性良好。通过对白假丝酵母菌基因组DNA进行梯度稀释,检测到经106倍稀释的白假丝酵母菌基因组DNA,检出限为3.2 copies/μL,对比报道的RT-qPCR的检出极限69 copies/μL[2],有更好灵敏度。按已优化的ddPCR检测条件进行重复性检测结果显示,批内及批间重复性实验均以白假丝酵母菌基因组DNA为模板,进行三个浓度的ddPCR检测。批间重复性试验结果显示相同浓度模板的反应之间阳性微滴数接近,且变异系数分别为1.00%、2.73%、2.42%,批内重复性试验结果显示相同浓度模板的反应之间阳性微滴数接近,变异系数分别为2.82%、2.24%、2.71%。表明建立的方法具有较好的重复性和稳定性。
这项研究的目的是建立及验证一种新的泛真菌ddPCR检测方法,并评估ddPCR检测在常规培养阴性的情况下,诊断新生儿侵袭性真菌病的能力。为了评价新建立的ddPCR技术检测新生儿IFI的临床适用性,本研究对深圳一医院有新生儿真菌感染高危因素和/或临床表现的83例患儿血样同时进行了常规血培养分析和ddPCR检测。发现本研究建立的ddPCR方法可能比其他诊断方法具有重要优势。首先,常规培养及鉴定技术检测至少需要3 d,而ddPCR检测由于无需标准品及标准曲线的建立,非常灵敏、简便,真菌目的基因从抽取血液到出结果仅仅需要6 h。这一优势在早期确定感染中有很大的价值。早期检测感染并区分假丝酵母菌的能力,对于是否引入抗真菌治疗非常重要。早期明确是否存在真菌感染可减少预防性和经验性滥用抗真菌药物,从而改善感染患者的预后。此外,常规的血培养最少需要血量2 mL,而新建立的ddPCR技术仅需要0.4 mL,这对新生儿,特别是早产儿是有利的。此外,经过Kappa一致性检验结果显示,Kappa=0.843,表示ddPCR法检测结果与确诊/临床诊断IFI的吻合度较高,表明两者一致性较好。本研究发现,ddPCR法诊断病人具有较高的灵敏度、特异度、阳性预测值、阴性预测值,约登指数为0.830,该方法可以较好的用于诊断。另外,ddPCR在排除感染可能性方面有价值(阴性预测值为95%),这可能允许撤回不必要的抗真菌治疗,从而减少药物潜在毒性、耐药性和成本。近期有研究发现,在培养阴性的情况下,PCR阳性结果应视为可能感染的迹象[22]。
由于本实验样本数较少,需要更大的样本量进一步验证该技术的临床诊断价值。此次设计的引物为泛真菌的引物,未能通过ddPCR确定具体菌属及耐药基因等情况,而血培养鉴定及进一步的药敏测试则可做到。现在已经有研究使用PCR技术,通过对引物的设计,对耐药基因及菌属行进一步确定[13]。本研究中出现2例均为临床诊断IFI而ddPCR检测阴性,考虑ddPCR反应仍有可能受到抑制剂的影响,导致结果阴性;而非IFI组存在2例ddPCR检测阳性,考虑存在交叉污染的可能。这项技术应用在病原体的诊断上也存在问题,只能检测无菌样本,如血液、脑脊液、胸腔积液等,并且容易交叉污染,因此需要严格的实验条件及熟练的人员进行操作。此外,ddPCR技术存在PCR技术固有的缺点,如受抑制剂的影响,故每建立一个体系时需尽可能优化。ddPCR技术是依赖微滴数区分阳性和阴性,应进一步进行大样本数据的收集,采用统计方法来定义阳性和阴性的截断值,以加强ddPCR的效用,进一步优化这一新技术将改进对极低滴度的病原体基因的检测。
综上,本研究选择真菌通用18S RNA为目标序列,通过引物设计、体系优化,建立了ddPCR检测真菌体系,并验证了特异性、灵敏度、重复性。本研究发现,确诊/临床诊断IFI与ddPCR检测阳性一致性好,且该技术有较高的灵敏度及特异性。对于疾病诊断及排除的意义,尚需更大量的临床样本进一步验证。ddPCR技术虽然有PCR固有的局限性,如容易污染及受抑制剂影响,并且现阶段试剂及耗材昂贵,但仍是研究新生儿感染筛查甚至诊断很有前途的工具。相对于3 d才能出结果的传统血培养,ddPCR技术需时短、需血量少等优势,非常适合新生儿特别是早产儿早期感染的诊断需求。ddPCR技术不仅检测速度快、灵敏度高,而且还可以对真菌DNA进行准确的量化检测。可在一定程度上提高IFI的早期诊断率,为预防性使用抗真菌药物及早期治疗IFI提供更加充分的依据。
| [1] |
Kelly MS, Benjamin DJ, Smith PB, et al. The epidemiology and diagnosis of invasive candidiasis among premature infants[J]. Clin Perinatol, 2015, 42(1): 105-117. DOI:10.1016/j.clp.2014.10.008 (  0) 0) |
| [2] |
Jiang X, Dong D, Bian L, et al. Rapid detection of Candida albicans by polymerase spiral reaction assay in clinical blood samples[J]. Front Microbiol, 2016, 7: 916. (  0) 0) |
| [3] |
Roilides E. Invasive Candidiasis in neonates and children[J]. Early Human Development, 2011, 87: S75-S76. DOI:10.1016/j.earlhumdev.2011.01.017 (  0) 0) |
| [4] |
Gupta P, Ahmad A, Khare V, et al. Comparative evaluation of pan-fungal real-time PCR, galactomannan and (1-3)-beta-Dglucan assay for invasive fungal infection in paediatric cancer patients[J]. Mycoses, 2017, 60(4): 234-240. DOI:10.1111/myc.2017.60.issue-4 (  0) 0) |
| [5] |
Cahan H, Deville JG. Outcomes of neonatal Candidiasis:the impact of delayed initiation of antifungal therapy[J]. Int J Pediatr, 2011, 2011: 813871. (  0) 0) |
| [6] |
唐仕芳, 陈龙, 赵锦宁, 等. 新生儿重症监护病房侵袭性真菌感染易感因素及早期诊断探讨[J]. 临床儿科杂志, 2011, 29(9): 810-812. DOI:10.3969/j.issn.1000-3606.2011.09.003 (  0) 0) |
| [7] |
Valero C, de la Cruz-Villar L, Zaragoza O, et al. New panfungal real-time PCR assay for diagnosis of invasive fungal infections[J]. J Clin Microbiol, 2016, 54(12): 2910-2918. DOI:10.1128/JCM.01580-16 (  0) 0) |
| [8] |
Greenberg RG, Benjamin DJ. Neonatal Candidiasis:diagnosis, prevention, and treatment[J]. J Infect, 2014, 69(Suppl 1): S19-S22. (  0) 0) |
| [9] |
Pinheiro LB, Coleman VA, Hindson CM, et al. Evaluation of a droplet digital polymerase chain reaction format for DNA copy number quantification[J]. Anal Chem, 2012, 84(2): 1003-1011. DOI:10.1021/ac202578x (  0) 0) |
| [10] |
Hindson BJ, Ness KD, Masquelier DA, et al. High-throughput droplet digital PCR system for absolute quantitation of DNA copy number[J]. Anal Chem, 2011, 83(22): 8604-8610. DOI:10.1021/ac202028g (  0) 0) |
| [11] |
Persaud D, Gay H, Ziemniak C, et al. Absence of detectable HIV-1 viremia after treatment cessation in an infant[J]. N Engl J Med, 2013, 369(19): 1828-1835. DOI:10.1056/NEJMoa1302976 (  0) 0) |
| [12] |
Sedlak RH, Cook L, Huang ML, et al. Identification of chromosomally integrated human herpesvirus 6 by droplet digital PCR[J]. Clin Chem, 2014, 60(5): 765-772. DOI:10.1373/clinchem.2013.217240 (  0) 0) |
| [13] |
Ramos JT, Villar S, Bouza E, et al. Performance of a quantitative PCR-based assay and beta-d-glucan detection for diagnosis of invasive Candidiasis in very-low-birth-weight preterm neonatal patients (CANDINEO Study)[J]. J Clin Microbiol, 2017, 55(9): 2752-2764. DOI:10.1128/JCM.00496-17 (  0) 0) |
| [14] |
Tang XL, Hua Y, Guan Q, et al. Improved detection of deeply invasive candidiasis with DNA aptamers specific binding to (1→3)-beta-D-glucans from Candida albicans[J]. Eur J Clin Microbiol Infect Dis, 2016, 35(4): 587-595. DOI:10.1007/s10096-015-2574-8 (  0) 0) |
| [15] |
陈超. 新生儿真菌感染的诊治[J]. 中国实用儿科杂志, 2011, 26(1): 3-6. (  0) 0) |
| [16] |
De Pauw B, Walsh TJ, Donnelly JP, et al. Revised definitions of invasive fungal disease from the European Organization for Research and Treatment of Cancer/Invasive Fungal Infections Cooperative Group and the National Institute of Allergy and Infectious Diseases Mycoses Study Group (EORTC/MSG) Consensus Group[J]. Clin Infect Dis, 2008, 46(12): 1813-1821. DOI:10.1086/590167 (  0) 0) |
| [17] |
中国侵袭性真菌感染工作组. 血液病/恶性肿瘤患者侵袭性真菌病的诊断标准与治疗原则(第五次修订版)[J]. 中华内科杂志, 2017, 56(6): 453-459. DOI:10.3760/cma.j.issn.0578-1426.2017.06.015 (  0) 0) |
| [18] |
Reissa E, Lasker BA, Iqbal NJ, et al. Molecular epidemiology of Candida parapsilosis sepsis from outbreak investigations in neonatal intensive care units[J]. Infect Genet Evol, 2008, 8(2): 103-109. DOI:10.1016/j.meegid.2007.10.007 (  0) 0) |
| [19] |
Ting J, Roberts A, Synnes A, et al. Invasive fungal infections in neonates in Canada:epidemiology and outcomes[J]. Pediatr Infect Dis J, 2018, 37(11): 1154-1159. DOI:10.1097/INF.0000000000001968 (  0) 0) |
| [20] |
Mennink-Kersten MA, Verweij PE. Non-culture-based diagnostics for opportunistic fungi[J]. Infect Dis Clin North Am, 2006, 20(3): 711-727. DOI:10.1016/j.idc.2006.06.009 (  0) 0) |
| [21] |
Khot PD, Fredricks DN. PCR-based diagnosis of human fungal infections[J]. Expert Rev Anti Infect Ther, 2009, 7(10): 1201-1221. DOI:10.1586/eri.09.104 (  0) 0) |
| [22] |
Halliday CL, Kidd SE, Sorrell TC, et al. Molecular diagnostic methods for invasive fungal disease:the horizon draws nearer?[J]. Pathology, 2015, 47(3): 257-269. DOI:10.1097/PAT.0000000000000234 (  0) 0) |
 2019, Vol. 21
2019, Vol. 21


