runt相关转录因子3(runt-related transcription factor 3, RUNX3)作为一种重要的抑癌基因,近年来显示与呼吸道疾病密切相关[1]。有研究发现卵清白蛋白(ovalbumin, OVA)激发的小鼠肺组织中核RUNX3蛋白减少,当使用抗炎剂治疗后,RUNX3蛋白在细胞核中分布增加,小鼠过敏性气道炎症和气道高反应性减弱[2]。另有学者采集哮喘患者和健康对照组外周血,分离培养CD4+ T细胞,并检测其中microRNA-145和RUNX3 mRNA水平,发现RUNX3 mRNA表达与microRNA-145呈负相关,并影响相关的细胞因子表达[3]。RUNX3参与一系列信号转导,在T细胞发育、分化过程中起到很重要的作用。RUNX3协同T-bet等调节因子以正反馈机制促进Th1分化,诱导干扰素γ(IFN-γ)的分泌[4]。另外,RUNX3还能协同T-bet及活化T细胞核因子抑制IL-4的表达,从而对Th2的分化产生影响[5]。研究证实,激活RUNX3可逆转哮喘患者Th1/Th2失衡,提示RUNX3可作为哮喘诊断的生物标志物或治疗的靶标[6]。
毛细支气管炎作为生命早期最常见的一种下呼吸道感染,与哮喘在很大程度上具有相似的发病机制[7-8],是儿童哮喘的重要危险因素之一。但是毛细支气管炎发展为哮喘只是一部分患儿,大多数患儿经急性期治疗后可痊愈,如何甄别毛细支气管炎中的哮喘高危儿目前尚缺乏有效手段。
本研究将检测外周血单个核细胞(peripheral blood mononuclear cells, PBMCs)中RUNX3基因在毛细支气管炎患儿中的表达情况,分析该基因mRNA水平与Th1细胞代表因子IFN-γ的相关性,以及和Th2细胞代表因子IL-4的相关性,以期明确PBMCs中RUNX3基因表达在毛细支气管炎中的临床意义,评估其在毛细支气管炎发展为哮喘中的指导价值,为早期甄别哮喘高危儿,实现哮喘的二级预防提供依据。
1 资料与方法 1.1 研究对象选取2017年10月至2018年3月于江苏省连云港市妇幼保健院就诊的毛细支气管炎患儿54例为毛细支气管炎组,其中有特应性体质患儿28例为特应性毛细支气管炎组,非特应性体质患儿26例为非特应性毛细支气管炎组;同时选取于我院行健康体检的健康婴幼儿48例为健康对照组,其中有特应性体质婴幼儿24例为特应性健康对照组,非特应性体质婴幼儿24例为非特应性健康对照组。特应性体质为婴幼儿时期经医生诊断有特应性皮炎史,诊断标准参考文献[9]。本研究已获得所有受试者家属或监护人同意并签署知情同意书。
纳入标准:病例组:(1)年龄3~24个月;(2)均符合《诸福棠实用儿科学》第8版的毛细支气管炎的诊断标准[10];(3)均处于疾病急性期的轻中度患儿,咳嗽气喘明显,肺部可闻及哮鸣音及中细湿罗音;(4)入组前未使用全身性糖皮质激素;(5)无继发细菌感染。健康对照组:(1)年龄3~24个月;(2)既往体健,无毛细支气管炎等喘息性疾病史。
排除标准:病例组:(1)排除剧烈喘憋、低氧血症、生命体征不稳定的重症毛细支气管炎患儿;(2)排除先天性气道发育异常、心血管疾病、气管异物、胃食管反流病等可表现为喘憋的疾病;(3)排除合并内分泌、心、肝、肾等严重疾病;(4)排除免疫缺陷、有机械通气史及免疫抑制剂使用史。
1.2 RNA提取和cDNA合成采集各组婴儿外周血3 mL于EDTA抗凝管中,使用淋巴细胞分离液(GE,美国)分离外周血淋巴细胞。按照TRIzol试剂(Invitrogen, 美国)说明书步骤提取外周血淋巴细胞中总RNA。将提取的RNA纯化后,使用RNase-free DNase处理以消除gDNA污染。RNA浓度使用NanoDrop 2000分光光度计(Thermo Scientific,美国)测定。按照PrimeScript RT试剂试剂盒(大连宝生物工程有限公司)说明书对提取的RNA进行逆转录后得到cDNA,然后用DEPC水将cDNA产物稀释到最终浓度,放置于-80℃冰箱中备用。
1.3 荧光定量PCR检测RUNX3 mRNA水平采用SYBR Premix Ex TaqTM Ⅱ试剂盒(大连宝生物工程有限公司)进行荧光定量PCR,分析4组外周血淋巴细胞中RUNX3 mRNA水平,OAZ1基因作为内参基因。引物由Primer Premier 5.0软件根据人类基因组序列设计,并由深圳华大基因股份有限公司合成。RUNX3:上游引物:5′-GTCGCCTTCAAGGTGGTGGC-3′,下游引物:5′-TAGGTCGCCACTTGGGTGGG-3′,片段长度227 bp;OAZ1:上游引物:5′-AGCAAGGACAGCTTTGCAGTTCTC-3′,下游引物:5′-GATGCCCCGGTCTCACAATC-3′,片段长度154 bp。荧光定量PCR反应体系:SYBR® Premix Ex TaqTM Ⅱ(2×)5 μL,ROX Reference Dye Ⅱ(50×)0.2 μL,上、下游引物各1 μL,模板DNA 1.5 μL,ddH2O补足至10 μL。反应条件:95℃ 30 s;95℃ 5 s,60℃ 31 s,40个循环;72℃ 10 s。根据测得的Ct值,计算RUNX3 mRNA的相对表达量,结果以2-△△Ct表示。
1.4 血清IL-4和IFN-γ浓度测定采集各组婴儿外周血2 mL于无抗凝剂的血清管中,4℃冰箱中倾斜放置1 h后,3 000 r/min离心得到血清,冻存于-80℃冰箱中备用。使用酶联免疫吸附法(ELISA)检测血清中IL-4、IFN-γ浓度,ELISA试剂盒购自深圳欣博盛生物公司,实验按照试剂盒说明书操作进行。
1.5 统计学分析采用SPSS 22.0统计软件对数据进行统计学分析。计量资料使用均数±标准差(x±s)表示,两组间比较采用两样本t检验;多组间比较使用单因素方差分析,组间两两比较采用SNK-q检验。计数资料采用例数表示,多组间比较采用卡方检验;相关性分析使用Pearson相关分析。P < 0.05为差异有统计学意义。
2 结果 2.1 临床资料分析4组婴儿分别在性别、年龄、出生体重、生产方式、喂养方式、早产、被动吸烟等方面比较差异均无统计学意义(P > 0.05);毛细支气管炎两亚组婴儿在咳嗽、发热和病程方面比较差异也无统计学意义(P > 0.05)。各组之间具有可比性,见表 1~2。
| 表 1 各亚组一般临床资料分析 |
|
|
| 表 2 毛细支气管炎两亚组患儿病情资料比较 (例) |
|
|
毛细支气管炎组患儿外周血淋巴细胞中RUNX3 mRNA水平(0.25±0.10)明显低于健康对照组(0.33±0.14,t=-3.424,P < 0.01)。毛细支气管炎两亚组及健康对照两亚组患儿外周血淋巴细胞中RUNX3 mRNA水平比较差异有统计学意义(F=6.089,P < 0.01),其中特应性毛细支气管炎组患儿外周血淋巴细胞中RUNX3 mRNA水平(0.22±0.10)显著低于非特应性毛细支气管炎组(0.28±0.10)、特应性健康对照组(0.35±0.14)和非特应性健康对照组(0.31±0.14)(P < 0.05)。
2.3 血清IL-4和IFN-γ浓度毛细支气管炎组血清IL-4浓度显著高于健康对照组(P < 0.05);特应性毛细支气管炎组、非特应性毛细支气管炎组和特应性健康对照组血清IL-4浓度显著高于非特应性健康对照组(P < 0.05)。毛细支气管炎组血清IFN-γ浓度显著低于健康对照组(P < 0.05);特应性毛细支气管炎组、非特应性毛细支气管炎组和特应性健康对照组血清IFN-γ浓度显著低于非特应性健康对照组,且特应性毛细支气管炎组血清IFN-γ浓度显著低于非特应性毛细支气管炎组和特应性健康对照组(P < 0.05)。见表 3~4。
| 表 3 毛细支气管炎组与健康对照组血清IL-4及IFN-γ浓度比较 (x±s,ng/mL) |
|
|
| 表 4 各亚组血清IL-4及IFN-γ浓度比较 (x±s,ng/mL) |
|
|
各亚组RUNX3 mRNA水平与血清IFN-γ浓度均呈正相关(P < 0.05);除非特应性健康对照组外,其余3个亚组RUNX3 mRNA水平与血清IL-4浓度均呈负相关(P < 0.05)。见图 1~2。
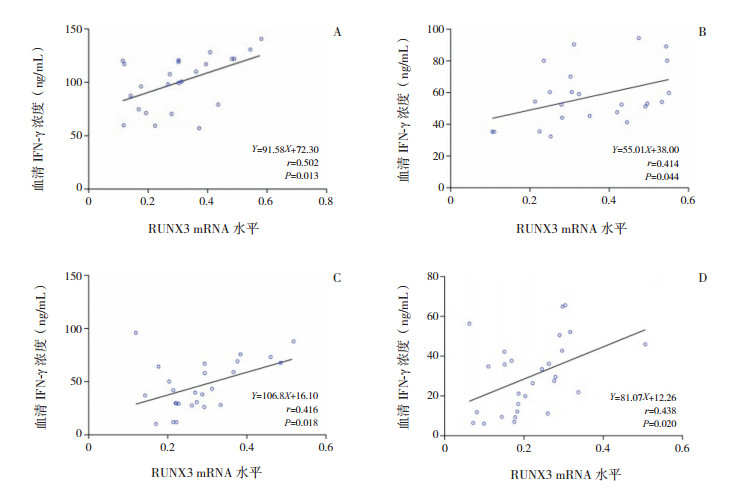
|
图 1 各亚组RUNX3 mRNA水平与血清IFN-γ相关分析图 A为非特应性健康对照组,B为特应性健康对照组,C为非特应性毛细支气管炎组,D为特应性毛细支气管炎组。 |
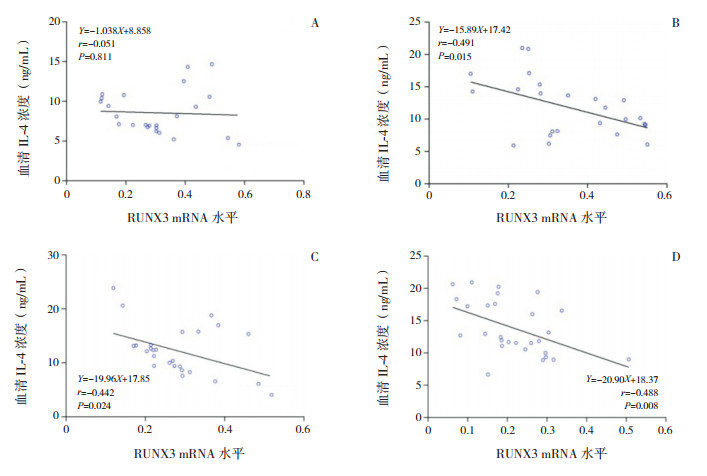
|
图 2 各亚组RUNX3 mRNA水平与血清IL-4相关分析图 A为非特应性健康对照组,B为特应性健康对照组,C为非特应性毛细支气管炎组,D为特应性毛细支气管炎组。 |
RUNX3作为免疫系统重要的调节因子,被认为与各种人类疾病的发生和发展有关,包括胃癌、结肠癌和其他疾病[11-13]。RUNX3缺陷小鼠会自发产生免疫学异常,如气道超敏性和结肠炎[14]。研究显示,RUNX3可调节树突状细胞上趋化因子受体CCR7,在RUNX3基因敲除小鼠中,CCR7表达增加,使树突状细胞向肺部引流淋巴结迁徙,导致哮喘症状[15]。另有研究显示,RUNX3基因敲除小鼠中出现嗜酸性粒细胞增多,导致肺部炎症反应和气道重构现象[16]。尽管目前对RUNX3的调节机制仍未完全清楚,但RUNX3在呼吸道疾病的发展中起着不可或缺的作用,RUNX3在呼吸道疾病发病中所起的调节机制仍有待进一步的研究和证实。
有研究者对年龄 < 1岁因呼吸道合胞病毒感染且需住院治疗的毛细支气管炎患儿进行了长达7年的前瞻性研究显示,其日后反复喘息及哮喘的发生率分别为68%和30%,均明显高于对照组[17]。我国也有研究显示,婴儿首次患毛细支气管炎后,其中约有40%~50%患儿将在其后不久反复发生喘息,最终演变为哮喘[18],可见毛细支气管炎是儿童哮喘的重要危险因素之一。本研究表明,毛细支气管炎患儿外周血淋巴细胞中RUNX3 mRNA水平明显低于健康婴儿,尤其是合并特应性体质的毛细支气管炎组RUNX3 mRNA的水平显著低于非特应性健康对照组。表明RUNX3 mRNA水平在毛细支气管炎患儿尤其是合并特应性皮炎患儿中明显减少。婴儿特应性皮炎史是特应性体质的表现,合并婴儿特应性皮炎的毛细支气管炎患儿是哮喘高危儿。本研究中,RUNX3 mRNA水平在哮喘高危儿中明显降低,提示RUNX3在毛细支气管炎发展为哮喘的机制中具有重要作用。
在T细胞分化过程中,CD4+ T细胞根据其产生的细胞因子不同分为Th1和Th2细胞。Th1细胞主要产生IFN-γ,可活化吞噬细胞,增强其抗感染能力,介导细胞免疫反应;而Th2细胞主要产生IL-4、IL-10等细胞因子,可促进B细胞分化,诱导B细胞合成IgE,介导体液免疫反应。二者以相互拮抗和自身促进的方式形成复杂有序的细胞因子网络,调节机体的免疫应答,其比例和功能失衡是哮喘发病机制中的重要环节[19]。有研究表明,毛细支气管炎与儿童哮喘间存在相似的细胞与体液免疫功能紊乱,如Th1/Th2细胞功能和比例失衡,Th2细胞因子呈强势状态,患儿呼吸道T淋巴细胞浸润,肺泡腔内嗜酸性细胞聚集、脱颗粒并释放大量炎性递质等[20-21]。本研究显示,毛细支气管炎组血清IL-4浓度明显高于健康对照组,尤其是合并特应性体质的毛细支气管炎组血清IL-4浓度明显高于非特应性健康对照组;而毛细支气管炎组血清IFN-γ浓度明显低于健康对照组,尤其是合并特应性体质的毛细支气管炎患儿血清IFN-γ浓度明显低于特应性及非特应性健康对照组和非特应性毛细支气管炎组。提示毛细支气管炎尤其是合并特应性体质的毛细支气管炎存在Th1/Th2细胞失衡,这与国内外多项研究一致[22-23]。
目前多项研究证实,RUNX3在CD4和CD8效应T细胞的发育和功能中起着重要作用[24-25]。RUNX3通过对ThPOK的抑制,将MHCI类选择的胸腺细胞分化为CD8+ T细胞谱系,RUNX3表达沉默可抑制Th1细胞因子的分泌,促进Th2细胞因子的分泌[26]。RUNX3蛋白在CD8+ T细胞和Th1细胞中表达水平很高,其表达依赖于RUNX3d的转录,而在CD4+ T细胞发育过程中,RUNX3蛋白的表达需要受到限制,并且单纯的CD4+ T细胞和Th2细胞只表达RUNX3p,为一种低效的翻译,这使得RUNX3蛋白保持在足够低的水平,从而维持T细胞可塑性[27]。在Th1细胞中,RUNX3d以T-bet相关的方式被上调,并与T-bet共同促进Th1细胞的分化,诱导IFN-γ的分泌[28],同时抑制IL-4的表达。可见,当RUNX3表达受到抑制时,可出现Th1/Th2的失衡。Qiu等[29]证实在哮喘病人的CD4+ T细胞中RUNX3表达减少,而RUNX3的过度表达可以恢复Th1/Th2的平衡。进一步研究发现miR-371、miR-138、miR-544、miR-145和miR-214可以直接作用于RUNX3的3′UTR,使RUNX3表达减少,当同时抑制这5个microRNAs,可以调节RUNX3的表达从而改善Th1/Th2的平衡。本研究显示,RUNX3 mRNA水平在毛细支气管炎患儿尤其是合并特应性体质患儿中明显降低,同时血清IFN-γ浓度在此类患儿中也明显降低;与之相反,合并特应性体质的毛细支气管炎患儿血清IL-4浓度明显增高;提示RUNX3 mRNA水平与血清IFN-γ浓度呈正相关,而与血清IL-4浓度呈负相关。由此我们推测,RUNX3对毛细支气管炎尤其是合并特应性体质的患儿可以通过影响其体内的IFN-γ和IL-4的合成,进而对Th1/Th2平衡产生影响,从而进一步验证了RUNX3对T细胞分化的作用。
综上所述,特应性体质毛细支气管炎患儿PBMCs中RUNX3基因的表达下降,血清IL-4升高,IFN-γ下降,RUNX3基因的表达与这两个细胞因子显著相关,与哮喘的改变相似。PBMCs中RUNX3基因表达量的检测对于甄别毛细支气管炎中的哮喘高危儿具有一定价值,可以为哮喘的二级预防提供依据。
| [1] |
Xiong Y, Castro E, Yagi R, et al. Thpok-independent repression of Runx3 by Gata3 during CD4+ T-cell differentiation in the thymus[J]. Eur J Immunol, 2013, 43(4): 918-928. DOI:10.1002/eji.201242944 (  0) 0) |
| [2] |
Diao M, Min J, Guo F, et al. Effects of salbutamol aerosol combined with magnesium sulfate on T-lymphocyte subgroup and Th1/Th2 cytokines of pediatric asthma[J]. Exp Ther Med, 2017, 13(1): 117-120. DOI:10.3892/etm.2016.3912 (  0) 0) |
| [3] |
Fan L, Wang X, Fan L, et al. MicroRNA-145 influences the balance of Th1/Th2 via regulating RUNX3 in asthma patients[J]. Exp Lung Res, 2016, 42(8-10): 417-424. DOI:10.1080/01902148.2016.1256452 (  0) 0) |
| [4] |
Tanriver Y, Diefenbach A. Transcription factors controlling development and function of innate lymphoid cells[J]. Int Immunol, 2014, 26(3): 119-128. DOI:10.1093/intimm/dxt063 (  0) 0) |
| [5] |
Ferrini ME, Hong S, Stierle A, et al. CB2 receptors regulate natural killer cells that limit allergic airway inflammation in a murine model of asthma[J]. Allergy, 2017, 72(6): 937-947. DOI:10.1111/all.13107 (  0) 0) |
| [6] |
Barnig C, Cernadas M, Dutile S, et al. Lipoxin A4 regulates natural killer cell and type 2 innate lymphoid cell activation in asthma[J]. Sci Transl Med, 2013, 5(174): 174ra26. (  0) 0) |
| [7] |
Lambrecht BN, Hammad H. The immunology of asthma[J]. Nat Immunol, 2014, 16(1): 45-56. (  0) 0) |
| [8] |
Hong JY, Bentley JK, Chung Y, et al. Neonatal rhinovirus induces mucous metaplasia and airways hyperresponsiveness through IL-25 and type 2 innate lymphoid cells[J]. J Allergy Clin Immunol, 2014, 134(2): 429-439. DOI:10.1016/j.jaci.2014.04.020 (  0) 0) |
| [9] |
马琳.变态反应性皮肤病[M]//胡亚美, 江载芳.诸福棠实用儿科学(上册).第8版.北京: 人民卫生出版社, 2015: 722-730.
(  0) 0) |
| [10] |
申昆玲, 徐保平.呼吸系统疾病[M]//胡亚美, 江载芳.诸福棠实用儿科学(上册).第8版.北京: 人民卫生出版社, 2015: 1276-1277.
(  0) 0) |
| [11] |
Xue M, Chen LY, Wang WJ, et al. HOTAIR induces the ubiquitination of Runx3 by interacting with Mex3b and enhances the invasion of gastric cancer cells[J]. Gastric Cancer, 2018, 21(5): 756-764. DOI:10.1007/s10120-018-0801-6 (  0) 0) |
| [12] |
Kang KA, Piao MJ, Ryu YS, et al. Cytoplasmic localization of RUNX3 via histone deacetylase-mediated SRC expression in oxidative-stressed colon cancer cells[J]. J Cell Physiol, 2017, 232(7): 1914-1921. DOI:10.1002/jcp.25746 (  0) 0) |
| [13] |
Yang H, Fu J, Yao L, et al. RUNX3 is a key modulator during the epithelial-mesenchymal transition of alveolar type Ⅱ cells in animal models of BPD[J]. Int J Mol Med, 2017, 40(5): 1466-1476. DOI:10.3892/ijmm.2017.3135 (  0) 0) |
| [14] |
Guo C, Ahmad T, Beckly J, et al. Association of caspase-9 and RUNX3 with inflammatory bowel disease[J]. Tissue Antigens, 2011, 77(1): 23-29. DOI:10.1111/j.1399-0039.2010.01569.x (  0) 0) |
| [15] |
Fainaru O, Shseyov D, Hantisteanu S, et al. Accelerated chemokine receptor 7-mediated dendritic cell migration in Runx3 knockout mice and the spontaneous development of asthma-like disease[J]. Proc Natl Acad Sci U S A, 2005, 102(30): 10598-10603. DOI:10.1073/pnas.0504787102 (  0) 0) |
| [16] |
Fainaru O, Woolf E, Lotem J, et al. Runx3 regulates mouse TGF-β-mediated dendritic cell function and its absence results in airway inflammation[J]. EMBO J, 2014, 23(4): 969-979. (  0) 0) |
| [17] |
Pawelczyk M, Kowalski ML. The role of human parainfluenza virus infections in the immunopathology of the respiratory tract[J]. Curr Allergy Asthma Rep, 2017, 17(3): 16. DOI:10.1007/s11882-017-0685-2 (  0) 0) |
| [18] |
兰伟平, 王婧, 代传林, 等. 丙酸氟替卡松气雾剂或布地奈德混悬液干预对毛细支气管炎反复喘息的疗效观察[J]. 中国当代儿科杂志, 2016, 18(4): 316-319. (  0) 0) |
| [19] |
Wu Q, Tang Y, Hu X, et al. Regulation of Th1/Th2 balance through OX40/OX40L signalling by glycyrrhizic acid in a murine model of asthma[J]. Respirology, 2016, 21(1): 102-111. DOI:10.1111/resp.12655 (  0) 0) |
| [20] |
Russell CD, Unger SA, Walton M, et al. The human immune response to respiratory syncytial virus infection[J]. Clin Microbiol Rev, 2017, 2(30): 481-502. (  0) 0) |
| [21] |
Fonseca W, Lucey K, Jang S, et al. Lactobacillus johnsonii supplementation attenuates respiratory viral infection via metabolic reprogramming and immune cell modulation[J]. Mucosal immunol, 2017, 10(6): 1569-1580. DOI:10.1038/mi.2017.13 (  0) 0) |
| [22] |
Fedele G, Schiavoni I, Nenna R, et al. Analysis of the immune response in infants hospitalized with viral bronchiolitis shows different Th1/Th2 profiles associated with respiratory syncytial virus and human rhinovirus[J]. Pediatr Allergy Immunol, 2018, 29(5): 555-557. DOI:10.1111/pai.12919 (  0) 0) |
| [23] |
孙平平, 马少春. Th1/Th2失衡及毛细支气管炎特异性免疫治疗的研究进展[J]. 国际免疫学杂志, 2017, 40(1): 113-116. DOI:10.3760/cma.j.issn.1673-4394.2017.01.024 (  0) 0) |
| [24] |
Clark RA. Resident memory T cells: Runx and hide[J]. Sci Immunol, 2018, 3(19). pii: eaar5172. https://www.ncbi.nlm.nih.gov/pubmed/29305464
(  0) 0) |
| [25] |
Shan Q, Zeng Z, Xing S, et al. The transcription factor RUNX3 guards cytotoxic CD8+effector T cells against deviation towards follicular helper T cell lineage[J]. Nat Immunol, 2017, 18(8): 931-939. DOI:10.1038/ni.3773 (  0) 0) |
| [26] |
Serroukh Y, Gu-Trantien C, Hooshiar Kashani B, et al. The transcription factors Runx3 and ThPOK cross-regulate acquisition of cytotoxic function by human Th1 lymphocytes[J]. Elife, 2018, 7. pii: e30496. https://www.ncbi.nlm.nih.gov/pmc/articles/PMC5844691/
(  0) 0) |
| [27] |
Kim B, Sasaki Y, Egawa T. Restriction of nonpermissive RUNX3 protein expression in T lymphocytes by the kozak sequence[J]. J Immunol, 2015, 195(4): 1517-1523. DOI:10.4049/jimmunol.1501039 (  0) 0) |
| [28] |
Wang Y, Godec J, Ben-Aissa K, et al. The transcription factors T-bet and Runx are required for the ontogeny of pathogenic interferon-γ-producing T helper 17 cells[J]. Immunity, 2014, 40(3): 355-366. DOI:10.1016/j.immuni.2014.01.002 (  0) 0) |
| [29] |
Qiu YY, Zhang YW, Qian XF, et al. miR-371, miR-138, miR-544, miR-145, and miR-214 could modulate Th1/Th2 balance in asthma through the combinatorial regulation of RUNX3[J]. Am J Transl Res, 2017, 9(7): 3184-3199. (  0) 0) |
 2019, Vol. 21
2019, Vol. 21


