规律成簇间隔短回文重复(clustered regularly interspaced short palindromic repeats, CRISPR)是细菌和古生菌细胞保护自身不受病毒侵害的防御系统。当微生物遭到病毒入侵时,CRISPR RNA通过互补配对结合到病毒基因组,通过核酸酶Cas特异性地切割病毒DNA,以实现自我保护[1-2]。CRISPR/Cas9系统已经成为成熟高效、简单方便的基因编辑技术,广泛应用于人、鼠、斑马鱼、细菌、果蝇、酵母、线虫和农作物细胞的基因编辑[3]。
基因单核苷酸多态性(single nucleotide polymorphisms, SNPs)是人类最常见的基因突变类型,其中一些SNPs已被证实与人类疾病直接相关[4-6]。而先天性失氯性腹泻(congenital chloride diarrhea, CCD)或炎症性肠病(inflammatory bowel disease, IBD)相关性腹泻患者中均存在SLC26A3基因的异常[7-9]。其中,SNP rs386833481是引起CCD发生的一个重要突变体,该SNP编码一个错义突变,将SLC26A3基因392位点的DNA碱基从胞嘧啶(cytosine, C)变为鸟嘌呤(guanine, G),从而使SLC26A3蛋白131位点的氨基酸从脯氨酸(proline, P)变为精氨酸(arginine, R)[10]。本研究利用CRISPR/Cas9技术构建了SLC26A3 c.392C > G(p.P131R)突变体细胞模型,同时利用电-细胞-基质阻抗传感(electric-cell-substrate impedance sensing, ECIS)分析和Western blot法检测相对于野生型(WT)细胞,该SNP对细胞单层通透性的影响及其潜在作用机制,从而为进一步揭示SLC26A3 SNPs的生物学功能及相关腹泻发生机制和治疗奠定基础,同时也为CRISPR/Cas9基因编辑工具的应用开拓新的领域。
1 材料与方法 1.1 材料人结直肠腺癌上皮细胞Caco-2(HTB-37™,美国模式培养物集存库)和Top10 E.coli菌株(本科室保存);质粒pSpCas9n(BB)-2A-Puro(PX462)V2.0(#62987,美国Addgene公司);连接酶T7、限制性内切酶BbsI和PlasmidSafe核酸外切酶(美国NEB公司);细胞基因组DNA提取试剂盒(德国Qiagen公司);质粒提取试剂盒(美国Invitrogen公司),胎牛血清、青霉素-链霉素双抗、非必需氨基酸、谷氨酰胺、0.25%胰酶-EDTA、EMEM培养基及培养瓶(75 cm2)、6孔板(美国Gibco公司);氨苄霉素、嘌呤霉素(美国Sigma公司);sgRNA和ssODN序列合成委托美国IDT公司;rs386833481的TaqMan荧光标记探针、BCA定量试剂盒(美国Thermo Fisher公司);TNF-α(美国R & D Systems公司);小鼠源SLC26A3单克隆抗体(GTX34204,美国GeneTex公司);兔源GAPDH多克隆抗体(SC-25778,美国Santa Cruz Biotechnology公司)。
1.2 细胞培养Caco-2细胞用含有10%胎牛血清、1%非必需氨基酸、2 mmol/L谷氨酰胺和100 µg/mL青霉素-链霉素双抗的EMEM培养基培养,并置于37℃、5% CO2的恒温培养箱[11]。
1.3 单导RNA的设计利用CRISPR在线工具(http://crispr.mit.edu/),根据CRISPR/Cas9靶点设计原则生成特异性识别SLC26A3基因392位点的上下游单导RNA(single guide RNA, sgRNA)序列[3]。将SLC26A3基因序列提交至网站,并获得相应的备选序列,在正义链5'端添加CACCG,反义链5'端添加AAAC。本研究为Caco-2细胞系设计了两个位于SNP侧翼的sgRNA(sgRNA1、sgRNA2)和一个独特的供体DNA模板单链DNA寡核苷酸(ssODN)。每个sgRNA又包括一个正义链和反义链用于克隆,引物序列见表 1。
| 表 1 sgRNA克隆的引物序列 |
|
|
将pSpCas9n(BB)-2A-Puro(PX462)V2.0质粒经BbsI酶切形成的pSpCas9-puro载体与退火后的sgRNA混合,在T7 DNA连接酶作用下,以37℃ 5 min→23℃ 5 min的条件作用6个循环,经PlasmidSafe核酸外切酶处理后,将连接产物pSpCas9-SLC26A3转化至Top10 E.coli感受态细胞中(由0.1 mol/L的CaCl2制备而成),用氨苄霉素抗性筛选已成功导入pSpCas9-SLC26A3的感受态细胞,测序鉴定[12]。
1.5 SLC26A3 c.392C > G(p.P131R)表达细胞的筛选当Caco-2细胞密度达到70%时,与抗性筛选后的感受态细胞共培养,同时加入ssODN,并转染GFP监测转染效率。因重组质粒pSpCas9-SLC26A3含有嘌呤霉素抗性,能够使Caco-2细胞在嘌呤霉素筛选时存活[3],因此重组质粒转染Caco-2细胞48 h后,加入嘌呤霉素至终浓度为1.0 µg/mL,72 h后筛选已成功导入pSpCas9-SLC26A3的Caco-2活细胞至6孔板,继续扩增培养。
1.6 提取细胞基因组DNA并对目标片段进行Taqman基因型分析及Sanger测序将空载体质粒pSpCas9-puro转染到Caco-2细胞中,同时在嘌呤霉素进行抗性筛选重组质粒转染细胞前后,各挑取3个孔的Caco-2细胞进行扩增培养。分别收集2×106个细胞,按照基因组DNA提取试剂盒说明书,提取基因组DNA(genome DNA, gDNA)。Taqman荧光标记探针靶向标记的rs386833481 SNP用于ViiA7序列检测系统中DNA的扩增。采用dH2O代替DNA作为阴性对照,以已知基因型的WT Caco-2细胞DNA样品作为阳性对照[13]。
提取阳性对照和嘌呤霉素抗性筛选后Caco-2细胞的gDNA,通过PCR法扩增目的基因,SLC26A3上游引物:5'-ACCCTCACCCTCTCGTCATC-3',下游引物:5'-TGCTGGGATTACAGGCGTGAG-3',片段长度440 bp,引物的设计与合成由美国Integrated DNA Technologies公司完成。将PCR产物进行琼脂糖凝胶电泳检测,回收目的DNA基因,进行Sanger测序。
Taqman基因型分析检测的是正义链上的序列,Sanger测序检测的是反义链上的序列。
1.7 ECIS分析检测细胞单层通透性利用ECIS(美国Applied Biophysics公司)分析检测细胞单层屏障功能,也称跨上皮电阻(transepithelial electric resistance, TEER)[14]。实验分为正常对照组、P131R组、TNF-α组和P131R+TNF-α组。正常对照组为WT Caco-2细胞,P131R组为SLC26A3 c.392C > G(p.P131R)表达的Caco-2细胞。两组细胞分别接种到用10 mmol/L半胱氨酸和纤维连接蛋白(20 µg)预处理过的每孔含有40个金电极(8W10E+电极板,美国Applied Biophysics公司)的阵列室中。当32 000 Hz频率下的电容达到10 nF时,表明细胞单层形成紧密连接。然后将细胞饥饿2 h,TNF-α组和P131R+TNF-α组分别在WT Caco-2细胞和SLC26A3 c.392C > G(p.P131R)表达的Caco-2细胞上用100 ng/mL TNF-α[15]处理2.5 h。记录单层细胞的TEER值,最终结果数据通过随时间变化的标准化电阻呈现。实验独立重复3次。
1.8 Western blot法检测SLC26A3 c.392C > G(p.P131R)表达细胞中SLC26A3蛋白的表达变化实验分为正常对照组和P131R组,正常对照组为WT Caco-2细胞,P131R组为SLC26A3 c.392C > G(p.P131R)表达的Caco-2细胞。细胞裂解液作用于两组Caco-2细胞,在冰上操作并收集到1.5 mL的离心管中,以13 000 r/min的速度在4℃离心机中离心10 min。通过BCA定量蛋白质浓度,将30 μg蛋白通过SDS-PAGE电泳,湿法电转移至PVDF膜,5%脱脂牛奶室温封闭1 h。分别加入SLC26A3一抗(1 : 1 000)、GAPDH一抗(1 : 2 000),于4℃摇床上过夜,再加入辣根过氧化物酶标记的山羊抗兔或山羊抗小鼠二抗(1 : 5 000,美国Santa Cruz公司),室温孵育1 h,再用TBST缓冲液洗涤3次,每次15 min,加入发光试剂显色。利用Fluorchem M成像仪(美国ProteinSimple公司)进行成像,利用AlphaVIEW SA软件进行量化。以GAPDH为内参,以目的条带与内参条带的灰度比值作为该蛋白的相对表达量。实验独立重复4次。
1.9 统计学分析采用SPSS 24.0统计软件对数据进行统计学分析,计量资料以均数±标准差(x±s)表示,两组间比较采用两独立样本t检验;多组间比较采用单因素方差分析,组间两两比较采用SNK-q检验。P < 0.05为差异有统计学意义。
2 结果 2.1 重组表达载体pSpCas9-SLC26A3的构建与鉴定将每个sgRNA的正义和反义寡核苷酸经退火后插入到表达Cas9/gRNA支架的pSpCas9n(BB)-2A-Puro(PX462)V2.0质粒BbsI位点,连接产物pSpCas9-SLC26A3转化到Top10 E.coli感受态细胞中,涂于含氨苄霉素的平板上,挑取单克隆细胞进行测序,选取连接正确的载体。在Addgene官网上选取pSpCas9n(BB)-2A-Puro(PX462)V2.0质粒BbsI剪切位点筛选克隆的测序引物:5'-GACTATCATATGCTTACCGT-3'(Lot: 14868230)。正确连接的重组载体测序结果表明,54~73位碱基是sgRNA的插入序列,见图 1。
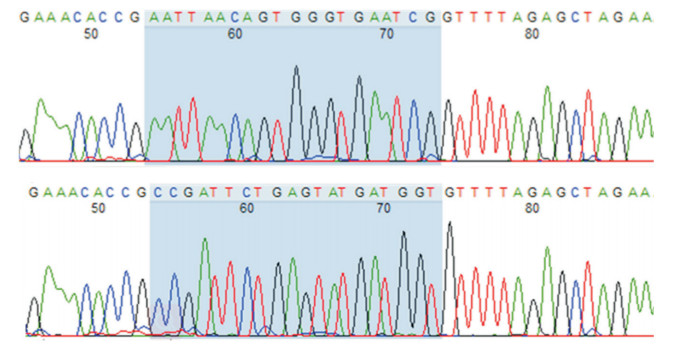
|
图 1 pSpCas9-SLC26A3载体的测序结果 上图和下图的54~73位碱基分别是sgRNA1和sgRNA2的插入序列(蓝色背景区域所示)。 |
成功获得SLC26A3 c.392C > G(p.P131R) SNP纯合子表达(CC型)的Caco-2细胞;阳性对照的基因型与空载体对照基因型一致,均为GG型;嘌呤霉素抗性筛选Caco-2细胞前检测到SLC26A3 c.392C > G(p.P131R)SNP杂合子表达(CG型);阴性对照未检测到基因型。见表 2。
| 表 2 Taqman基因型分析结果 |
|
|
嘌呤霉素抗性筛选后的Caco-2细胞目的基因序列与拟编辑为SLC26A3 c.392C > G(p.P131R)的序列吻合,进一步证实了SLC26A3 c.392C > G(p.P131R)的基因编辑成功,见图 2。
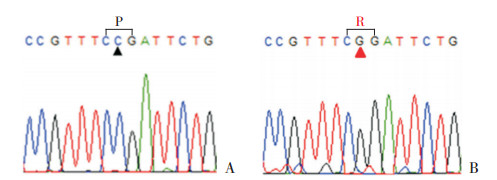
|
图 2 目的序列的测序结果 A:阳性对照细胞的测序结果;B:嘌呤霉素抗性筛选后的Caco-2细胞的测序结果。箭头所示为突变位点。[P]脯氨酸;[R]精氨酸。 |
鉴于肠黏膜屏障功能障碍是各种腹泻发生的直接原因,因此本课题组利用ECIS分析检测了SLC26A3 c.392C > G(p.P131R) SNP对Caco-2肠上皮细胞单层通透性的影响。结果发现:正常对照组、P131R组、TNF-α组和P131R+TNF-α组肠上皮细胞单层通透性比较差异有统计学意义(n=3,F=705.580,P < 0.001)。与正常对照组肠上皮细胞(0.996±0.014)相比,P131R组细胞单层通透性(0.901±0.010)明显增加(P=0.0002)。同时,与TNF-α组(0.672±0.225)相比,P131R+TNF-α组肠上皮细胞单层通透性(0.513±0.006)明显增加(P < 0.001),提示SLC26A3 c.392C > G(p.P131R)细胞对TNF-α诱导的细胞膜通透性的增加更为敏感。见图 3。
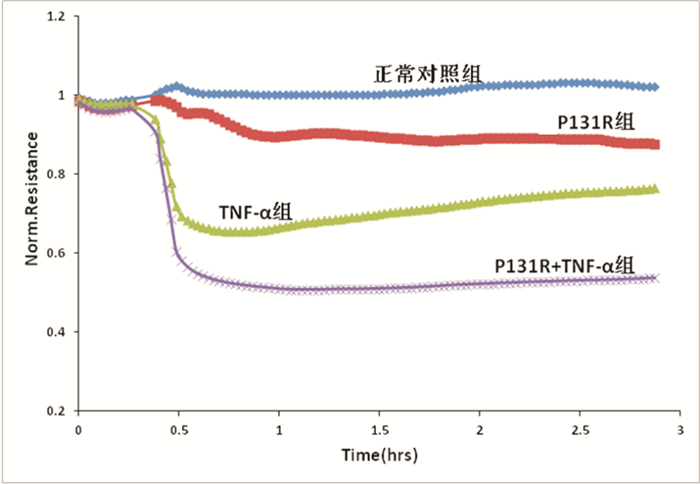
|
图 3 SLC26A3 c.392C>G(p.P131R)对肠上皮细胞单层通透性的影响 |
为了明确SLC26A3 c.392C > G(p.P131R)细胞单层通透性增加的潜在机制,本课题组利用Western blot对SLC26A3 c.392C > G(p.P131R)表达细胞中SLC26A3的蛋白水平进行了检测。结果发现,与正常对照组肠上皮细胞SLC26A3蛋白相对表达水平(0.974±0.098)相比,SLC26A3蛋白在P131R组细胞中的相对表达水平(0.460±0.150)显著下降(n=4,t=5.744,P=0.001),见图 4。
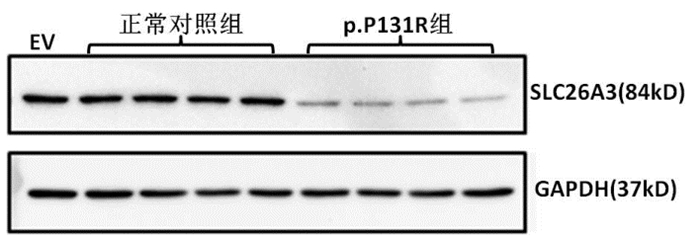
|
图 4 Western blot检测正常对照组和P131R组细胞中SLC26A3蛋白的表达情况 P131R组SLC26A3蛋白表达水平低于正常对照组,每组平行样本数为4。 |
CRISPR/Cas9基因编辑系统被广泛应用于生物学及医学各领域,包括基因表达与表观遗传学调控,遗传物质的动态可视化,动物(细胞)模型的建立,基因疗法的探索等[3, 16]。在gRNA引导下,Cas9核酸酶靶向特定DNA序列并诱发DNA双链断裂(double strand break, DSB)。DSB可由两种内源性的修复机制修复,即易于出错的非同源末端连接(non-homologous end joining, NHEJ)和同源重组介导的修复(homology-directed repair, HDR),从而实现基因敲除、敲入和染色体转为等编辑[3, 17]。在易出错的NHEJ过程中,DSB末端由内源性DNA修复机制处理并重新连接,这可能导致连接位点的随机突变。在基因的编码区域内发生插入/删除突变引起移码和过早终止密码子的发生,从而导致基因敲除。在HDR过程中,需要提供质粒或ssODN形式的修复模板,该途径具有高保真度和精确编辑的特点。DNA的单链剪切也可以诱导HDR。在本研究中,通过提供ssODN的修复模板,最终实现了HDR修复,从而避免了插入/删除等随机突变的发生。
近两年,哈佛大学Komor等[18]的实验室创建了新的Cas9融合蛋白,可作为“单碱基编辑器”。这些融合蛋白包含dCas9或Cas9切口酶及大鼠胞苷脱氨酶APOBEC1,它能将C转化为尿嘧啶(U),而无需切割DNA。之后,通过DNA复制或修复,U被转化成胸腺嘧啶(T)。这种碱基编辑策略有望治疗数百种遗传病。虽然Komor等[18]的工作成果是激动人心的,因为它不再需要DNA切割和修复,但仍然存在一些需要解决的问题。首先,最明显的问题是酶,我们不仅仅需要C→T或G→腺嘌呤(A)的酶,还需要G→C或T→A的酶。第二个潜在的问题是编辑窗口小。为了达到最佳的编辑效果,目标碱基应该在第4~8位。由于前间隔序列邻近基序的要求限制了可靶定序列的数量,利用其他的Cas9酶也许能扩展编辑范围。因此,目前大部分研究中采取的仍然是HDR修复DSB的策略进行基因编辑。
SNPs是人类最常见的遗传变异类型。大量研究发现:SLC26A3基因SNPs在CCD的发生发展中起着关键作用[6, 19-20]。此外,炎症动物模型和IBD相关性腹泻患者SLC26A3的表达下降[8-9]。同时近期有文献报道,克罗恩病(Crohn's disease, CD)和溃疡性结肠炎(ulcerative colitis, UC)均与SLC26A3基因SNPs及其表达相关[21-22]。因此,我们认为SLC26A3功能缺陷或表达受损可能是该类患者发生腹泻的重要机制之一。在本研究中,本课题组利用CRISPR/Cas9技术中HDR介导的DSB修复机制筛选SLC26A3 c.392C > G(p.P131R)表达细胞过程中,遇到的主要问题及解决方案:(1)重组真核表达质粒pSpCas9-SLC26A3构建时,连接产物转化到Top10感受态细胞中,涂于含氨苄霉素的平板后,次日平板上并未长出可挑选克隆,通过多次实践,我们发现热休克的最优条件为42℃,30 s,在热休克改变细胞膜的流动性时,目的DNA才能通过细胞表面的内陷高效进入Top10感受态细胞。(2)实验中一开始用常规0.5 µg/mL嘌呤霉素抗性筛选时,Taqman基因型分析结果均显示为杂合等位基因。因此我们检测了Caco-2细胞对嘌呤霉素的敏感性,分别用终浓度为0、0.5、1.0、1.5、2.0 µg/mL的嘌呤霉素处理WT Caco-2细胞72 h后,结果发现1.0、1.5、2.0 µg/mL组的细胞全部凋亡,而0.5 µg/mL组仍有三分之一细胞存活,因此本研究将筛选SLC26A3 c.392C > G(p.P131R)表达细胞的嘌呤霉素浓度调整为1.0 µg/mL,最终成功获得了纯合子基因的突变体细胞,并进行了基因测序。此外,最新研究表明:SLC26A3可作为上皮屏障的组成部分与紧密连接蛋白ZO-1相互作用,SLC26A3的下调或敲除可通过影响紧密连接蛋白的表达从而破坏上皮屏障,SLC26A3的过表达可逆转紧密连接蛋白的变化[22],结合本研究中ECIS和Western blot的结果,提示SLC26A3 c.392C > G(p.P131R)可能通过降低SLC26A3蛋白的表达而导致肠上皮细胞通透性增加从而引起相关性腹泻的发生,该发现仍需进一步的体外体内实验来证实。
综上所述,本研究首次建立了表达CCD相关基因SLC26A3 c.392C > G(p.P131R)多态性的细胞模型,同时发现该SNP通过降低SLC26A3蛋白的表达而增加细胞单层的通透性,从而为进一步深入研究相关腹泻的发生机制和治疗提供了更多的理论依据,也为CRISPR/Cas9基因编辑工具的应用开拓新的领域。
| [1] |
Jiang F, Doudna JA. CRISPR-Cas9 structures and mechanisms[J]. Ann Rev of Biophys, 2017, 46(1): 505-529. (  0) 0) |
| [2] |
Erdmann V, Barciszewski J. From nucleic acids sequences to molecular medicine[M]. Berlin: Springer, 2012: 121-143.
(  0) 0) |
| [3] |
Ran FA, Hsu PD, Wright J, et al. Genome engineering using the CRISPR-Cas9 system[J]. Nat Protoc, 2013, 8(11): 2281-2308. DOI:10.1038/nprot.2013.143 (  0) 0) |
| [4] |
Zhang W, He T, Wang Q, et al. Interleukin-1 receptor-associated kinase-2 genetic variant rs708035 increases NF-κB activity through promoting TRAF6 ubiquitination[J]. J Biol Chem, 2014, 289(18): 12507-12519. DOI:10.1074/jbc.M113.538009 (  0) 0) |
| [5] |
Heruth DP, Katherine S, Nini Z, et al. Genetic association of single nucleotide polymorphisms with acetaminophen-induced hepatotoxicity[J]. J Pharmacol Exp Ther, 2018, 367(1): 95-100. DOI:10.1124/jpet.118.248583 (  0) 0) |
| [6] |
Katherine S, Heruth DP, Nini Z, et al. Identification of novel regulatory genes in APAP induced hepatocyte toxicity by a genome-wide CRISPR-Cas9 screen[J]. Sci Rep, 2019, 9(1): 1396. DOI:10.1038/s41598-018-37940-6 (  0) 0) |
| [7] |
Matsunoshita N, Nozu K, Yoshikane M, et al. Congenital chloride diarrhea needs to be distinguished from Bartter and Gitelman syndrome[J]. J Hum Genet, 2018, 1(63): 887-892. (  0) 0) |
| [8] |
Chatterjee I, Kumar A, Castilla-Madrigal RM, et al. CDX2 upregulates SLC26A3 gene expression in intestinal epithelial cells[J]. Am J Physiol Gastrointest Liver Physiol, 2017, 313(3): G256-G264. DOI:10.1152/ajpgi.00108.2017 (  0) 0) |
| [9] |
Asano K, Matsushita T, Umeno J, et al. A genome-wide association study identifies three new susceptibility loci for ulcerative colitis in the Japanese population[J]. Nat Genet, 2009, 41(12): 1325-1329. DOI:10.1038/ng.482 (  0) 0) |
| [10] |
Wedenoja S, Pekansaari E, Höglund P, et al. Update on SLC26A3 mutations in congenital chloride diarrhea[J]. Hum Mutat, 2011, 32(7): 715-722. DOI:10.1002/humu.21498 (  0) 0) |
| [11] |
Freedman JC, Hendricks MR, McClane BA, et al. The potential therapeutic agent mepacrine protects Caco-2 cells against Clostridium perfringens enterotoxin action[J]. mSphere, 2017, 2(4): pii:e00352-17. (  0) 0) |
| [12] |
Cong L, Ran FA, Cox D, et al. Multiplex genome engineering using CRISPR/Cas systems[J]. Science, 2013, 339(6121): 819-823. DOI:10.1126/science.1231143 (  0) 0) |
| [13] |
Katherine S, Suman C, Dmitry G, et al. Identification of novel single nucleotide polymorphisms associated with acute respiratory distress syndrome by exome-seq[J]. PLoS One, 2014, 9(11): e111953. DOI:10.1371/journal.pone.0111953 (  0) 0) |
| [14] |
Kho DT, Johnson RH, O'Carroll SJ, et al. Biosensor technology reveals the disruption of the endothelial barrier function and the subsequent death of blood brain barrier endothelial cells to sodium azide and its gaseous products[J]. Biosensors (Basel), 2017, 7(4): pii:E41. (  0) 0) |
| [15] |
Ding X, Li D, Li M, et al. SLC26A3(DRA) prevents TNFalpha-induced barrier dysfunction and dextran sulfate sodiuminduced acute colitis[J]. Lab Invest, 2018, 98(4): 462-476. DOI:10.1038/s41374-017-0005-4 (  0) 0) |
| [16] |
Ding Y, Li H, Chen LL, et al. Recent advances in genome editing using CRISPR/Cas9[J]. Front Plant Sci, 2016, 7: 703. (  0) 0) |
| [17] |
Bernheim A, Calvo-Villamañán A, Basier C, et al. Inhibition of NHEJ repair by type Ⅱ-A CRISPR-Cas systems in bacteria[J]. Nat Commun, 2017, 8(1): 2094. DOI:10.1038/s41467-017-02350-1 (  0) 0) |
| [18] |
Komor AC, Kim YB, Packer MS, et al. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage[J]. Nature, 2016, 533(7603): 420-424. DOI:10.1038/nature17946 (  0) 0) |
| [19] |
Bhardwaj S, Pandit D, Sinha A, et al. Congenital chloride diarrhea-novel mutation in SLC26A3 gene[J]. Indian J Pediatr, 2016, 83(8): 859-861. DOI:10.1007/s12098-015-1944-7 (  0) 0) |
| [20] |
Thiagarajah JR, Kamin DS, Acra S, et al. Advances in evaluation of chronic diarrhea in infants[J]. Gastroenterology, 2018, 154(8): 2045-2059. DOI:10.1053/j.gastro.2018.03.067 (  0) 0) |
| [21] |
邵晓晓, 夏宣平, 曹曙光, 等. 溶质相关载体26A3基因多态性及其在结肠组织中的表达水平与克罗恩病的关系[J]. 中华消化杂志, 2017, 37(10): 684-691. DOI:10.3760/cma.j.issn.0254-1432.2017.10.008 (  0) 0) |
| [22] |
Shao XX, Lin DP, Sun L, et al. Association of ulcerative colitis with solute-linked carrier family 26 member A3 gene polymorphisms and its expression in colonic tissues in Chinese patients[J]. Int J Colorectal Dis, 2018, 33(9): 1169-1172. DOI:10.1007/s00384-018-3097-4 (  0) 0) |
 2019, Vol. 21
2019, Vol. 21


