2. 深圳清华大学研究院等离子体纳米生物芯片研发中心, 广东 深圳 518057
缺氧缺血性脑病(hypoxic-ischemic encephalo-pathy, HIE)是常见的可造成严重危害的新生儿脑病,发生率约为1.5‰,是新生儿死亡、严重脑瘫、神经感觉缺陷和认知障碍的重要病因之一[1-2]。目前尚未有针对缺氧缺血性脑损伤的有效治疗措施,其疾病机制也尚不明确。研究显示,表观遗传学与氧化应激反应密切相关,可参与调节活性氧(reactive oxygen species, ROS),并与癫痫、缺血性脑中风等神经系统疾病的细胞凋亡、坏死等生物学过程有关[3-5]。在HIE中,表观遗传修饰也被证实参与调节疾病的致病过程[6]。但是HIE中,大脑组织线粒体DNA(mitochondrial DNA, mtDNA)的表观遗传修饰,如5-羟甲基胞嘧啶(5-hydroxymethylcytosine, 5hmC)的修饰水平的变化尚未见报道。
本研究构建了新生儿HIE大鼠模型,通过比较对照组和模型组之间mtDNA的5hmC表达水平的差异探讨HIE模型鼠mtDNA中表观遗传修饰的作用。
1 材料与方法 1.1 动物造模与样本处理随机将24只7日龄雄性Sprague-Dawley(SD)大鼠随机分为对照组、24 h模型组和48 h模型组,每组8只。采用动脉结扎的方法造模,分离各组大鼠右颈总动脉,模型组大鼠用线结扎血管两端(相隔一定距离),缝合伤口,等待大鼠苏醒,苏醒后的大鼠放置于8%氧气的容器内2 h;对照组不进行动脉结扎及缺氧处理[7]。实验结束后,解剖各组大鼠,收集大脑皮质组织,使用Mitochondrial DNA Isolation Kit(Biovision,德国)提取各组大鼠大脑皮质组织mtDNA。操作按照试剂盒说明书进行。
1.2 线粒体羟甲基化测序和检测用琼脂糖凝胶电泳检测提取mtDNA的完整性,用Nanodrop2000检测DNA纯度,并用Qubit对DNA浓度及总量进行定量。超声将mtDNA打断成为350 bp左右的片段,采用化学氧化法结合重亚硫酸盐转化测序技术(oxidative bisulfite sequencing, oxBS-Seq)测序并构建文库,片段经末端修复、加A碱基、加接头后纯化产物。高温变形后用氧化剂对DNA进行氧化反应,随后进行Bisulfite(BS)处理、PCR扩增,所得文库经质检合格后,在illumina X ten PE150平台上机测序。
采用试剂盒MethylFlashTM Hydroxymethylated DNA Quantification Kit(Epigentek,美国)检测大鼠大脑皮质组织mtDNA的5hmC甲基化水平,实验步骤按照说明书进行。
1.3 Western blot检测按照试剂盒Tissue Mitochondria Isolation Kit(上海碧云天生物技术有限公司)说明书分离大鼠大脑皮质组织线粒体。液氮研磨大脑皮质组织线粒体后,使用RIPA在低温条件下进行组织裂解,离心去除沉淀,收集上清。使用BCA蛋白定量试剂盒(上海碧云天生物技术有限公司)方法进行蛋白定量。使用10%SDS聚丙烯酰胺凝胶电泳分离蛋白并将凝胶上的蛋白转移到PVDF膜上,4℃条件下过夜孵育1011易位甲基胞嘧啶双加氧酶1(ten-eleven translocation protein1, TET1)多克隆抗体(美国Abcam公司,1 : 1 000)、TET2多克隆抗体(美国Invitrogen公司,1 : 1 000)和DNA甲基转移酶(DNA methyltransferase 1, DNMT1)多克隆抗体(武汉博士德生物工程有限公司,1 : 100)一抗,室温下孵育HRP标记山羊抗鼠或山羊抗兔IgG多克隆抗体(广州永诺生物科技有限公司,1 : 3 000)二抗1 h后,加入ECL化学发光液及显色液显色。兔抗人线粒体外膜转位酶20(translocase of outer mitochondrial membrane 20,TOMM20,武汉博士德生物工程有限公司,1 : 100)单克隆抗体作为内参蛋白。胶片显影后扫描,进行灰度值定量分析,结果以目的蛋白与内参蛋白的灰度值比进行表示。
1.4 生物信息学分析进行单个位点差异5hmC分析,对BS和oxBS测序数据中的单个CpG位点进行卡方检验:oxBS数据中,两个样本统计检验结果P < 0.05的位点定义为差异5-甲基胞嘧啶(5mC)位点;按照公式计算其羟甲基化水平:
羟甲基化水平=甲基化和羟甲基化水平-真实甲基化水平。
BS数据中,两个样本统计检验结果P < 0.05的位点,且该位点样本间修饰水平变化趋势与oxBS数据中样本间修饰水平变化趋势相反,则该位点定义为5hmC位点。
1.5 统计学分析采用SPSS 22.0统计软件对数据进行统计学分析。计量资料以均数±标准差(x±s)形式表示,两组间比较采用t检验,多组间比较采用方差分析,组间两两比较采用SNK-q检验;计数资料采用百分率(%)表示,对测序数据进行卡方检验筛选有意义位点。P < 0.05为差异有统计学意义。
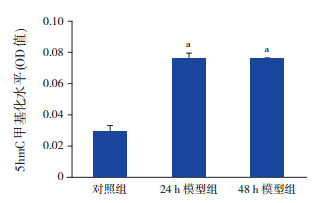
2 结果 2.1 各组大鼠大脑皮质mtDNA的5hmC甲基化水平变化对照组、模型组24 h、模型组48 h大鼠大脑皮质mtDNA的5hmC甲基化水平比较差异有统计学意义(F=93.810,P < 0.001)。模型组24 h、模型组48 h的5hmC甲基化水平显著高于对照组(P < 0.05),两模型组之间比较差异无统计学意义(P > 0.05)。见图 1。
|
图 1 各组大鼠大脑皮质mtDNA的5hmC甲基化水平比较(n=8) a示与对照组比较,P < 0.05。 |
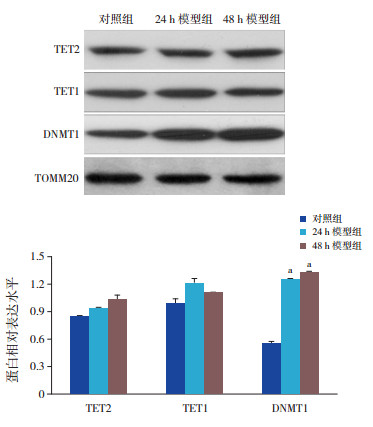
Western blot检测甲基化过程重要酶TET1、TET2和DNMT1的表达结果显示:TET1和TET2的蛋白表达水平在各组间比较差异无统计学意义(P < 0.05);但与对照组比较,DNMT1的表达水平在24 h组和48 h组均显著上调(P < 0.05)。见图 2。
|
图 2 Western blot检测各组大鼠大脑皮质线粒体中甲基化过程重要酶TET1、TET2和DNMT1的蛋白表达水平 上图为各组蛋白表达电泳图;下图为各组蛋白相对表达量统计图(n=8),a示与对照组比较,P < 0.05。 |
基于BS和oxBS测序数据中的单个CpG位点进行卡方检验,筛选差异羟甲基化位点。结果显示,相比对照组,24 h模型组9个基因15个位点呈现5hmC甲基化水平差异(P < 0.05,表 1)。在48 h模型组中,5hmC甲基化水平在线粒体8个基因的14个位点与对照组相比呈现差异(P < 0.05,表 2)。
| 表 1 24 h模型组和对照组多个线粒体基因位点的5hmC甲基化率比较 (%) |
|
|
| 表 2 48 h模型组和对照组多个线粒体基因位点的5hmC甲基化率比较 (%) |
|
|
尽管已有研究提示甲基化修饰与新生大鼠大脑的缺血缺氧损伤有关,但是有关mtDNA中的甲基化修饰研究仍较为缺乏。本研究发现缺血缺氧大鼠大脑皮质组织mtDNA的5hmC水平比对照组显著升高,mtDNA甲基化过程的关键酶DNMT1的表达也升高,其中,在24 h模型组中有15个mtDNA的位点5hmC水平呈现差异,48 h模型组中有14个mtDNA的位点呈现5hmC水平差异。
线粒体是细胞内细胞器,含有mtDNA,是一种环状双链DNA,长度为16 569 bp,编码tRNA、rRNA和呼吸链重要组分蛋白,包括ND基因(ND1、ND2、ND3、ND4、ND4L、ND5和ND6)、细胞色素b(cytochrome b, cyb)、细胞色素C氧化酶(CO1、CO2和CO3)、ATPase 6和ATPase 8[8-9]。与核DNA一样,mtDNA也可被甲基化修饰,目前发现甲基化形式有5hmC和5mC [10]。5hmC由TET蛋白(包括TET1、TET2和TET3)催化而形成[11]。具体作用机制是:DNMT1在线粒体基质中与CpG富集的mtDNA位点结合,促进mtDNA甲基化的形成和维持,进一步影响5mC向5hmC的转化[11-12]。本研究中,我们发现在大鼠HIE模型中,缺氧缺血处理后24 h和48 h均出现大脑皮质组织mtDNA中5hmC总量的升高,同时DNMT1也呈现高表达,这一对应的关系,支持DNMT1在mtDNA甲基化中的促进作用。但是,未观察到TET1和TET2的蛋白表达水平有改变。此前有研究表明衰老模型大鼠脑组织中5mC和5hmC合成涉及的酶DNMT1、TET1、TET2和TET3的表达变化,发现在额叶皮质中,衰老降低了mtDNMT1信使RNA水平而不影响TET1~TET3[13]。而在小脑中,TET2和TET3表达增加但DNMT1则未受影响。由此提示并非所有的甲基化酶都在同一时间或者同一处理背景下发生变化。
新生儿缺氧可导致大脑发育过程中发生不可逆损伤,增加新生儿大脑中枢神经系统畸形的风险及后代长期神经系统并发症[14]。缺氧与DNA修饰有关。在发育过程中,整个不同的大脑皮质层中均出现DNA甲基化程序启动的现象。研究显示,胎儿中大脑皮质细胞水平的DNA甲基化水平在刺激下可发生变化,如酒精可改变潜在的表型异常导致皮质变薄,系统性红斑狼疮患者抗氧化反应降低也与DNA甲基化有关。值得注意的是,作为大脑中的两个关键甲基化标记,随着神经系统发育逐渐成熟,5mC和5hmC的修饰分布也发生相应改变,提示缺氧对DNA修饰的调控作用[15]。然而缺氧环境对大脑皮质mtDNA的影响则知之甚少。多项研究表明,在缺氧环境下,线粒体参与能量调节,可生成受体操纵性钙通道并诱导细胞凋亡[16-18]。在本研究中,我们进一步观察到缺氧对新生大鼠大脑皮质组织mtDNA修饰水平的影响。在大脑皮质组织mtDNA中检测到9个基因15个位点和8个基因14个位点在造模后24 h和48 h呈现5hmC修饰水平差异,提示缺血缺氧的环境可以造成大脑皮质细胞mtDNA的5hmC修饰水平差异,而这些mtDNA的甲基化修饰可导致mtDNA基因异常表达。
此前的研究发现mtDNA的5hmC修饰与细胞增殖及凋亡等行为密切相关。研究显示,5hmC修饰可参与基因激活及表达量增强从而调节细胞分化、发育和衰老等生物学行为[19-21]。此外,在疾病的发生过程中,DMNT转移酶可通过线粒体介导的细胞凋亡途径,影响Bcl-2和BAX的表达,最终导致细胞凋亡[22-23]。相反,去线粒体甲基化可引发一系列生物学行为的改变,包括细胞增殖增加和细胞凋亡减少等现象[24]。在本研究中,我们观察到模型大鼠中线粒体CO1、ATP6和ND2基因位点存在5hmC修饰水平差异,提示5hmC在模型中可能对基因转录具有激活作用。我们推测这种激活可能促进MT-CO1、ATP6和ND2的表达,调节与线粒体活性氧的产生和细胞凋亡的发生有关的分子事件[17-18]。此外,在胎儿发育期间,缺氧这样剧烈的环境条件可能通过刺激各种表观遗传修饰而影响分子水平的脑发育,并因此增加后代神经障碍的易感性。因此,本研究结果提示表观遗传修饰可能是胎儿发育过程中早期环境因素调节基因组信息输出并影响生命后期表型发育的主要机制。
综上,本研究发现5hmC修饰水平的差异可能与缺氧环境下线粒体的氧化应激调节有关,可能是HIE发展过程中造成大脑损伤的重要机制之一。
| [1] |
Volpe JJ. Neonatal encephalopathy:an inadequate term for hypoxic-ischemic encephalopathy[J]. Ann Neurol, 2012, 72(2): 156-166. DOI:10.1002/ana.v72.2 (  0) 0) |
| [2] |
Laptook AR, Shankaran S, Tyson JE, et al. Effect of therapeutic hypothermia initiated after 6 hours of age on death or disability among newborns with hypoxic-ischemic encephalopathy:arandomized clinical trial[J]. JAMA, 2017, 318(16): 1550-1560. DOI:10.1001/jama.2017.14972 (  0) 0) |
| [3] |
Qureshi IA, Mehler MF. Emerging role of epigenetics in stroke:part 1:DNA methylation and chromatin modifcations[J]. Arch Neurol, 2010, 67(11): 1316-1322. (  0) 0) |
| [4] |
Hwang JY, Aromolaran KA, Zukin RS. Epigenetic mechanisms in stroke and epilepsy[J]. Neuropsychopharmacology, 2013, 38(1): 167-182. DOI:10.1038/npp.2012.134 (  0) 0) |
| [5] |
Thompson JW, Dave KR, Young JI, et al. Ischemic preconditioning alters the epigenetic profile of the brain from ischemic intolerance to ischemic tolerance[J]. Neurotherapeutics, 2013, 10(4): 789-797. DOI:10.1007/s13311-013-0202-9 (  0) 0) |
| [6] |
Felling RJ, Song H. Epigenetic mechanisms of neuroplasticity and the implications for stroke recovery[J]. Exp Neurol, 2015, 268: 37-45. DOI:10.1016/j.expneurol.2014.09.017 (  0) 0) |
| [7] |
Wang Y, Cheung PT, Shen GX, et al. Hypoxic-ischemic brain injury in the neonatal rat model:relationship between lesion size at early MR imaging and irreversible infarction[J]. AJNR Am J Neuroradiol, 2006, 27(1): 51-54. (  0) 0) |
| [8] |
Blanch M, Mosquera JL, Ansoleaga B, et al. Altered mitochondrial DNA methylation pattern in alzheimer diseaserelated pathology and in parkinson disease[J]. Am J Pathol, 2016, 186(2): 385-397. DOI:10.1016/j.ajpath.2015.10.004 (  0) 0) |
| [9] |
Asin-Cayuela J, Gustafsson CM. Mitochondrial transcription and its regulation in mammalian cells[J]. Trends Biochem Sci, 2007, 32(3): 111-117. DOI:10.1016/j.tibs.2007.01.003 (  0) 0) |
| [10] |
Iacobazzi V, Castegna A, Infantino V, et al. Mitochondrial DNA methylation as a next-generation biomarker and diagnostic tool[J]. Mol Genet Metab, 2013, 110(1-2): 25-34. DOI:10.1016/j.ymgme.2013.07.012 (  0) 0) |
| [11] |
Shock L S, Thakkar P V, Peterson E J, et al. D N A methyltransferase 1, cytosine methylation, and cytosine hydroxymethylation in mammalian mitochondria[J]. Proc Natl Acad Sci U S A, 2011, 108(9): 3630-3635. DOI:10.1073/pnas.1012311108 (  0) 0) |
| [12] |
Ito S, D'Alessio AC, Taranova OV, et al. Role of Tet proteins in 5mC to 5hmC conversion, ES-cell self-renewal and inner cell mass specifcation[J]. Nature, 2010, 466(7310): 1129-1133. DOI:10.1038/nature09303 (  0) 0) |
| [13] |
Öztürk NC, Resendiz M, Öztürk H, et al. DNA methylation program in normal and alcohol-induced thinning cortex[J]. Alcohol, 2017, 60: 135-147. DOI:10.1016/j.alcohol.2017.01.006 (  0) 0) |
| [14] |
Zinovkina LA, Zinovkin RA. DNA methylation, mitochondria, and programmed aging[J]. Biochemistry (Mosc), 2015, 80(12): 1571-1577. DOI:10.1134/S0006297915120044 (  0) 0) |
| [15] |
Garcia EG, Olmos CF, Matilla MF, et al. DNA hypomethylation and decreased hydroxymethylation is associated with decreased antioxidant response in systemic lupus erythematous patients[J]. Ann Rheum Dis, 2017, 76(Suppl 2): 291. (  0) 0) |
| [16] |
Naveenkumar SK, Hemshekhar M, Sundaram MS, et al. Cell-free methemoglobin drives platelets to apoptosis via mitochondrial ROS-mediated activation of JNK and p38 MAP kinase[J]. Biochem Biophys Res Commun, 2017, 491(1): 183-191. DOI:10.1016/j.bbrc.2017.07.073 (  0) 0) |
| [17] |
Shi Y, Hu Y, Wang J, et al. Genetic diversities of MT-ND1 and MT-ND2 genes are associated with high-altitude adaptation in yak[J]. Mitochondrial DNA A DNA Mapp Seq Anal, 2018, 29(3): 485-494. (  0) 0) |
| [18] |
Niedzwiecka K, Tisi R, Penna S, et al. Two mutations in mitochondrial ATP6 gene of ATP synthase, related to human cancer, affect ROS, calcium homeostasis and mitochondrial permeability transition in yeast[J]. Biochim Biophys Acta Mol Cell Res, 2018, 1865(1): 117-131. DOI:10.1016/j.bbamcr.2017.10.003 (  0) 0) |
| [19] |
Dzitoyeva S, Chen H, Manev H. Effect of aging on 5-hydroxymethylcytosine in brain mitochondria[J]. Neurobiol Aging, 2012, 33(12): 2881-2891. DOI:10.1016/j.neurobiolaging.2012.02.006 (  0) 0) |
| [20] |
Chowdhury B, Seetharam A, Wang Z, et al. A study of alterations in DNA epigenetic modifcations (5mC and 5hmC) and gene expression influenced by simulated microgravity in human lymphoblastoid cells[J]. PLoS One, 2016, 11(1): e0147514. DOI:10.1371/journal.pone.0147514 (  0) 0) |
| [21] |
Han D, Lu X, Shih AH, et al. A highly sensitive and robust method for genome-wide 5hmC profiling of rare cell populations[J]. Mol Cell, 2016, 63(4): 711-719. DOI:10.1016/j.molcel.2016.06.028 (  0) 0) |
| [22] |
Sun N, Zhang J, Zhang C, et al. DNMTs inhibitor SGI-1027 induces apoptosis in Huh7 human hepatocellular carcinoma cells[J]. Oncol Lett, 2018, 16(5): 5799-5806. (  0) 0) |
| [23] |
Ding K, Jiang J, Chen L, et al. Methylenetetrahydrofolate dehydrogenase 1 silencing expedites the apoptosis of non-small cell lung cancer cells via modulating DNA methylation[J]. Med Sci Monit, 2018, 24: 7499-7507. DOI:10.12659/MSM.910265 (  0) 0) |
| [24] |
Tong H, Zhang L, Gao J, et al. Methylation of mitochondrial DNA displacement loop region regulates mitochondrial copy number in colorectal cancer[J]. Mol Med Rep, 2017, 16(4): 5347-5353. DOI:10.3892/mmr.2017.7264 (  0) 0) |
 2019, Vol. 21
2019, Vol. 21

