随着新生儿诊疗技术的进步,极早产儿的存活率大幅提高,然而部分早产儿仍会发展为支气管肺发育不良(bronchopulmonary dysplasia, BPD),其主要病理学特征是肺泡简单化、结构异常、肺血管发育异常,可导致呼吸道感染加重及终身肺功能下降,中-重度BPD预后差[1-2]。因此,阐明正常肺发育的生理机制对于研究肺发育疾病具有重要意义。
环状RNA(circular RNA, circRNA)是一类在真核生物中广泛存在的RNA分子,通常由前1个外显子的“首部”和后1个外显子的“尾部”发生反向剪接以共价键的方式形成封闭的共价环状结构[3]。近些年,circRNA的生物学功能逐渐被揭示,包括:(1)miRNA海绵:circRNA包含miRNA靶位点,竞争性结合miRNA,起到miRNA海绵的作用,从而解除miRNA对靶基因的抑制作用,增加靶基因的表达水平;(2)与RNA结合蛋白(RNA binding protein, RBP)结合;(3)被翻译成肽[4-5]。随着高通量测序技术和生物信息学的发展,最近在植物、昆虫和哺乳动物中发现了丰富的circRNA[6-8]。本课题组前期研究中通过对Sprague-Dawley大鼠和C57BL/6J小鼠肺组织的circRNA表达谱进行差异筛选[9],验证了多条circRNA在肺发育过程中存在差异表达,其中circ4:150439343|150477468和circ15:73330849|73343359是本课题组在C57BL/6J小鼠胎肺组织中发现并验证的差异表达倍数较大的两条circRNA(表 1)。然而目前有关这两条circRNA的具体作用仍不清晰。有鉴于此,本研究选择肺发育3个关键代表时间点,即孕16.5 d(E16.5 d)、孕18.5 d(E18.5 d)及生后2 d(P2 d)进行研究,其分别代表小鼠肺发育的小管期、囊泡期和肺泡期。应用qRT-PCR检测circ4:150439343|150477468和circ15:73330849|73343359在这3个时间点的相对表达量的变化趋势,并通过GO和KEGG进一步分析它们的生物学功能和信号通路,探讨这两条circRNA在小鼠肺发育中的作用及病理生理机制,以期为新生儿肺部相关疾病的诊断和治疗提供新的干预靶点。
| 表 1 circ4:150439343|150477468和circ15:73330849|73343359相关测序结果 |
|
|
健康成年C57BL/6J小鼠36只(6~8周),由南京大学南京生物医学研究所提供,饲养于SPF级环境中。将雌鼠与雄鼠按照1 : 2比例合笼,次日上午检查时发现阴道栓记为雌鼠妊娠第0.5天。在E16.5 d、E18.5 d及P2 d,用4%水合氯醛麻醉(0.1 mL/10 g)孕鼠,快速取出胎鼠,采用颈椎脱臼法处死胎鼠及新生小鼠,快速取出肺组织。每组取1份左肺组织放入4%多聚甲醛中,其余肺组织经液氮速冻后储存在-80℃冰箱中。
1.2 苏木精-伊红染色向胎鼠及新生小鼠肺组织中加入4%多聚甲醛固定24 h。包埋在石蜡中,切片厚度为4 μm;二甲苯脱蜡3次;100%乙醇脱水3次,95%乙醇脱水3次,蒸馏水冲洗5 min;苏木素染色5 min,用流水稍冲洗5 min;1%盐酸乙醇分化30 s,自来水冲洗30 s,蒸馏水过水5 s;5%伊红染色3 min;80%乙醇-95%乙醇-无水乙醇彻底脱水;用二甲苯透明,最后中性树胶封盖,用Pannoramic MIDI扫描切片。
1.3 qRT-PCR法检测circ 4:150439343|150477468及circ15:73330849|73343359相对表达量用TRIzol提取肺组织总RNA,RNA质控合格后进行逆转录(Q232-02,南京诺唯赞生物科技股份有限公司)。按照PCR试剂盒(Low Rox Premixed,Q131-01,南京诺唯赞生物科技股份有限公司)说明书步骤测定肺组织中circ4:150439343|150477468和circ15:73330849|73343359相对表达量。qRT-PCR反应体系(10 μL):SYBR Green 5 μL,上下游引物各0.2 μL,DEPC水3.6 μL,cDNA 1 μL。qRT-PCR反应条件:95℃ 5 min;95℃ 15 s,60℃ 15 s,72℃ 1 min,40个循环;最后72℃ 10 min。以GAPDH为内参,计算circRNA的相对表达量= 2-△△CT[10]。引物由锐博生物科技有限公司设计并合成(表 2)。
| 表 2 用于qRT-PCR的引物序列 |
|
|
用miRanda(http://www.microrna.org/microrna/home.do)和TargetScan(http://www.targetscan.org)软件预测差异表达的circRNA的靶向miRNA。用Gene Ontology(GO, http://www.geneontology.org/)和Kyoto Encyclopedia of Genes and Genomes(KEGG, http://www.genome.jp/)分析亲本基因来预测circRNA功能。采用序列比对方法与circBase数据库中的环状序列进行比较,如果两者相似度超过90%,则认为两者是同1个circRNA[11]。
1.5 统计学分析使用SPSS 17.0统计软件和GraphPad Prism 6.0软件对数据进行统计学分析。计量资料以均数±标准差(x±s)表示,多组间比较采用方差分析,组间两两比较采用SNK-q检验,P < 0.05为差异有统计学意义。
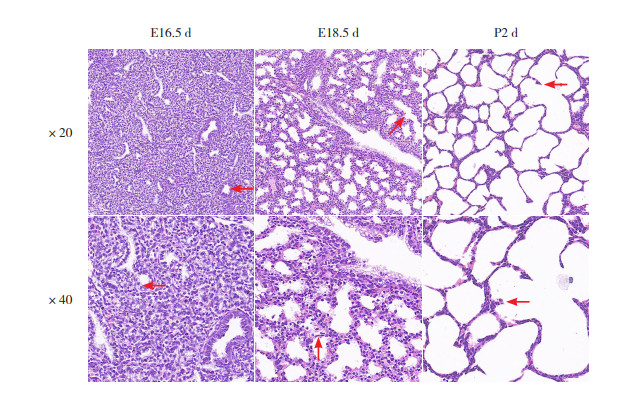
2 结果 2.1 小鼠肺发育光镜观察在E16.5 d,可见细支气管管腔被覆多层柱状上皮细胞,开始出现肺泡Ⅱ型及部分肺泡Ⅰ型上皮细胞;在E18.5 d,支气管管腔数量增加且明显扩张,间质变薄,肺泡Ⅱ型及部分肺泡Ⅰ型上皮细胞较前增多;在P2 d,肺泡上皮逐渐变薄,薄壁肺泡隔清晰可见,提示肺泡结构更加成熟。见图 1。
|
图 1 肺发育过程中小鼠肺发育的形态学变化 E16.5 d:20倍镜下可见细支气管管腔(箭头所示),40倍镜下可见管腔被覆柱状上皮细胞(箭头所示);E18.5 d:20倍镜下可见支气管管腔明显扩大,肺泡Ⅱ型上皮细胞明显增多(箭头所示),40倍镜下可见少许肺泡Ⅰ型上皮细胞(箭头所示);P2 d:可见肺泡隔及次级间隔(箭头所示)。 |
在晚期肺发育的3个关键时期,即E16.5 d、E18.5 d和P2 d这3个时间点,circ4:150439343|150477468的相对表达量连续上调(P < 0.05);circ15:73330849|73343359的相对表达量先下调再上调,P2 d的表达量明显高于其他两个时间点(P < 0.05)。见表 3。
| 表 3 qRT-PCR法检测小鼠肺发育过程中肺组织circ4: 150439343|150477468、circ15:73330849|73343359表达水平 |
|
|
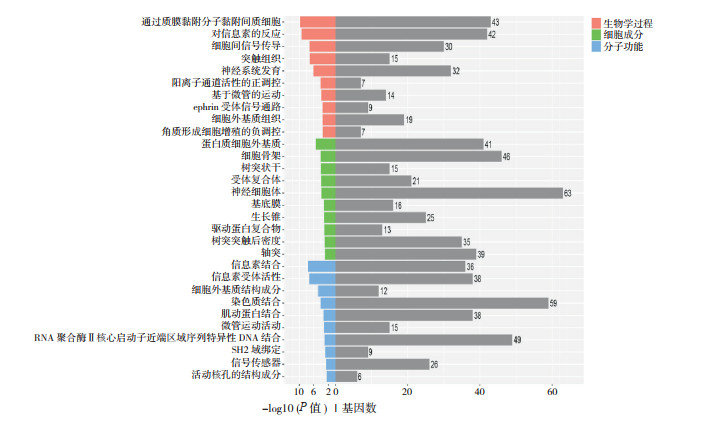
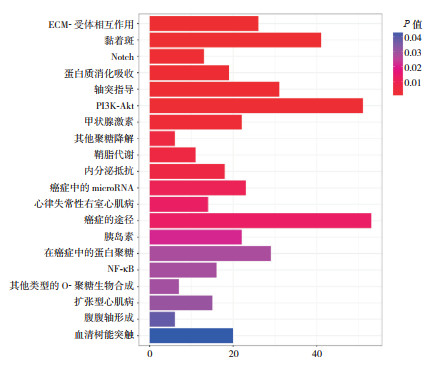
经过与circBase数据库进行比对,发现circ4: 150439343|150477468、circ15:73330849|73343359分别对应mmu_circ_0001213和mmu_circ_0005897。通过miRanda和TargetScan软件预测结果表明,circ4:150439343|150477468可能与miR-3102-5p.2-5p结合,circ15:73330849|7334335可能与miR-6769b-5p和miR-6989-3p结合。这两条circRNA从生物学过程而言,主要富集于通过质膜黏附分子黏附同质细胞、对信息素的反应、细胞间信号传导、细胞外基质组织等;从细胞成分而言,主要富集于蛋白质细胞外基质、细胞骨架、受体复合体、基底膜等;从分子功能而言,主要富集于信息素结合、信息素受体活性、细胞外基质结构成分、染色质结合等(图 2)。KEGG通路分析主要涉及Notch、PI3K-Akt、NF-κB、甲状腺激素、胰岛素、内分泌抵抗等信号通路(图 3)。
|
图 2 与circ4:150439343|150477468、circ15:73330849|73343359相互作用的miRNA靶基因的GO功能富集分析图 橘色代表生物学过程,绿色代表细胞成分,蓝色代表分子功能。各绘制出排名前10的GO功能。 |
|
图 3 与circ4:150439343|150477468、circ15:73330849|73343359相互作用的miRNA靶基因的KEEG富集分析图 横坐标代表miRNA靶基因注释在通路中的基因数,纵坐标代表通路。图中绘制出排名前20的信号通路。 |
随着产前激素和肺泡表面活性物质的应用,新生儿呼吸窘迫综合征患儿的存活率明显改善。但BPD在NICU中仍有较高的发病率和病死率,其病理生理学机制仍不明确,且缺乏有效的治疗方法[12]。因此继续探索正常肺部发育的分子机制,促进肺发育、修复和再生,或可发现治疗肺发育疾病的新靶标。circRNA是一类成环的非编码小RNA,具有miRNA海绵作用,即circRNA竞争性结合miRNA,解除miRNA对下游靶基因的抑制,增加靶基因的表达[3]。虽然已经报道了一些疾病模型的circRNA表达谱,但与小鼠肺发育相关的circRNA尚未报道。因此本课题组将着力于探索circRNA在小鼠肺发育过程中的具体作用。
本次研究发现在小鼠肺发育的3个代表性时间点(E16.5 d、E18.5 d、P2 d),circ4:150439343|150477468的相对表达量连续上调;circ15:73330849|73343359的相对表达量先下调再上调,P2 d时表达水平显著高于E16.5 d及E18.5 d时,这可能与小鼠生后肺发育已基本成熟有关。
我们预测circ4:150439343|150477468可能与miR-3102-5p.2-5p结合,circ15:73330849|73343359可能与miR-6769b-5p和miR-6989-3p结合。目前尚未有文献报道circ4:150439343|150477468和circ15:73330849|73343359的表达情况和功能。目前存有的报道表明miR-6769b-5p在白色念珠菌感染的巨噬细胞中明显下调,可能与炎症反应有关[13]。KEGG分析结果则显示,这两条circRNA主要涉及Notch、PI3K-Akt、NF-κB、甲状腺激素、胰岛素、内分泌抵抗等信号通路。而Notch、PI3K-Akt、NF-κB信号通路正与肺发育密切相关[14-15]。例如,Notch信号通路是高度保守的细胞间信号传导途径,它通过直接接触将信号传输至相邻细胞,适合短距离细胞通信。在器官发育和组织再生过程中,Notch信号通路在调节细胞增殖和分化中起重要的作用[16]。肺泡发育是由远端肺的内皮细胞、上皮细胞和间质基质协调发生的。Notch信号在血管和实质间隔的细胞分化中发挥关键作用,以协调肺泡和微血管系统的发育[17]。在肺发育过程中,肺血管中Notch基因的表达量逐渐升高,表明Notch信号通路在肺泡微脉管系统发育过程中发挥重要作用[18]。在胎肺中,Notch信号通路介导神经内分泌、纤毛细胞的分化及肺泡Ⅰ型和Ⅱ型上皮细胞的生成[19]。对Notch缺陷型小鼠进行生后存活分析表明,Notch2信号是肺泡Ⅱ型上皮细胞增殖和成熟所必需的[20]。Tsao等[21]研究表明,肺泡上皮细胞上的Notch信号通过上皮-间质相互作用影响远端细支气管和肺泡的形态发生。因此,我们推测这两条circRNA很可能通过Notch信号通路参与肺泡上皮细胞及肺血管系统的形成及成熟,与肺发育密切相关,影响肺发育的机制还需后续进一步研究。
综上,本次研究发现这两条circRNA在肺发育的3个代表性时间点差异表达,且生物信息学结果亦表明可能与肺发育密切相关。但是circRNA研究尚处于起步阶段,这两条circRNA尚未有文献报道其相关功能,在后续研究中本课题组将探讨这两条circRNA对肺发育的调节作用及其机制,为有效治疗新生儿肺部疾病提供新思路。
| [1] |
Revhaug C, Bik-Multanowski M, Zasada M, et al. Immune system regulation affected by a murine experimental model of bronchopulmonary dysplasia: genomic and epigenetic findings[J]. Neonatology, 2019, 116(3): 269-277. (  0) 0) |
| [2] |
Baker CD. Long-term ventilation for children with chronic lung disease of infancy[J]. Curr Opin Pediatr, 2019, 31(3): 357-366. (  0) 0) |
| [3] |
Kristensen LS, Andersen MS, Stagsted LVW, et al. The biogenesis, biology and characterization of circular RNAs[J]. Nat Rev Genet, 2019, 20(11): 675-691. DOI:10.1038/s41576-019-0158-7 (  0) 0) |
| [4] |
Du WW, Zhang C, Yang W, et al. Identifying and characterizing circRNA-protein interaction[J]. Theranostics, 2017, 7(17): 4183-4191. DOI:10.7150/thno.21299 (  0) 0) |
| [5] |
Rong D, Sun H, Li Z, et al. An emerging function of circRNA-miRNAs-mRNA axis in human diseases[J]. Oncotarget, 2017, 8(42): 73271-73281. DOI:10.18632/oncotarget.19154 (  0) 0) |
| [6] |
Sun P, Li G. CircCode: a powerful tool for identifying circRNA coding ability[J]. Front Genet, 2019, 10: 981. DOI:10.3389/fgene.2019.00981 (  0) 0) |
| [7] |
Zhang G, Diao S, Zhang T, et al. Identification and characterization of circular RNAs during the sea buckthorn fruit development[J]. RNA Biol, 2019, 16(3): 354-361. DOI:10.1080/15476286.2019.1574162 (  0) 0) |
| [8] |
Hu X, Zhu M, Liu B, et al. Circular RNA alterations in the Bombyx mori midgut following B. mori nucleopolyhedrovirus infection[J]. Mol Immunol, 2018, 101: 461-470. DOI:10.1016/j.molimm.2018.08.008 (  0) 0) |
| [9] |
Shen YQ, Pan JJ, Sun ZY, et al. Differential expression of circRNAs during rat lung development[J]. Int J Mol Med, 2019, 44(4): 1399-1413. (  0) 0) |
| [10] |
Livak KJ, Schmittgen TD. Analysis of relative gene expression data using real-time quantitative PCR and the 2(-delta delta C(T)) method[J]. Methods, 2001, 25(4): 402-408. (  0) 0) |
| [11] |
Langmead B, Trapnell C, Pop M, et al. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome[J]. Genome Biol, 2009, 10(3): R25. (  0) 0) |
| [12] |
Morty RE. Recent advances in the pathogenesis of BPD[J]. Semin Perinatol, 2018, 42(7): 404-412. DOI:10.1053/j.semperi.2018.09.001 (  0) 0) |
| [13] |
Wu CX, Cheng J, Wang YY, et al. Microrna expression profiling of macrophage line raw264.7 infected by candida albicans[J]. Shock, 2017, 47(4): 520-530. DOI:10.1097/SHK.0000000000000766 (  0) 0) |
| [14] |
Kiyokawa H, Morimoto M. Notch signaling in the mammalian respiratory system, specifically the trachea and lungs, in development, homeostasis, regeneration, and disease[J]. Dev Growth Differ, 2020, 62(1): 67-79. (  0) 0) |
| [15] |
Espín-Palazón R, Traver D. The NF-κB family: key players during embryonic development and HSC emergence[J]. Exp Hematol, 2016, 44(7): 519-527. DOI:10.1016/j.exphem.2016.03.010 (  0) 0) |
| [16] |
Liu J, Sato C, Cerletti M, et al. Notch signaling in the regulation of stem cell self-renewal and differentiation[J]. Curr Top Dev Biol, 2010, 92(10): 367-409. (  0) 0) |
| [17] |
Hussain M, Xu C, Ahmad M, et al. Notch signaling: linking embryonic lung development and asthmatic airway remodeling[J]. Mol Pharmacol, 2017, 92(6): 676-693. DOI:10.1124/mol.117.110254 (  0) 0) |
| [18] |
Taichman DB, Loomes KM, Schachtner SK, et al. Notch1 and jagged1 expression by the developing pulmonary vasculature[J]. Dev Dyn, 2002, 225(2): 166-175. DOI:10.1002/dvdy.10146 (  0) 0) |
| [19] |
Liu Y, Sadikot RT, Adami GR, et al. FoxM1 mediates the progenitor function of typeⅡepithelial cells in repairing alveolar injury induced by Pseudomonas aeruginosa[J]. J Exp Med, 2011, 208(7): 1473-1484. DOI:10.1084/jem.20102041 (  0) 0) |
| [20] |
Xu K, Nieuwenhuis E, Cohen BL, et al. Lunatic fringe-mediated notch signaling is required for lung alveogenesis[J]. Am J Physiol Lung Cell Mol Physiol, 2010, 298(1): L45-L56. DOI:10.1152/ajplung.90550.2008 (  0) 0) |
| [21] |
Tsao PN, Matsuoka C, Wei SC, et al. Epithelial notch signaling regulates lung alveolar morphogenesis and airway epithelial integrity[J]. Proc Natl Acad Sci U S A, 2016, 113(29): 8242-8247. DOI:10.1073/pnas.1511236113 (  0) 0) |
 2020, Vol. 22
2020, Vol. 22


