患儿男,1日龄,因哭声弱1 d,反复唇周青紫2 h入院。患儿系第2胎第2产,39+4周顺产出生,1 min、5 min Apgar评分均为10分,出生体重3 100 g,出生史无特殊。母孕期定期产检,孕23周时外院胎儿B超示后颅窝池略宽,约9 mm。后于我院行MRI检查示胎儿后颅窝池略宽,前后径最宽处约10.1 mm,行无创DNA检查无异常。否认家族遗传病史。其兄1岁10个月,体健。
入院体格检查:T 36.4℃,P 145次/min,R 42次/min,体重2.83 kg,血压66/40 mm Hg,身长49 cm,头围33 cm,胸围32 cm;神志清楚,反应正常,营养中等,肤色红润,哭声弱,哭时伴口唇发绀,呼气相口唇呈“噗噗”吹气样表现。枕部突出,眼距宽,眼裂小,双眼向外下斜,高腭弓,双手通贯掌,右足扁平、外翻。余无特殊。入院胸片示新生儿肺炎、心脏增大。血常规检查示WBC 15.76×109/L(参考值:5.0~21.0×109/L),血红蛋白172 g/L(参考值:130~200 g/L),血小板198×109/L(参考值:125~350×109/L),L 11.4%(参考值:20.0%~40.0%),N 81.3%(参考值:50.0%~70.0%);C反应蛋白(CRP)38.2 mg/L(参考值:0~5.0 mg/L)。肝肾功能、电解质正常。心脏超声示先天性心脏病:动脉导管未闭(2.3 mm);房间隔缺损Ⅱ孔型(2.5 mm)。肝胆胰脾、双肾B超无异常。头颅MRI检查示胼胝体压部略显短;大枕大池;少许蛛网膜下腔出血。染色体核型:46 XY。
入院后予低流量鼻导管吸氧,以及头孢噻肟钠抗感染治疗。7 d后,复查感染指标均正常。入院后予泵奶喂养逐渐过渡至人工喂养。患儿吸吮力弱,喂养时伴有口唇发绀,予吸氧可缓解。入院7 d后患儿家长自行要求出院。
患儿出生有特殊面容,喂养困难,其临床表现不能用感染、缺氧等原因进行解释,为了尽快明确诊断,在征得患儿家长同意后,于出院前采集患儿及父母外周血各2 mL,送至第三方医学检验所检测(北京康旭医学检验所有限公司),进行染色体拷贝数变异与全外显子测序分析。
提取患儿及家系外周血中的基因组,通过低深度高通量测序对患儿进行染色体拷贝数分析,未见异常。使用覆盖39 Mb人类基因组的IDT探针(IDT xGen Exome Research Panel包含约430 000个单独合成的探针)对患儿外周血的基因组进行靶向区域捕获测序。通过数据处理,获得靶向区域突变位点信息,使用PolyPhen、SIFT、Mutation Taster软件对原始数据进行蛋白质功能损伤分析。根据患儿临床症状、基因遗传方式、变异频率、测序深度等信息最终选出需要进一步验证的突变位点。对突变位点的致病性按照美国医学遗传学与基因组学学会(ACMG)遗传变异分类标准与指南进行分析和注释。对于疑似突变位点,对患儿及家系采用Sanger测序进行验证。
高通量全外显子组测序分析显示患儿CTCF基因杂合缺失4 bp,即c.778_781delAAAG。对该位点使用SIFT Indel算法,缺失的效果是“有害的”,因为它会导致第260位后氨基酸发生移码,同时在移码后的第2位引入终止密码,使编码的CTCF蛋白为截断性蛋白(p.Lys260ValfsTer2),这意味着生成的CTCF只有它的N-末端区域,而缺少其11个锌指结构域和C末端区域。对父母进行CTCF测序未发现携带这种突变。见图 1。
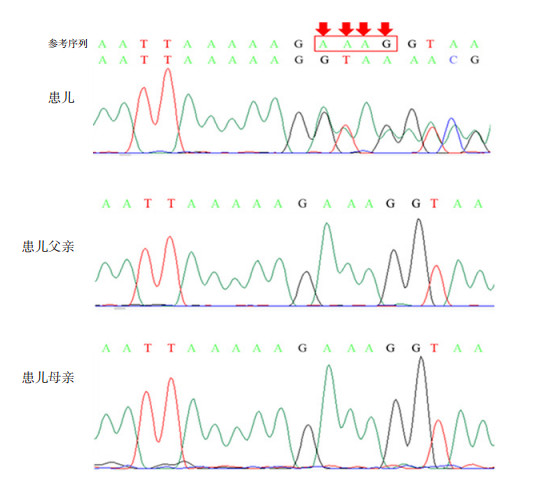
|
图 1 患儿及患儿父母CTCF Sanger测序峰图 箭头所示为CTCF c.778_781位点AAAG杂合缺失位置。 |
经Sanger测序验证,患儿父母均未携带该变异,该变异为新发突变。结合患儿临床表现及遗传学分析结果,患儿被诊断为常染色体显性智力障碍21型。患儿生后6月龄因喂养困难及反复感染夭折。
讨论:Gregor等[1] 2013年首次报道了CTCF(CCCTC结合因子;MIM 604167)突变与患者不同严重程度的智力残疾、小头畸形和生长发育迟缓有关。以“CTCF gene mutation”和“CTCF基因”为关键词,从PubMed、中国知网、万方等数据库进行检索,检索时间自2013年1月至2020年4月,发现由CTCF基因突变致病或可能致病的患者的临床详细信息涉及49例(包含染色体拷贝数变异),其中39例为新生突变。49例患儿中,3例为中国患儿,分别为1例1岁7个月女童携带c.615_618delGAAA(p.Lys206Profs*15),1例7岁11个月男童携带c.329dupT(p.Gly111fs*29),1例8岁7个月女童携带c.1699C > T(p.Arg567Trp)[1-5]。少数病例未被检索到详细的临床特征,不包括在这49例患儿中。如Meng等[6]应用全外显子组测序对出生100 d左右的危重症新生儿进行辅助诊断,其中1例29 d男性患儿为CTCF基因c.1103G > T(p.R368L)杂合新生突变,其父母未见变异;Willsey等[7]对不同地区511个复杂神经发育障碍的抽动秽语综合征患儿进行全外显子组测序数据分析,发现1例男性患儿为CTCF基因c.1244G > A(p.R415Q)突变,其父母未见变异;Retterer等[8]分析了3 040例包含各系统发育异常患儿的全外显子组测序数据,发现2个神经系统异常的患儿分别存在CTCF基因c.1288_1298del11(p.H430Cfs*10)和c.1028A > G(p.Y343C)的变异位点,未明确是否为新发突变。本例患儿存在CTCF基因c.778_781delAAAG(p.Lys260ValfsTer2)杂合缺失,而未发现其父母携带这种突变,为新发突变。
在上述具有临床详细信息的49例患儿中,CTCF基因突变数据共计33个,错义突变20个,无义突变2个,插入或缺失9个,剪切突变2个,还有5个在16q21处包含CTCF基因缺失的不同染色体变异,如1.5 Mb deletion、0.58 Mb deletion、1.1 Mb deletion、1.2 Mb deletion、0.28 Mb deletion。有约37%的患儿出现宫内发育迟缓,78%的患儿出现喂养困难,27%的患儿出现身材矮小,52%的患儿出现小头畸形,79%的患儿出现智力障碍,77%的患儿出现行为异常,10%的患儿出现癫痫,26%的患儿出现听力障碍,61%的患儿出现视力障碍,45%的患儿出现反复感染[1-5]。
CTCF基因位于16q22.1,约76.8 kbp,由13个外显子组成,编码727个氨基酸,是具有11个高度保守的锌指结构域与约46个功能性的核酸结合位点的转录调控因子。近年来的研究发现,CTCF是脊椎动物体内的关键调控蛋白之一,参与细胞中的多种生物学过程,如基因转录的抑制与激活[9-11]。CTCF参与保护常染色质区,防止异染色质区域的扩展,进而阻断转录增强子的调控起到绝缘子功能[12]。参与印记基因表达调控[13-14],也可影响DNA复制,如CTCF结合于母源等位基因ICR处,使其DNA复制时间较父源等位基因延后[15]。CTCF参与X染色体失活,可影响mRNA可变剪接[16-17]与染色质重塑[18-19]。
本例患儿与已报道CTCF基因突变患儿的部分临床表型有不同程度的吻合[2-3]。如携带CTCF基因c.688delG、c.773_776del、c.804_805delTG、c.782-2A > G、c.782-1G > T和c.329dupT的患儿均有轻度上睑下垂、眼距宽、眼裂小、薄嘴唇、小下颌、高腭弓等面部畸形;携带CTCF基因c.329dupT和c.782-1G > T的患儿出生时有喂养困难;携带CTCF基因c.782-1G > T、c.329dupT和c.804_805delTG的患儿合并动脉导管未闭和/或房间隔缺损心脏畸形;携带CTCF基因c.688delG、c.773_776del、c.804_805delTG和c.782-2A > G的患儿合并有通贯掌或足部畸形;携带CTCF基因c.773_776del、c.782-2A > G、c.782-1G > T和c.329dupT的患儿有宫内发育迟缓。本例患儿虽未出现宫内发育迟缓,但出生后有喂养困难、面容异常、先天性心脏病、手足异常、胼胝体发育不良等临床表型。因此,结合患儿临床表现和基因检测结果,患儿被确诊为常染色体显性智力障碍21型。
综上所述,CTCF基因突变可导致认知和成长的可变损害,高通量测序技术有助于提高对新生儿罕见疾病的识别与诊断。本病例提示,对于临床上有不明原因喂养困难,不能用感染、缺氧等原因进行解释的新生儿,应尽早进行遗传学分析,以帮助早期诊断和遗传咨询。
| [1] |
Gregor A, Oti M, Kouwenhoven EN, et al. De novo mutations in the genome organizer CTCF cause intellectual disability[J]. Am J Hum Genet, 2013, 93(1): 124-131. DOI:10.1016/j.ajhg.2013.05.007 (  0) 0) |
| [2] |
Konrad EDH, Nardini N, Caliebe A, et al. CTCF variants in 39 individuals with a variable neurodevelopmental disorder broaden the mutational and clinical spectrum[J]. Genet Med, 2019, 21(12): 2723-2733. DOI:10.1038/s41436-019-0585-z (  0) 0) |
| [3] |
Chen F, Yuan HM, Wu WY, et al. Three additional de novo CTCF mutations in Chinese patients help to define an emerging neurodevelopmental disorder[J]. Am J Med Genet C Semin Med Genet, 2019, 181(2): 218-225. (  0) 0) |
| [4] |
Bastaki F, Nair P, Mohamed M, et al. Identification of a novel CTCF mutation responsible for syndromic intellectual disability-a case report[J]. BMC Med Genet, 2017, 18(1): 68. DOI:10.1186/s12881-017-0429-0 (  0) 0) |
| [5] |
Hori I, Kawamura R, Nakabayashi K, et al. CTCF deletion syndrome: clinical features and epigenetic delineation[J]. J Med Genet, 2017, 54(12): 836-842. DOI:10.1136/jmedgenet-2017-104854 (  0) 0) |
| [6] |
Meng LY, Pammi M, Saronwala A, et al. Use of exome sequencing for infants in intensive care units: ascertainment of severe single-gene disorders and effect on medical management[J]. JAMA Pediatr, 2017, 171(12): e173438. DOI:10.1001/jamapediatrics.2017.3438 (  0) 0) |
| [7] |
Willsey AJ, Fernandez TV, Yu DM, et al. De novo coding variants are strongly associated with tourette disorder[J]. Neuron, 2017, 94(3): 486-499.e9. DOI:10.1016/j.neuron.2017.04.024 (  0) 0) |
| [8] |
Retterer K, Juusola J, Cho MT, et al. Clinical application of whole-exome sequencing across clinical indications[J]. Genet Med, 2016, 18(7): 696-704. DOI:10.1038/gim.2015.148 (  0) 0) |
| [9] |
Kim S, Yu NK, Kaang BK. CTCF as a multifunctional protein in genome regulation and gene expression[J]. Exp Mol Med, 2015, 47(6): e166. DOI:10.1038/emm.2015.33 (  0) 0) |
| [10] |
Klenova EM, Nicolas RH, Paterson HF, et al. CTCF, a conserved nuclear factor required for optimal transcriptional activity of the chicken c-myc gene, is an 11-Zn-finger protein differentially expressed in multiple forms[J]. Mol Cell Biol, 1993, 13(12): 7612-7624. DOI:10.1128/MCB.13.12.7612 (  0) 0) |
| [11] |
Vostrov AA, Quitschke WW. The zinc finger protein CTCF binds to the APBβ domain of the amyloid β-protein precursor promoter. Evidence for a role in transcriptional activation[J]. J Biol Chem, 1997, 272(52): 33353-33359. DOI:10.1074/jbc.272.52.33353 (  0) 0) |
| [12] |
Bell AC, West AG, Felsenfeld G. The protein CTCF is required for the enhancer blocking activity of vertebrate insulators[J]. Cell, 1999, 98(3): 387-396. DOI:10.1016/S0092-8674(00)81967-4 (  0) 0) |
| [13] |
Bell AC, Felsenfeld G. Methylation of a CTCF-dependent boundary controls imprinted expression of the Igf2 gene[J]. Nature, 2000, 405(6785): 482-485. DOI:10.1038/35013100 (  0) 0) |
| [14] |
Matsuzaki H, Kuramochi D, Okamura E, et al. Recapitulation of gametic DNA methylation and its post-fertilization maintenance with reassembled DNA elements at the mouse Igf2/H19 locus[J]. Epigenetics Chromatin, 2020, 13(1): 2. DOI:10.1186/s13072-019-0326-1 (  0) 0) |
| [15] |
Bergström R, Whitehead J, Kurukuti S, et al. CTCF regulates asynchronous replication of the imprinted H19/Igf2 domain[J]. Cell Cycle, 2007, 6(4): 450-454. DOI:10.4161/cc.6.4.3854 (  0) 0) |
| [16] |
Shukla S, Kavak E, Gregory M, et al. CTCF-promoted RNA polymerase Ⅱ pausing links DNA methylation to splicing[J]. Nature, 2011, 479(7371): 74-79. DOI:10.1038/nature10442 (  0) 0) |
| [17] |
López Soto EJ, Lipscombe D. Cell-specific exon methylation and CTCF binding in neurons regulate calcium ion channel splicing and function[J]. Elife, 2020, 9: e54879. DOI:10.7554/eLife.54879 (  0) 0) |
| [18] |
Kang H, Cho H, Sung GH, et al. CTCF regulates Kaposi's sarcoma-associated herpesvirus latency transcription by nucleosome displacement and RNA polymerase programming[J]. J Virol, 2013, 87(3): 1789-1799. DOI:10.1128/JVI.02283-12 (  0) 0) |
| [19] |
Saldaña-Meyer R, Rodriguez-Hernaez J, Escobar T, et al. RNA interactions are essential for CTCF-mediated genome organization[J]. Mol Cell, 2019, 76(3): 412-422.e5. DOI:10.1016/j.molcel.2019.08.015 (  0) 0) |
 2021, Vol. 23
2021, Vol. 23

