2. 青岛大学生物化学与分子生物学实验室, 山东 青岛 266000
神经母细胞瘤(neuroblastoma, NB)是儿童最常见的颅外实体肿瘤[1],大量研究揭示了人类神经母细胞瘤的分子特征,包括基因组、表观基因组和转录组水平的异常[2]。约25%的NB患儿存在MYCN基因扩增,而MYCN基因的扩增和高危型风险分组、预后差有关[3]。自MYCN扩增被发现以来,越来越多的MYCN扩增型NB分子机制得到了阐释。MYCN编码的mRNA和其表达的蛋白质,参与多个基因的调控,如MYCN编码的转录因子N-myc调节下游p53、S期相关蛋白激酶2(S-phase associated kinase 2, SKP2)、双微体2(murine double minute 2, MDM2)等基因,还调节一些微小RNA(miRNA)和长链非编码RNA(lncRNA)的表达[4]。而许多基因的失调,也可导致MYCN的扩增。虽然MYCN扩增型NB的分子机制一直是研究热点,但机制非常复杂,至今仍不明确。
MYCN扩增是NB预后不良的标志[5],但MYCN扩增型NB患者的5年生存率也可达50%[6]。所以需要确定新的生物标志物,以便更好地进行风险分层,并进一步深入了解NB的生物学基础。本文将MYCN扩增型NB和MYCN非扩增型NB表达的mRNA相比较,初步探究这些差异表达基因(differentially expressed genes, DEGs)的功能,并进一步筛选影响MYCN扩增型NB预后的基因,评估这些基因对其预后的预测价值。
1 资料与方法 1.1 数据来源NB患儿临床资料和基因数据均来自TARGET数据库(https://ocg.cancer.gov/programs/target)。共获取154例NB患儿的数据,其中女63例,男91例;诊断年龄3 d至16.5岁,中位诊断年龄为2.9岁。根据国际NB分期系统(International Neuroblastoma Staging System, INSS)分期,Ⅱb期1例,Ⅲ期6例,Ⅳ期126例,Ⅳ-s期21例;根据儿童肿瘤组(Children's Oncology Group, COG)危险分层,高危型127例,中危型13例,低危型14例。RNA转录组数据来自TARGET数据库中RNA测序数据,共包括18 911个mRNA。根据NB患儿有无MYCN扩增分为MYCN扩增组(n=33)和MYCN非扩增组(n=121)。
1.2 差异表达分析采用edgeR 3.9(http://www.bioconductor.org/pa-ckages/release/bioc/html/edgeR.html)识别MYCN扩增组与MYCN非扩增组间DEGs[7]。使用multtest 2.36.0(http://www.bioconductor.org/packages/release/bioc/html/multtest.html)将P值调整为假发现率(false discovery rate, FDR)[8];以FDR < 0.05和差异倍数(fold change, FC)的对数绝对值(|logFC|) > 1为差异有统计学意义。
1.3 功能富集分析和信号转导通路分析应用DAVID Bioinformatics Resources 6.8数据库(http://david.ncifcrf.gov)进行Gene Ontology(GO)功能分析和KEGG信号转导通路富集分析,并在Cytoscape中使用BiNGO插件预测潜在的功能[9]。FDR < 0.05为差异有统计学意义。
1.4 统计学分析应用SPSS 22.0和R 3.6.0软件进行数据处理。计数资料以例数表示,组间比较采用χ2检验。非正态分布计量资料以中位数和四分位数间距[M(P25,P75)]表示,组间比较采用Mann-Whitney U秩和检验。采用单因素和多因素Cox比例风险回归模型(简称Cox回归模型)分析影响MYCN扩增型NB患儿预后的基因。根据多因素Cox回归模型分析的结果,计算风险评分。风险评分=基因表达量×多因素Cox回归系数,是预测预后的指标。根据Akaike信息准则(Akaike information criterion, AIC),AIC值越小,模型越好。多因素Cox回归模型分析得到的基因,风险评分最低[10]。按风险评分的中位值将患儿分为高风险组和低风险组,采用R软件的survminer包绘制生存分析曲线。ROC曲线分析基于DEGs的风险评分对MYCN扩增型NB预后的预测价值。P≤0.05为差异有统计学意义。
2 结果 2.1 MYCN扩增组和MYCN非扩增组一般情况比较两组间患儿诊断年龄、性别、INSS分期差异无统计学意义(P > 0.05)。MYCN扩增组COG危险分层中高危型患儿比例高于MYCN非扩增组(P < 0.05)。见表 1。
| 表 1 MYCN扩增组和MYCN非扩增组一般资料比较 |
|
|
与MYCN非扩增组比较,MYCN扩增组共发现582个DEGs,其中206个DEGs表达上调,376个DEGs表达下调。部分DEGs见表 2。
| 表 2 MYCN扩增组与MYCN非扩增组中部分DEGs |
|
|
GO功能富集分析结果表明,上调的DEGs主要富集在翻译起始(44个)、蛋白质转移(40个)、核糖体(40个)、细胞外基质构成(19个)等(图 1A);表达下调的DEGs,主要富集在质膜蛋白复合物(34个)、突触组织(32个)、神经递质水平(28个)、受体调节活性(25个)和抗原处理和呈递(18个)等(图 1B)。
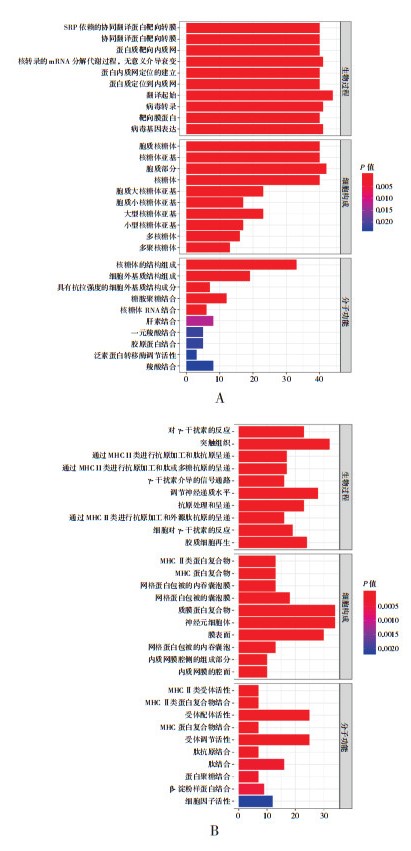
|
图 1 DEGs的GO分析 图A为上调DEGs的GO富集分析结果,图B为下调DEGs的GO富集分析结果。 |
KEGG信号转导通路分析,上调的DEGs主要涉及核糖体(41个)、蛋白质的消化和回收(8个)等(图 2A),而细胞黏附分子(27个)、类风湿关节炎(19个)等是表达下调基因参与的主要通路(图 2B)。
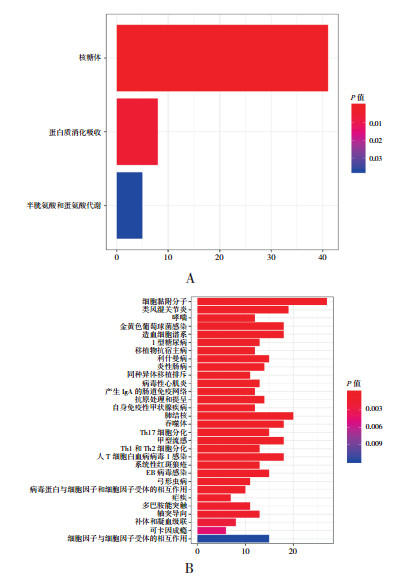
|
图 2 DEGs的KEGG分析 图A为上调DEGs的KEGG分析结果,图B为下调DEGs的KEGG分析结果。 |
以|logFC| > 2为条件,进一步筛选得到89个差异表达的mRNA。经单因素Cox回归模型分析,发现15个基因的mRNA对MYCN扩增型NB预后产生显著性影响(P≤0.05),分别为FLVCR2、FAM49A、PKIB、SCN7A、C17orf107、RGS9、INSRR、PRSS12、NTRK1、CRABP1、MBP、RP11-566K11.2、ERBB3、XAGE1A和XAGE1B,见表 3。
| 表 3 影响MYCN扩增型NB预后的单因素Cox回归模型分析 |
|
|
对单因素分析结果中差异具有统计学意义的15个基因进行多因素Cox回归模型分析,发现5个基因的mRNA可用于预测MYCN扩增型NB的预后,分别为FLVCR2、SCN7A、PRSS12、NTRK1、XAGE1A,见表 4。
| 表 4 影响MYCN扩增型NB预后的单因素Cox回归模型分析 |
|
|
基于AIC,多因素Cox回归模型分析得到的FLVCR2、SCN7A、PRSS12、NTRK1和XAGE1A的风险评分最低,为预测预后的最佳生物标志物。根据风险评分的中位值将患儿分为高风险组和低风险组。两组间患儿诊断年龄、性别差异无统计学意义(P > 0.05);高风险组INSS分期中Ⅳ期患儿比例及COG危险分层中高危型患儿比例均高于低风险组(P < 0.05)。见表 5。生存分析发现,高风险组的总生存率明显低于低风险组(P < 0.05),见图 3。
| 表 5 高风险组和低风险组患儿一般资料的比较 |
|
|
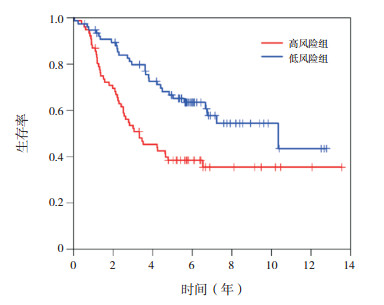
|
图 3 高风险组和低风险组生存曲线 |
基于5个mRNA的风险评分的ROC曲线下面积为0.729(95%可信区间:0.650~0.808,P < 0.001),当约登指数为0.376时,风险评分的最佳截断值为1.316,灵敏度为53.2%,特异度为84.4%, 阴性预测值为84.4%,阳性预测值为53.2%,见图 4。

|
图 4 基于5个mRNA风险评分预测MYCN扩增型NB预后的ROC曲线 |
本研究发现MYCN扩增型NB和MYCN非扩增型NB在基因表达上有许多不同,因此存在很多差异明显的mRNA。这些mRNA与MYCN扩增关系密切。虽然MYCN扩增与预后不良相关,但研究发现MYCN无法作为靶向治疗的直接靶标,需要找寻其他有效靶标[11]。而本研究为未来进一步研究NB的分子靶标提供参考。经过DEGs功能分析,初步探讨了促进MYCN扩增型NB发生的分子机制。本研究发现,FLVCR2、SCN7A、PRSS12、NTRK1、XAGE1A基因对MYCN扩增组NB患儿预后具有显著性影响,基于上述5个mRNA的风险评分对MYCN扩增组NB患儿预后有预测价值,故这5个mRNA可以作为MYCN扩增型NB的预测预后的生物标志物。
在GO分析中,上调的DEGs生物功能主要富集在蛋白质的翻译以及转运至膜的过程,细胞构成主要富集在核糖体,分子功能主要富集在核糖体和细胞外基质的结构组成,KEGG结果也显示上调的DEGs主要参与了核糖体通路的表达。因此,可以预测,MYCN扩增型NB的核糖体功能表达更加活跃,蛋白质的的翻译和转移也更加高效。而已有研究表明,MYCN扩增的NB细胞真核转录延长[12]。下调的DEGs生物功能主要富集在突触组织,神经递质的转移,分子功能主要富集在膜受体的活动上,由此可知,MYCN扩增型NB神经细胞表达更加活跃,侵袭能力更加强大。而已有研究表明,MYCN扩增促进NB细胞代谢和侵袭[13]。
研究表明,FLVCR2、SCN7A、PRSS12、NTRK1和XAGE1A参与多种生物过程和分子机制。FLVCR2主要促进主要易化子超家族(MFS)的跨膜转运蛋白,参与生长、钙交换和体内平衡的调节[14];SCN7A/Nax通道可作为体液的钠水平传感器,通过改变神经元兴奋性来控制钠摄入,增强的SCN7A / Nax表达通过增加背根神经节中神经元的兴奋性而导致骨癌疼痛[15];PRSS12编码的蛋白是由神经细胞分泌的,被认为对认知功能很重要,因为其功能丧失会导致严重的非综合征性精神发育迟滞[16]。高水平的NTRK1/TrkA受体在低阶段NB中表达,NTRK1/TrkA的表达影响NB细胞的免疫原性,其特征在于患者预后良好并且经常发生自发消退[17]。NTRK1还通过表观遗传学来调节NB的分化[18];有外显子水平分析将MYCN和NTRK1鉴定为替代外显子表达的主要决定因素,并且可以有力地预测原发性NB的预后[19]。XAGE1在头颈部鳞状细胞癌中的表达和预后有相关性,它和GAGE1是头颈部鳞状细胞癌预后的独立危险因素[19]。
自引入RNA测序和表达微列阵以来,使用mRNA表达特征作为个体患者结局的预测因子已成为目前研究的热点。本研究讨论了MYCN扩增型NB和MYCN非扩增型NB表达基因的差别并且筛选出可以预测MYCN扩增型NB预后的5个mRNA,分析了这5个基因作为预后生物标志物的预测价值。目前NB的临床治疗仍然存在很大的挑战,特别是高危患儿,即使运用多种治疗模式,也很难进一步提高生存率[20]。近年来,分子靶向治疗一直是研究热点,原因在于其可以精准地在某个关键环节上发挥作用。本研究通过层层筛选出的基因,也为NB的分子靶向治疗提供了新的素材和思路。
TARGET是大型和全面的癌症基因组数据库,基于这5个mRNA的风险评分是一个稳妥的生存预测模型。但即使通过一系列统计学算法,确定了这5个mRNA可以作为预测预后的生物标志物,临床试验等实验研究仍然是必要的,以避免假阳性的可能。并且需要一系列的实验来揭示这些mRNA在NB中的作用及机制。
| [1] |
Matthay K K, Maris J M, Schleiermacher G, et al. Neuroblastoma[J]. Nat Rev Dis Primers, 2016, 2: 16078. DOI:10.1038/nrdp.2016.78 (  0) 0) |
| [2] |
Tsubota S, Kadomatsu K. Origin and initiation mechanisms of neuroblastoma[J]. Cell Tissue Res, 2018, 372(2): 211-221. DOI:10.1007/s00441-018-2796-z (  0) 0) |
| [3] |
Huang M, Weiss WA. Neuroblastoma and MYCN[J]. Cold Spring Harb Perspect Med, 2013, 3(10): a014415. (  0) 0) |
| [4] |
赖祥萍, 赖天霞, 廖伟. 神经母细胞瘤中N-myc的作用机制研究进展[J]. 医学综述, 2017, 23(5): 926-930. DOI:10.3969/j.issn.1006-2084.2017.05.020 (  0) 0) |
| [5] |
Campbell K, Gastier-Foster JM, Mann M, et al. Association of MYCN copy number with clinical features, tumor biology, and outcomes in neuroblastoma:a report from the Children's Oncology Group[J]. Cancer, 2017, 123(21): 4224-4235. DOI:10.1002/cncr.30873 (  0) 0) |
| [6] |
Maris JM. Recent advances in neuroblastoma[J]. N Engl J Med, 2010, 362(23): 2202-2211. DOI:10.1056/NEJMra0804577 (  0) 0) |
| [7] |
Nikolayeva O, Robinson MD. edgeR for differential RNA-seq and ChIP-seq analysis:an application to stem cell biology[J]. Methods Mol Biol, 2014, 1150: 45-79. (  0) 0) |
| [8] |
Rosenblum M, Qian T, Du Y, et al. Multiple testing procedures for adaptive enrichment designs:combining group sequential and reallocation approaches[J]. Biostatistics, 2016, 17(4): 650-662. DOI:10.1093/biostatistics/kxw014 (  0) 0) |
| [9] |
Maere S, Heymans K, Kuiper M. BiNGO:a Cytoscape plugin to assess overrepresentation of gene ontology categories in biological networks[J]. Bioinformatics, 2005, 21(16): 3448-3449. (  0) 0) |
| [10] |
Xia L, Wang Y, Meng Q, et al. Integrated bioinformatic analysis of a competing endogenous RNA network reveals a prognostic signature in endometrial cancer[J]. Front Oncol, 2019, 9: 448. DOI:10.3389/fonc.2019.00448 (  0) 0) |
| [11] |
Fletcher JI, Ziegler DS, Trahair TN, et al. Too many targets, not enough patients:rethinking neuroblastoma clinical trials[J]. Nat Rev Cancer, 2018, 18(6): 389-400. DOI:10.1038/s41568-018-0003-x (  0) 0) |
| [12] |
Delaidelli A, Negri GL, Jan A, et al. MYCN amplified neuroblastoma requires the mRNA translation regulator eEF2 kinase to adapt to nutrient deprivation[J]. Cell Death Differ, 2017, 24(9): 1564-1576. (  0) 0) |
| [13] |
Ruiz-Pérez MV, Henley AB, Arsenian-Henriksson M. The MYCN protein in health and disease[J]. Genes (Basel), 2017, 8(4): pii:E113. DOI:10.3390/genes8040113 (  0) 0) |
| [14] |
Meyer E, Ricketts C, Morgan NV, et al. Mutations in FLVCR2 are associated with proliferative vasculopathy and hydranencephaly-hydrocephaly syndrome (Fowler syndrome)[J]. Am J Hum Genet, 2010, 86(3): 471-478. DOI:10.1016/j.ajhg.2010.02.004 (  0) 0) |
| [15] |
Ke CB, He WS, Li CJ, et al. Enhanced SCN7A/Nax expression contributes to bone cancer pain by increasing excitability of neurons in dorsal root ganglion[J]. Neuroscience, 2012, 227: 80-89. DOI:10.1016/j.neuroscience.2012.09.046 (  0) 0) |
| [16] |
Mitsui S, Osako Y, Yuri K. Mental retardation-related protease, motopsin (prss12), binds to the BRICHOS domain of the integral membrane protein 2a[J]. Cell Biol Int, 2014, 38(1): 117-123. DOI:10.1002/cbin.10164 (  0) 0) |
| [17] |
Pajtler KW, Rebmann V, Lindemann M, et al. Expression of NTRK1/TrkA affects immunogenicity of neuroblastoma cells[J]. Int J Cancer, 2013, 133(4): 908-919. (  0) 0) |
| [18] |
Li Z, Takenobu H, Setyawati AN, et al. EZH2 regulates neuroblastoma cell differentiation via NTRK1 promoter epigenetic modifications[J]. Oncogene, 2018, 37(20): 2714-2727. DOI:10.1038/s41388-018-0133-3 (  0) 0) |
| [19] |
Schramm A, Schowe B, Fielitz K, et al. Exon-level expression analyses identify MYCN and NTRK1 as major determinants of alternative exon usage and robustly predict primary neuroblastoma outcome[J]. Br J Cancer, 2012, 107(8): 1409-1417. DOI:10.1038/bjc.2012.391 (  0) 0) |
| [20] |
姚伟, 李凯, 郑珊. 神经母细胞瘤靶向治疗的应用现状和展望[J]. 中华小儿外科杂志, 2018, 39(10): 792-796. DOI:10.3760/cma.j.issn.0253-3006.2018.10.017 (  0) 0) |
 2020, Vol. 22
2020, Vol. 22


